久しぶりに構造方程式モデリングをしようと思う。
ということで、Rで構造方程式モデリングをする場合のパッケージはlavaanとsemの二つがあるけど、なにせ使うのは7,8年ぶりなのでちょっと最近のそれぞれの様子を確認してみた。
なんと描画ができるではないか!
ということでチャレンジ。
(こっちも参考にした)
モデル作り
とりあえずは簡単な媒介モデル。
model<-'
sQ1_1 ~ Q2_3_2 + f1
Q2_3_3~ f1
'
SEM実行
r<-sem(model, data=na.omit(data[c("sQ1_1","Q2_3_2","f1)]) )
summary(r, standardized = TRUE,fit.measure=TRUE)
結果
lavaan 0.6-8 ended normally after 12 iterations
Estimator ML
Optimization method NLMINB
Number of model parameters 5
Number of observations 176
Model Test User Model:
Test statistic 0.000
Degrees of freedom 0
Model Test Baseline Model:
Test statistic 18.147
Degrees of freedom 3
P-value 0.000
User Model versus Baseline Model:
Comparative Fit Index (CFI) 1.000
Tucker-Lewis Index (TLI) 1.000
Loglikelihood and Information Criteria:
Loglikelihood user model (H0) -494.071
Loglikelihood unrestricted model (H1) -494.071
Akaike (AIC) 998.142
Bayesian (BIC) 1013.994
Sample-size adjusted Bayesian (BIC) 998.161
Root Mean Square Error of Approximation:
RMSEA 0.000
90 Percent confidence interval - lower 0.000
90 Percent confidence interval - upper 0.000
P-value RMSEA <= 0.05 NA
Standardized Root Mean Square Residual:
SRMR 0.000
Parameter Estimates:
Standard errors Bootstrap
Number of requested bootstrap draws 1000
Number of successful bootstrap draws 1000
Regressions:
Estimate Std.Err z-value P(>|z|) Std.lv Std.all
sQ1_1 ~
Q2_3_2 0.176 0.072 2.432 0.015 0.176 0.180
f1 -0.117 0.083 -1.414 0.157 -0.117 -0.110
Q2_3_2 ~
f1 -0.239 0.088 -2.726 0.006 -0.239 -0.218
Variances:
Estimate Std.Err z-value P(>|z|) Std.lv Std.all
.sQ1_1 0.942 0.080 11.848 0.000 0.942 0.947
.Q2_3_2 0.998 0.131 7.617 0.000 0.998 0.953
間接効果の検証
媒介モデルが成立するかどうかは、
- 説明変数から目的変数へのパスがそもそも有意である
- 説明変数から媒介変数へのパスと媒介変数から目的変数へのパスが有意である
- 媒介変数を介した間接効果が有意である
という3つが必要。
ということで、間接効果の検証をする。
要するに、説明変数から媒介変数へのパス係数と媒介変数から目的変数へのパス係数の掛け算が、有意になるかどうか(0でないかどうか)を調べるというものです。
ついでに今回はパラメータ推定にブートストラップをしてみる(semでse="bootstrap")のと、出力に信頼区間も表示させてみます(summaryでci=TRUE)。
model <-'
sQ1_1 ~ a*Q2_3_2+b*f1
Q2_3_2~ c*f1
#間接効果
bc:=b*c
#全体効果
total:= a+bc
'
r.sem<- sem(model,
data=na.omit(data.clust[c("sQ1_1","Q2_3_2","f1","f2","f3","f4","f5")]),
se="bootstrap")
summary(r.sem,standardized = TRUE,fit.measure=TRUE,ci=TRUE)
結果
lavaan 0.6-8 ended normally after 12 iterations
Estimator ML
Optimization method NLMINB
Number of model parameters 5
Number of observations 176
Model Test User Model:
Test statistic 0.000
Degrees of freedom 0
Model Test Baseline Model:
Test statistic 18.147
Degrees of freedom 3
P-value 0.000
User Model versus Baseline Model:
Comparative Fit Index (CFI) 1.000
Tucker-Lewis Index (TLI) 1.000
Loglikelihood and Information Criteria:
Loglikelihood user model (H0) -494.071
Loglikelihood unrestricted model (H1) -494.071
Akaike (AIC) 998.142
Bayesian (BIC) 1013.994
Sample-size adjusted Bayesian (BIC) 998.161
Root Mean Square Error of Approximation:
RMSEA 0.000
90 Percent confidence interval - lower 0.000
90 Percent confidence interval - upper 0.000
P-value RMSEA <= 0.05 NA
Standardized Root Mean Square Residual:
SRMR 0.000
Parameter Estimates:
Standard errors Bootstrap
Number of requested bootstrap draws 1000
Number of successful bootstrap draws 1000
Regressions:
Estimate Std.Err z-value P(>|z|) ci.lower ci.upper Std.lv Std.all
sQ1_1 ~
Q2_3_2 (a) 0.176 0.073 2.397 0.017 0.035 0.319 0.176 0.180
f1 (b) -0.117 0.085 -1.386 0.166 -0.285 0.056 -0.117 -0.110
Q2_3_2 ~
f1 (c) -0.239 0.083 -2.894 0.004 -0.409 -0.079 -0.239 -0.218
Variances:
Estimate Std.Err z-value P(>|z|) ci.lower ci.upper Std.lv Std.all
.sQ1_1 0.942 0.078 12.136 0.000 0.778 1.085 0.942 0.947
.Q2_3_2 0.998 0.136 7.340 0.000 0.721 1.251 0.998 0.953
Defined Parameters:
Estimate Std.Err z-value P(>|z|) ci.lower ci.upper Std.lv Std.all
bc 0.028 0.025 1.120 0.263 -0.010 0.087 0.028 0.024
total 0.204 0.071 2.868 0.004 0.068 0.347 0.204 0.204
ということでbcの値が0を超えてないので、残念ながら今回のモデルは媒介モデルとは言えない。
つまり、f1がQ2_3_2を経由してQ1_1に影響を与えているとは言えない、という結果でした。
適合度指標
上記のようにsummaryでfit.measure=TRUEとしておくと主要な適合度指標も一緒に表示してくれます(デフォルトはFALSEです)が、それとは別に適合度指標を表示させたい場合には、fitMeasures()を使います。
> fitMeasures(r.sem)
npar fmin chisq df pvalue
5.000 0.000 0.000 0.000 NA
baseline.chisq baseline.df baseline.pvalue cfi tli
18.147 3.000 0.000 1.000 1.000
nnfi rfi nfi pnfi ifi
1.000 1.000 1.000 0.000 1.000
rni logl unrestricted.logl aic bic
1.000 -494.071 -494.071 998.142 1013.994
ntotal bic2 rmsea rmsea.ci.lower rmsea.ci.upper
176.000 998.161 0.000 0.000 0.000
rmsea.pvalue rmr rmr_nomean srmr srmr_bentler
NA 0.000 0.000 0.000 0.000
srmr_bentler_nomean crmr crmr_nomean srmr_mplus srmr_mplus_nomean
0.000 0.000 0.000 0.000 0.000
cn_05 cn_01 gfi agfi pgfi
1.000 1.000 1.000 1.000 0.000
mfi ecvi
1.000 0.057
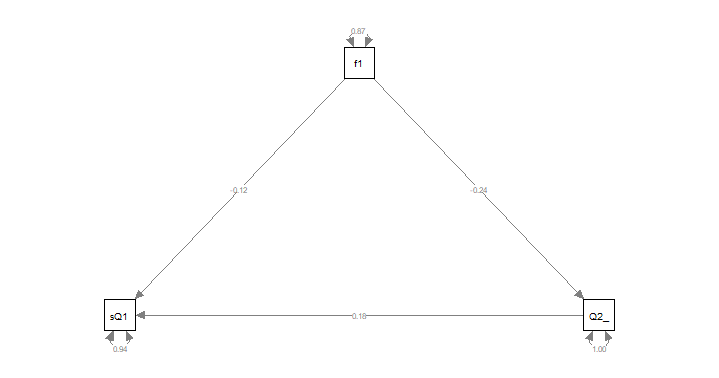
モデル描画 その1 semPaths()
モデルを描画させる方法として簡単なのは、semPlotパッケージにあるsemPaths()を使う方法です。
library(semPlot)
semPaths(r.sem, "model", "est", intercepts = FALSE)
ただ、すこし微妙です。
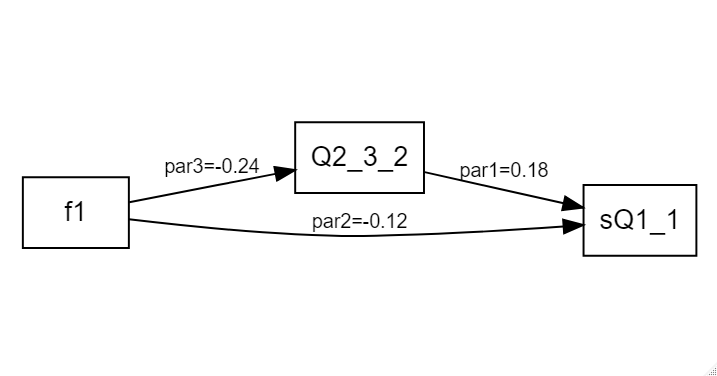
モデル描画 その2 semパッケージとDiagrammeRパッケージ
最初に挙げた参考URLにかかれてあるものを、ほぼデットコピーです。(苦笑
library(sem)
library(DiagrammeR)
# semパッケージで構造方程式モデリングを行うためのモデルの書式変換
tmp <- semPlotModel(r.sem)
pars <- tmp@Pars
pars$label <- paste0("par",1:length(pars$par))
pars$edge[pars$edge == "~>"] <- "->"
tmp.m <- paste(paste(pars$lhs, pars$edge, pars$rhs),
pars$label,
pars$est,
sep = ",",
collapse = "\n")
model.s <- sem::specifyModel(textConnection(tmp.m))
# semパッケージのsem関数を使ったモデリング
res.s <- sem::sem(model.s,
data=na.omit(data.clust[c("sQ1_1","Q2_3_2","f1")]) )
# 描画
g <- sem::pathDiagram(res.s,edge.labels="both")
grViz(g)
こっちの方が見やすくて配置もいい感じです。
その他参考