困難な人数による比較
第二波が来ていると言われている。確かに連日の報道によれば例えば東京都のPCR検査陽性数は非常事態宣言が行われていた時を超えている。だがその背景にはいわゆる夜の町クラスター対策として新宿区見舞金制度が導入されたことにより、特に水商売に従事する若年層がPCR検査を受けやすくなった事があげられている。そして、実際ローリスクと言われる50代以下の世代を集計すると既に前回のピークを上回っている事が解る。
よって単純な人数の比較からでは実際の深刻度が解らなくなってしまいかねない。
Covid-19/コロナウィルスにおいて重要なのは
0. 隔離施設のキャパシティをアクティブな感染者が超えないこと
1.ICU、人工呼吸器などを使う重症者がそれらのキャパシティを越えないこと
2. 死者が増加しないこと
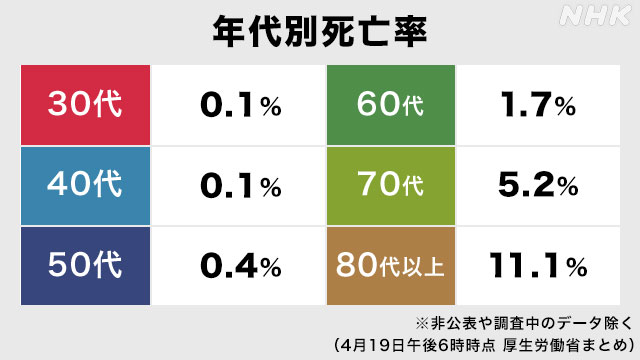
である。0 は一定期間の陽性者数と相関するが深刻さの指標となる1,2は単なる人数からはわからない。そこで、既に解っている年代別の死亡率を判明している年代別陽性者数で集計することにより今後重症者に推移する潜在推定死者数を推計し、危険度として推移を集計してみた。
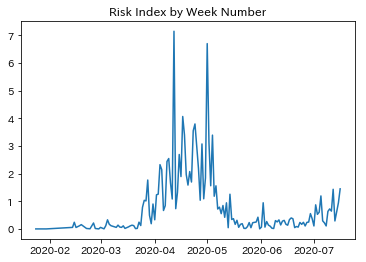
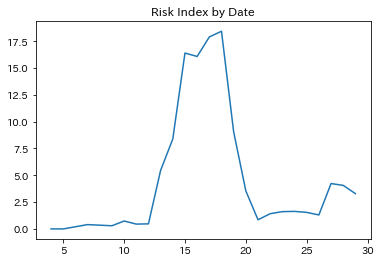
以下が日にち別の集計グラフ
一見すれば明らかなように増加傾向であるが前回のピークに比べると遥かに低い水準であることが解る。現在の陽性者が重症者に推移するには一定の時間がかかる。よって次のようなことが言える
PCR陽性者の増加傾向から隔離施設に関しては数週間以内にキャパシティが切迫する可能性が高い
しかし、潜在推定死者数から深刻度は非常事態宣言したころよりは低い
同様に重症者向け施設の不足は当面発生しない
事が解る。背景は冒頭にあげた若年層のPCR検査数増加と老人介護施設などハイリスクグループが存在する施設などでのソーシャルディスタンスの実施度合いが高まっている事等だろう。
一方、若年感染者の数自体が増えていることは事実でこれがハイリスクグループに飛び火すれば一気に深刻度が上昇しかねないともいえる。現状に関しては多角的な数値を正確に分析した上で経済との兼ね合いを図りながら規制を考えて欲しい。
以下、Jupyter などで走る分析用コード。localdirにデータソース 東京都 新型コロナウイルス陽性患者発表詳細をダウンロードすることで同じ結果が得られる。
# coding: utf-8
import pandas as pd
import numpy as np
import matplotlib.pyplot as plt
import datetime as dt
localdir = "/Downloads/"
csv_data = localdir +"130001_tokyo_covid19_patients.csv"
age_dic = {'不明':0.001,'10歳未満':0.001,"'-":0.001,'90代':0.111,'100歳以上':0.111,'70代':0.052,'80代':0.111,'60代':0.017,'50代':0.004,'40代':0.001,'30代':0.001,'20代':0.001,'10代':0.001}
high_risks = ('90代','100歳以上','70代','80代','60代')
labels = ('公表_年月日','患者_年代')
dataset = pd.read_csv(csv_data,encoding = 'utf-8',header=0,)
dataset = dataset.loc[:,labels]
print (set(dataset.loc[:,'患者_年代']))
dataset.loc[:,'date'] = pd.to_datetime(dataset.loc[:,'公表_年月日'])
dataset.loc[:,'weekday'] = dataset.date.dt.week
dataset.loc[:,'val'] = dataset.loc[:,'患者_年代']
high_risk_table = dataset[~dataset.val.isin(high_risks)]
dataset.val = dataset.val.replace(age_dic)
datahighrisk = high_risk_table.loc[:,('date','val')]
databyweek = dataset.loc[:,('weekday','val')]
databydate = dataset.loc[:,('date','val')]
# dataset = dataset.fillna(0)
print(dataset)
highriskcount = datahighrisk.groupby('date').count()
deathbyweek = databyweek.groupby('weekday').sum()
deathbyday = databydate.groupby('date').sum()
plt.title("Risk Index by Date")
plt.plot(deathbyweek.index,deathbyweek.val)
plt.show()
plt.title("Risk Index by Week Number")
plt.plot(deathbyday.index,deathbyday.val)
plt.show()
plt.title("Count of Low Risk Group")
plt.plot(highriskcount.index,highriskcount.val)
plt.show()