PubMedでは論文の要旨(Abstract)が公開されていますが、数百報、数千報の要旨を手作業で調べるのはとても面倒です。
そこで、本記事ではワードクラウドを使って大量の論文をテキストマイニングします。ワードクラウドを使えば、論文の中でよく使われている単語が分かります。
環境
本記事における分析は、全てGoogle Colaboratoryで実装しました。
論文の要旨を取得するまで
今回は1000報の論文を分析しますが、それらをひとつひとつ手作業でダウンロードしていては時間がもったいないので、プログラミングで自動ダウンロードさせます。
まずは、biopythonをインストールします。
!pip install biopython
次に、biopythonを読み込み、自分のメールアドレスを登録します。
from Bio import Entrez
Entrez.email = "自分のメールアドレス"
調べたいキーワードを指定します。これらのキーワードの検索結果が、変数pmidsにリスト形式で格納されます。
term = "covid-19 age risk"
handle = Entrez.esearch(db="pubmed", term=term, retmax=1000)
record = Entrez.read(handle)
pmids = record["IdList"]
検索結果のうち、PMIDのみをpmidsに格納しました。PMIDはPubMedに保存されている論文の識別番号です。
続いて、PMIDを入力して、論文の要旨を取得させる関数を作成します。
def get_abstract(pmid):
try:
handle = Entrez.efetch(db="pubmed", id=pmid, rettype="medline", retmode="xml")
return " ".join(Entrez.read(handle)["PubmedArticle"][0]["MedlineCitation"]["Article"]["Abstract"]["AbstractText"])
except:
return "emoriiin979"
PubMedには要旨が存在しない論文も保存されていて、それらからAbstractTextキーのデータを取得しようとするとエラーが出るので、要旨が存在しない場合は私の名前が出力されるようにしました。
こうすることで、後で私の名前を分析に使わないよう指定すれば、正常に動作させることができます。
それでは、この関数を使ってPubMedのAPIにアクセスし、要旨を取得します。
from tqdm import tqdm
from time import sleep
text = ""
for i in tqdm(range(len(pmids))):
text += " " + get_abstract(pmids[i])
sleep(0.5)
今回はWordCloudライブラリでワードクラウドを作成するため、994報(6報は要旨なしでした)の要旨を一つの文字列にまとめています。
1回PubMedにアクセスするごとに0.5秒の休止時間を設けているため、1000報の場合20分くらい取得に時間がかかります。
ワードクラウドを作成する
分析に使う文字列が得られたので、いよいよワードクラウドを作成します。
まずは、ライブラリの読み込みと分析に使用する辞書をダウンロードします。
import nltk
from nltk import tokenize
from nltk import stem
from nltk.corpus import stopwords
nltk.download("punkt")
nltk.download("wordnet")
nltk.download("stopwords")
次に、文字列を単語に分解(トークン化)します。
例えば、"I like an apple."という文字列を与えた場合、["I", "like", "an", "apple", "."]に分解されます。
words = tokenize.word_tokenize(text)
filtered_words = [w for w in words if w not in stopwords.words("english")]
ここで、分析に使用しない単語(stopwords)の除去も同時に行っています。
次に、単語のレンマ化(見出し語化)を行います。例えば、"apples"という単語を与えた場合、"apple"に変換されます。
lemmatizer = stem.WordNetLemmatizer()
lem_text1 = ""
for word in filtered_words:
lem_text1 += lemmatizer.lemmatize(word) + " "
これで、分析に使う単語リストの正規化が完了しました。
最後に、WordCloudライブラリでワードクラウドを作成します。
from wordcloud import WordCloud
wc1 = WordCloud(background_color="white", width=600, height=400, min_font_size=15)
wc1.generate(lem_text1)
wc1.to_file("wordcloud1.png")
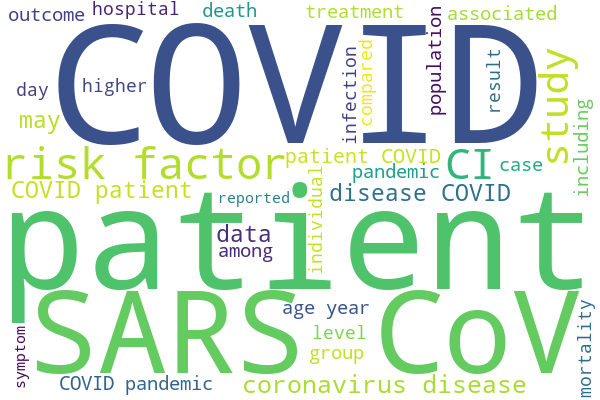
作成されたワードクラウドは、以下の通りです。
この図で文字サイズが大きくなっているのは、論文中での出現頻度が高い単語です。
今回は検索キーワードに"covid-19"を入れているので、当然ながら"COVID"や"CoV"の出現頻度が高くなっています。
必要ない単語を除外する
"COVID"や"CoV"といった単語は出力されて当然であり、これらは重要性の低い情報なので、これらの単語を分析の対象外としたいと思います。
まずは、現時点でのワードクラウドから必要なさそうな単語を選択します。
more_stopwords = [
"patient",
"study",
"infection",
"pandemic",
"result",
"coronavirus",
"among",
"outcome",
"data",
"may",
"included"
]
そして、これらの単語をfiltered_wordsから除去して、再びワードクラウドを作成しなおします。
lem_text2 = ""
for word in filtered_words:
tmp = lemmatizer.lemmatize(word)
if tmp not in more_stopwords:
if "COVID" not in tmp and "CoV" not in tmp:
lem_text2 += tmp + " "
wc2 = WordCloud(background_color="white", width=600, height=400, min_font_size=15)
wc2.generate(lem_text2)
wc2.to_file("wordcloud2.png")
"COVID"と"CoV"については、"COVID-19"などの表記ゆれが存在しており完全一致の除外ができなかったため、個々に部分一致の除外を行っています。
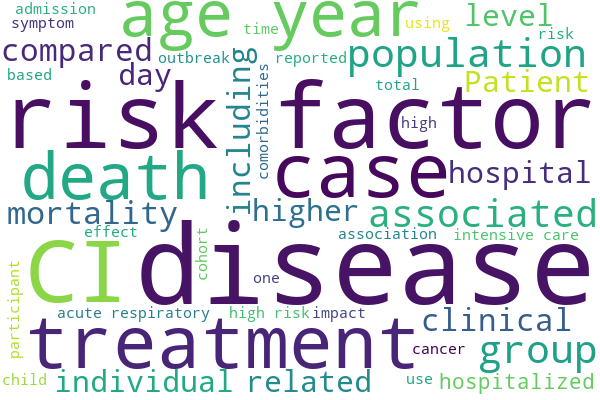
これらの単語を除外した結果は、以下の通りになりました。
先ほど選択した単語は、ある程度除外されたように見えます。
この工程を繰り返していけば、いずれ必要な単語のみのワードクラウドができるかと思われます。
最後に
この記事では、biopythonとWordCloudを使って、PubMedの論文要旨における使用単語の出現頻度について分析しました。
今回の分析の感想としては、NLTKのトークン化やレンマ化の精度が少々悪かったかなと感じました。
トークン化ではカンマ(,)やピリオド(.)などが除去できていなかったり、レンマ化では"accompanies"が"accompany"に変換できずそのまま出力されるなどがあったりしたので、これらの精度を高めるのが今後の課題のような気がします。
また、ワードクラウドだけではなく、単語の出現頻度には他にも分析方法が存在するようなので、これらについても色々調べて使ってみたいと思います。
以上です。