論文出したいから、データをオープンにしたいなぁ
筆者はせっかくシーケンスをしたバクテリアゲノムがFASTQファイルのままHDDで封印されているケースを何度も目撃してきた。しかし、ドラフトゲノム はおおよそほとんど全ての遺伝子配列を習得できるし、微生物研究を行う上では超有用である。研究ターゲットを絞る、PCRの際に参考にするetcなど無限の応用可能性を秘めている。そこで以前に最もシンプルと思われる方法でのアセンブル方法を紹介した。
もし、その方法でアセンブルをしたとして、この配列を研究で利用した場合にはオープンソースにするべきであろう。そこでDDBJを通じてデータを公開したときのメモ的なものを公開する。大量のサンプルでなければゲノムの登録作業はそこまで時間はかからない (トータル1-2週間もあれば全然できる) 。当方は共著者を含めて何度かドラフトゲノム を登録している。初めて自分で登録した時、また後輩を指導したときに苦労した点を新しくやる人に向けてをまとめていく。本稿を読む前にTogoTVを見て欲しい (以下にリンクあり)、TogoTVではトランスクリプトーム解析で得られたものの登録方法を公開している。。本稿はこれに追加し、バクテリアのドラフトゲノムの登録の要領をまとめている。しかし、筆者は既にバクテリアのフィールドを離れており、一部知識が間違っているかもしれない。間違っている点があれば、ぜひ指摘をお願いしたい。
データの準備
ゲノムデータの公開をするには、Fastqの生データとアセンブル済みのドラフトゲノム があればOKである。最も楽に提出するにはDFAST web版でアノテーションをかけるといいと思う。バクテリアドラフトゲノム を報告する論文といえばMicrobiology resource announcementが有名であるが、筆者が十数報読んでたどり着いた解析スタメンたちを以下の記事にて解説している。既に解析を終えている場合は、このまま読み進めて、解析を終えていない場合は以下の記事を参考にしてから次の項へと飛んでほしい。
まずやるべきこと (生Fastq登録)
まずやるべきことはTogoTVを見ることである。動画なので、見ながらやれば早い。その後、サイトの説明を読むとやるべきことがわかる。
TogoTV: https://togotv.dbcls.jp/20190523.html
DRAサイト: https://www.ddbj.nig.ac.jp/dra/submission.html#dra-data-submission
登録するものの構造としては
BioProject (Study)
研究プロジェクトの内容
「なぜ」そのサンプルをシークエンスしたのか
BioSample (Sample)
生物学的、物理的にユニークなサンプル
「何を」シークエンスしたのか
をまず登録する。BioProjectの登録はクリックとタイプだけで終了するので超簡単だが、BioSampleはなかなか複雑である。メタデータと呼ばれるサンプル情報を明確に、しかもルールに沿って記載する必要がある。下記の「メタデータについて」と「登録例」を擦り倒すくらい読んで準備しておくと良い。BioSampleはfastq登録の数少ない関門なので、ここを乗り越えるとあとはスムーズにいく。
メタデータについて: https://www.ddbj.nig.ac.jp/biosample/submission.html#metadata
メタデータ登録例: https://www.ddbj.nig.ac.jp/biosample/attribute.html
そして、次のステップでは、これらを
DRA Experiment
特定のサンプルから構築したライブラリーについての説明
「どのように」シークエンスをしたのか
複数の Experiment は一つの Sample を参照できるが、逆はできない
DRA Run
Experiment と Run を投稿した後、データファイルの検証処理を開始
Run にリンクしている全てのデータファイルは 1 つの SRA ファイルにマージされます
とリンクさせる。ここで要注意だが、ここより先の作業はGoogle Chromeでは行うことができないっぽい。筆者はSafariで行ったが、対応しているもので作業するといい。
Fastqの生データとドラフトゲノム の登録のどちらを先にやるべきかと悩むが、当方は今後Fastqから登録することに決めた。理由はFastqから登録して、BioProject (Study)やBioSample (Sample)を作ればFastaファイルを登録する際にも用いることができるからである。さらに、ゲノムサイズのデータ登録をする際はMSSを利用する必要があり、時間がかかるため多分先にFastqファイルを登録するのが正解。なお、特にDRA Experimentなどについて言えることだが、シーケンスの方法について細かく聞かれるので実験ノートを手元に置いておくか、シーケンスに詳しい人に聞いておくといい。あらかじめ、埋めるフォーマットを見ておくと必要な項目がわかる。
TogoTV通りに行なっていくと、だいたい1時間くらいで作業を終えることができた。非常にわかりやすかった。Submissionを終えてから1週間ほどでDDBJの職員様から返信をいただき、登録完了のお知らせをいただいた。データについては登録しても、指定した日までConfidentialにできるので、論文に掲載したい場合は早めに登録しておくといい。
ちなみにDRAの方も使用できる用語は決められている。こちらもよく読んで、わからなければメールで聞く形で進めるといい。
用語: https://www.ddbj.nig.ac.jp/dra/submission.html#how-to-submit-dra-data
続いてMSS (Fasta登録)
続いてドラフトゲノムを登録していく。MSSはMass Submission Systemのことで、簡単に言えばでかいデータを登録するときに利用するシステムらしい。MSSよりもお手軽に利用できるNSSSはバクテリアゲノムデータにはサイズ的に利用不可能となっている。
配列が長い(目安は 500 kb 以上)
エントリあたりに多数(概ね30以上)のFeature がある
WGS, CON, TSA, TLS, HTC, HTG, EST, GSS, STS の登録
に該当したら諦めてMSSを利用する。MSSはメールでのやり取りを行うシステムで、職員さんと協力して登録するような感じになっている。
MSSについて: https://www.ddbj.nig.ac.jp/ddbj/mss.html
MSSによる登録は第二関門である。Fastqファイルの登録と違う点はファイルの準備が正直めんどくさいことである。規定がたくさんあり、非常に難しい。そこで、利用できるのがDFASTである。
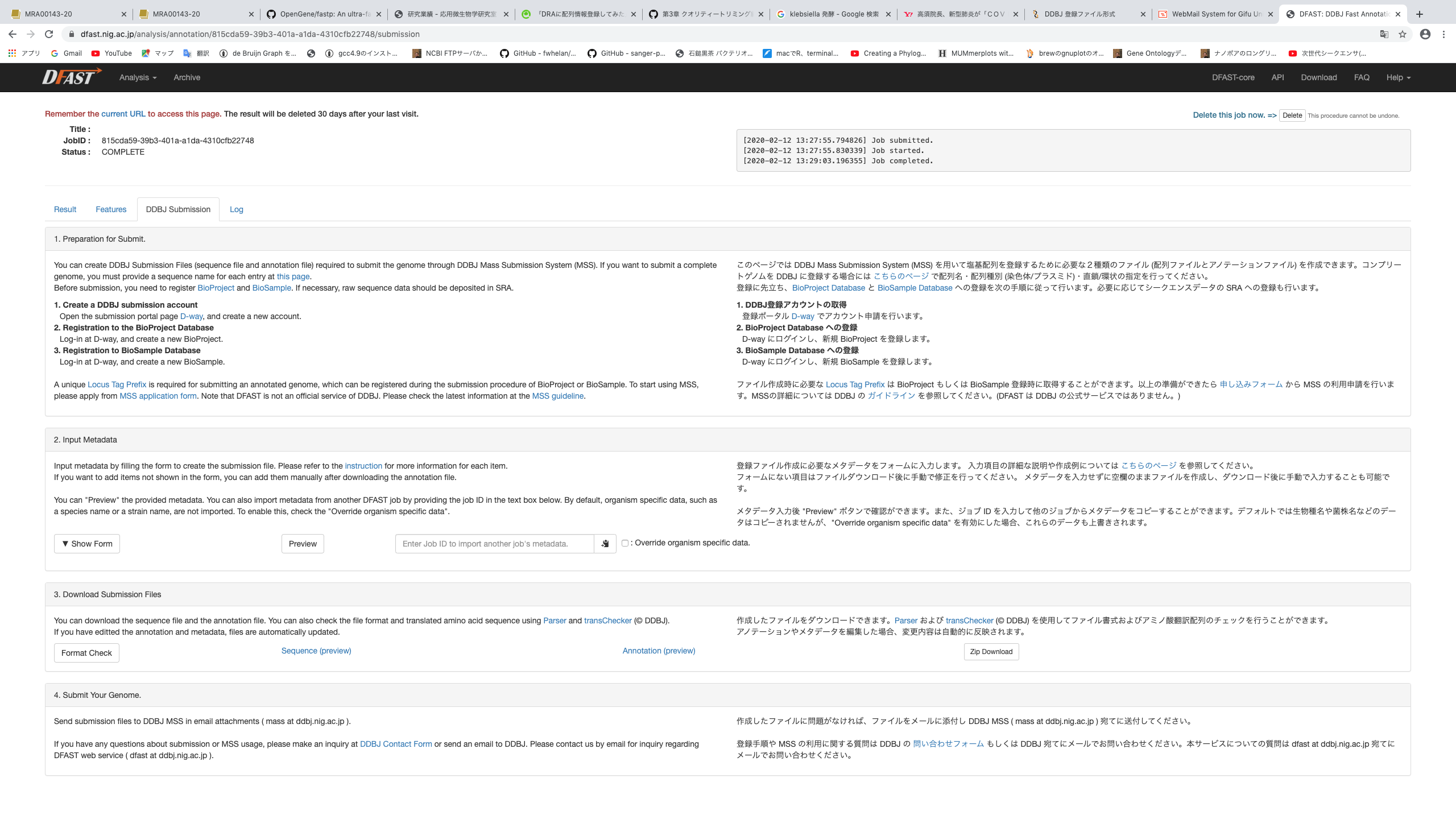
上記はアノテーション後のサイトの様子である。DDBJ submission用にフォームがあり、丁寧に流れまで記載してくださっている。特筆すべきはParser および transCheckerを自動でやってくれるところである。ボタンを押せばダメなところを教えてくれる。Parser および transCheckerについてはMSSのURL参照。この際BioProjectとBioSamole、SRA番号が必要になる。したがって、あらかじめFastqファイルを登録する感じが正しいのだと思う。
注意ポイントとしては、筆者の名前の書き方が決まっているので、例をよく見て書くことくらいだろうか。後は、Genome Coverageはシーケンスにより得られた総塩基数をゲノムサイズで割って計算すれば良いらしい。ただ概算値を記載するルールなので、「整数で」とのことである。やり方としてはfastpでも出力されるし、seqkit statsも使えそうである。
もしもMRAにて論文を出す予定があれば、MRA用の原稿のうち方法論のところを先に書いて、データを登録、その間に残りを仕上げるという形にすればスムーズに行きそうである。
まとめ
- 論文を出す前に登録を終えておくと便利。
- FASTQ -> ドラフトゲノム
- DFASTを使うと楽にゲノム登録できる。(この方法は推奨されている)