cdd2cog
参考: https://github.com/aleimba/bac-genomics-scripts/tree/master/cdd2cog
The script assigns COG (cluster of orthologous groups) categories to proteins. For this purpose, the query proteins need to be blasted with RPS-BLAST+ (Reverse Position-Specific BLAST) against NCBI's Conserved Domain Database (CDD). Use cds_extractor.pl beforehand to extract multi-fasta protein files from GENBANK or EMBL files.
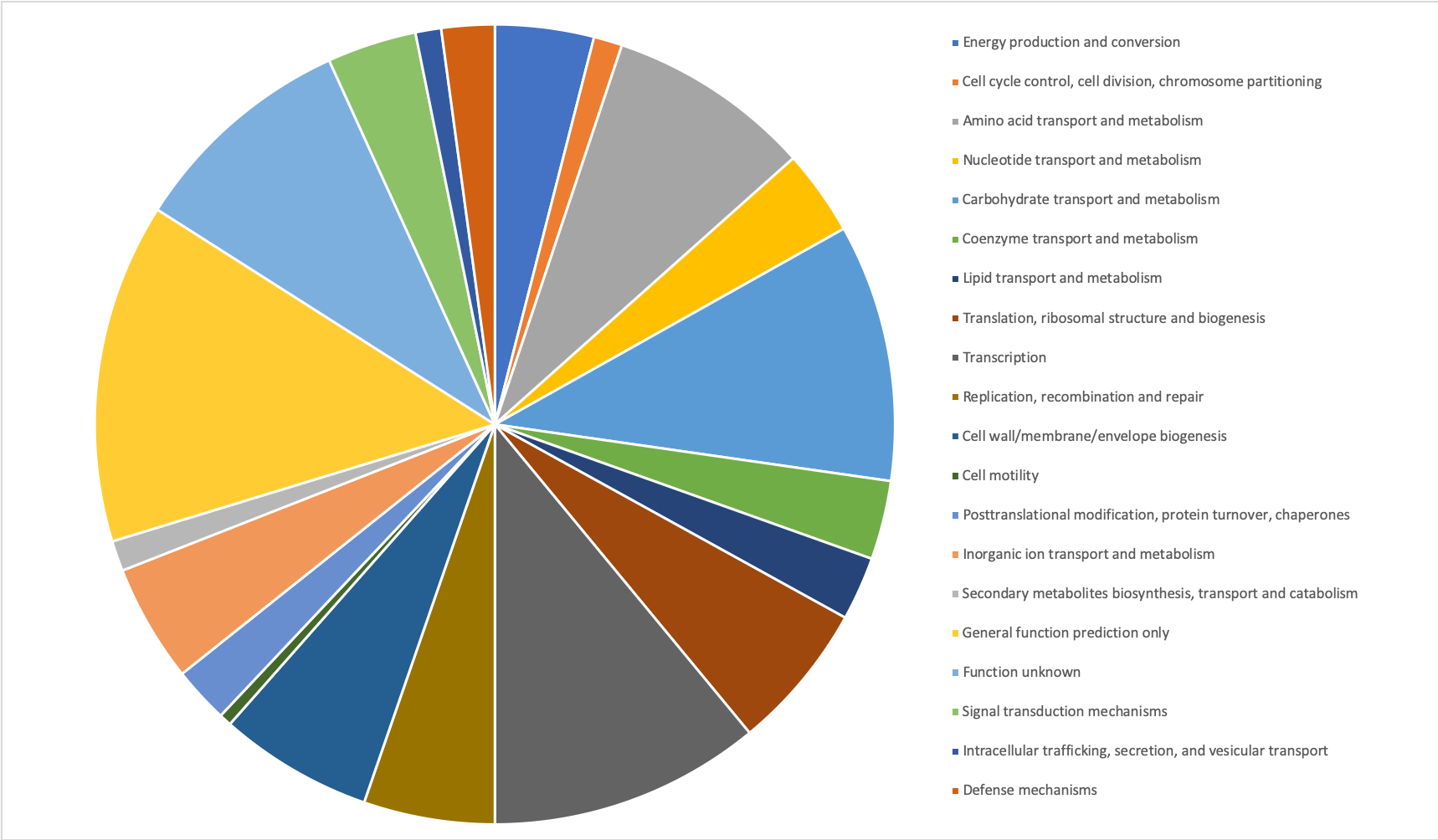
ネットによればCOGとは
COGは全ゲノムにコードされているタンパク質配列の全てのオーソログを定義しているデータベースのこと。オーソログクラスターに含まれる生物種とタンパク質名、系統樹を収録している。各クラスターは、個々のタンパクやパラログから構成されており、保存ドメインと一致する。
オーソログといえば、種分岐によって共通の祖先遺伝子から生じた相同な遺伝子のことだが、これを利用するメリットはCOGを利用することでおおよそどの様な機能を持った遺伝子があるかを推定できる点にある。当然全てがわかるわけでは無いものの、おおよその特徴を知る点では有意義な解析とも言える。今回はこれを用いて、アノテーションされた遺伝子を大まかにクラスタリングし、機能性を考えるということをする。
やり方
流れとしてはRPS-BLASTをCOGデータベースに対して行い、Perlのスクリプトを動かして、分類を行うという感じである。
# Perlスクリプトダウンロード
wget https://raw.githubusercontent.com/aleimba/bac-genomics-scripts/master/cdd2cog/cdd2cog.pl
# NCBIのConserved Domain Databaseの情報
wget ftp://ftp.ncbi.nlm.nih.gov/pub/mmdb/cdd/cddid.tbl.gz
gunzip cddid.tbl.gz
wget ftp://ftp.ncbi.nlm.nih.gov/pub/mmdb/cdd/little_endian/Cog_LE.tar.gz
tar xvfz Cog_LE.tar.gz
## 分類分けなどの情報
wget ftp://ftp.ncbi.nlm.nih.gov/pub/COG/COG/fun.txt
wget ftp://ftp.ncbi.nlm.nih.gov/pub/COG/COG/whog
# rpsblast+
rpsblast -query hoge.faa -db Cog -out rps-blast.out -evalue 1e-2 -outfmt 6
利用するデータはDFASTなどで作成。Protein.faaを用いると良い。
# cdd2cog
perl cdd2cog.pl -r rps-blast.out -c cddid.tbl -f fun.txt -w whog
注意点
cdd2cog.plには欠陥が報告されており、cdd2cog.plの302行目を
my $pssm_id = $1 if $line[1] =~ /^gnl\|CDD\|(\d+)/; # get PSSM-Id from the subject hit
に変更しないとうまく作動しない。subject idに何かしらのエラーが起きてしまう様である。
DFASTでは直接RPS-BLAST+がかけられる
多分、Genbank形式などから直接outファイルが作れるのだろうが、わからなかった。そのうち公開予定である。