Day4: 機械学習 Advent Calendar 2024
「不均衡データ処理」シリーズの2つ目の記事です。
1: 不均衡データ処理【概要】
2: 不均衡データ処理【オーバーサンプリング】
3: 不均衡データ処理【アンダーサンプリング】 / coming soon
4: 不均衡データ処理【ハイブリッド手法】 / coming soon
5: 未定
② オーバーサンプリングの理論と比較
オーバーサンプリングとは
オーバーサンプリングは、少数派クラスのデータを増やして、クラス間のバランスを取るための手法である。
クラス不均衡の問題が存在する場合、モデルは多数派クラスに偏った予測を行う傾向があり、少数派クラスの精度が低下する。
この問題を解決するために、少数派クラスのサンプルを増加させることで、モデルが少数派クラスに対しても適切に学習できるようにする。
代表的なオーバーサンプリング手法
オーバーサンプリング手法にはさまざまな方法が存在する。代表的なものを以下に紹介する
-
SMOTE(Synthetic Minority Over-sampling Technique)
SMOTEは、少数派クラスのデータポイントの近傍に位置する新しいサンプルを合成する手法である
ランダムに選ばれた少数派クラスのデータポイントのペアから新しいデータ点を生成するため、単純なコピーではなく、データの多様性を維持しながら増加させる -
ADASYN(Adaptive Synthetic Sampling)
ADASYNは、SMOTEの進化版であり、学習が難しいデータポイントに焦点を当ててサンプルを生成する
具体的には、少数派クラスの中でも、近くに他のサンプルが少ない(もしくはデータが分散している)領域を特定し、その周辺のサンプルを合成することで、学習の効率を高める -
Borderline-SMOTE
Borderline-SMOTEは、SMOTEの亜種であり、特に決定境界付近のデータポイントに焦点を当ててサンプルを合成する手法である
決定境界に近いサンプルは、モデルにとって重要であるため、これらのサンプルに対して新しいデータを生成することで、モデルの学習を強化する
なお、SMOTEには様々な亜種がある。
100種以上のSMOTEの亜種をまとめた論文1があるそうだが、私は読んでいない。
オーバーサンプリングの理論
オーバーサンプリングは、主に以下の理論に基づいている:
-
データの分布均一化:
少数派クラスのサンプルが増えることで、データセット内のクラスの分布が均等に近づき、学習がバランスよく行えるようになる -
近傍データの利用:
オーバーサンプリングの多くの手法は、既存の少数派クラスのサンプルを基に新しいデータポイントを生成する。これにより、少数派クラスのデータが増加するが、単純なコピーではなく、新しい情報が加わることで過学習のリスクを減少させる
SMOTE
SMOTE (Synthetic Minority Over-sampling Technique) は、以下の数式で表現される。
$$
\mathbf{x'} = \mathbf{x_i} + \lambda (\mathbf{x_j} - \mathbf{x_i})
$$
ここで、$\mathbf{x_i}$ と $\mathbf{x_j}$ は少数派クラスのデータポイントで、$\lambda$ はランダムに選ばれたスカラー値(0から1の間)である。$\mathbf{x'}$ は新しく生成されたサンプルで、$\mathbf{x_i}$ と $\mathbf{x_j}$ の間に位置する。
from imblearn.over_sampling import SMOTE
smote = SMOTE(random_state=42)
X_smote, y_smote = smote.fit_resample(X, y)
実装に関する詳細は以下
ADASYN
ADASYN (Adaptive Synthetic Sampling) は、SMOTEに基づいた手法であり、クラスの不均衡がより大きい少数派クラスに焦点を当てている。ADASYNは、学習が難しいサンプルや、境界付近のサンプルに対して合成データを重点的に生成する。これにより、少数派クラスのサンプルがより効果的に分布するように生成される。
ADASYNの生成方法は、SMOTEの方法と似ているが、重要な違いは、合成サンプルを生成する際に、境界付近のサンプルに対して重みを付けて生成される点である。これにより、少数派クラスのサンプルがより均等に分布することを目指す。
$$
\mathbf{x'} = \mathbf{x_i} + \lambda (\mathbf{x_j} - \mathbf{x_i})
$$
ここで、$\mathbf{x_i}$ と $\mathbf{x_j}$ は少数派クラスのデータポイントで、$\lambda$ はランダムに選ばれたスカラー値(0から1の間)である。ADASYNでは、重要なサンプルに対して多くの合成データが生成される。
from imblearn.over_sampling import ADASYN
adasyn = ADASYN(random_state=42)
X_adasyn, y_adasyn = adasyn.fit_resample(X, y)
実装に関する詳細は以下
BorderlineSMOTE
BorderlineSMOTE は、SMOTEの改良版であり、特に決定境界付近にあるサンプルに注目して新しいデータを生成する手法である。通常のSMOTEではランダムに近隣サンプルを選んで合成データを生成するが、BorderlineSMOTEは少数派クラスの決定境界に近いサンプルに重点を置いている。
この手法は、クラスの境界付近で誤分類されやすいサンプルを優先的に選び、その周囲に新たなサンプルを生成することにより、より効果的なデータ増強を行う。これにより、分類モデルは境界付近の難しいサンプルに対しても強化され、精度の向上が期待される。
$$
\mathbf{x'} = \mathbf{x_i} + \lambda (\mathbf{x_j} - \mathbf{x_i})
$$
ここで、$\mathbf{x_i}$ と $\mathbf{x_j}$ は決定境界に近い少数派クラスのデータポイントで、$\lambda$ はランダムに選ばれたスカラー値(0から1の間)であり、新しいサンプル$\mathbf{x'}$ を生成する。
from imblearn.over_sampling import BorderlineSMOTE
borderline_smote = BorderlineSMOTE(random_state=42)
X_borderline_smote, y_borderline_smote = borderline_smote.fit_resample(X, y)
実装に関する詳細は以下
オーバーサンプリング前後のモデル性能
概要
オーバーサンプリングを適用した場合と適用しなかった場合で、モデルの性能にどのような違いが出るかを実際に比較してみる。
ここでは、3つの異なるデータを使用して、以下のような手順で性能を比較する。
データについては、当シリーズの最初の記事を見ていただきたい。
-
モデル構築:
- 各データセットに対して、
RandomForestClassifierを用いて訓練
- 各データセットに対して、
-
性能評価指標:
- accuracy
- precision
- recall
- f1-score
オーバーサンプリングを行うことで、少数派クラスの予測精度が向上することが期待される。また、特に少数派クラスが重要なタスクにおいては、オーバーサンプリング手法が有効であると考えられる。
具体的には、以下のような結果が期待される。
- オーバーサンプリング前: 少数派クラスの精度が低く、多数派クラスに偏った予測がされる
- オーバーサンプリング後: 少数派クラスの予測精度が向上し、全体的な精度も改善される。特にF1スコアやROC-AUCの指標で改善が見られる
実験結果
Sample 1
分布図
性能比較
python
import numpy as np
import matplotlib.pyplot as plt
from imblearn.over_sampling import SMOTE, ADASYN, BorderlineSMOTE
from sklearn.model_selection import train_test_split
from sklearn.ensemble import RandomForestClassifier
from sklearn.metrics import classification_report
mean_0 = [2, 0]
mean_1 = [5, 0]
cov_0 = [[1, 0], [0, 50]]
cov_1 = [[0.1, 0], [0, 100]]
X_class_0 = np.random.multivariate_normal(mean_0, cov_0, 800)
X_class_1 = np.random.multivariate_normal(mean_1, cov_1, 200)
y_class_0 = np.zeros(800)
y_class_1 = np.ones(200)
X = np.vstack([X_class_0, X_class_1])
y = np.hstack([y_class_0, y_class_1])
X += np.random.normal(0, 1.0, X.shape)
class_0_count = 960
class_1_count = 240
class_0_indices = np.where(y == 0)[0]
class_1_indices = np.where(y == 1)[0]
sampled_class_0_indices = np.random.choice(class_0_indices, size=class_0_count, replace=True)
sampled_class_1_indices = np.random.choice(class_1_indices, size=class_1_count, replace=True)
X_resampled = np.vstack([X[sampled_class_0_indices], X[sampled_class_1_indices]])
y_resampled = np.hstack([y[sampled_class_0_indices], y[sampled_class_1_indices]])
smote = SMOTE(random_state=42)
X_smote, y_smote = smote.fit_resample(X, y)
adasyn = ADASYN(random_state=42)
X_adasyn, y_adasyn = adasyn.fit_resample(X, y)
borderline_smote = BorderlineSMOTE(random_state=42)
X_borderline_smote, y_borderline_smote = borderline_smote.fit_resample(X, y)
def train_and_evaluate(X_train, y_train, X_test, y_test):
model = RandomForestClassifier(n_estimators=100, random_state=42)
model.fit(X_train, y_train)
y_pred = model.predict(X_test)
return classification_report(y_test, y_pred)
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.3, random_state=42)
smote = SMOTE(random_state=42)
X_smote, y_smote = smote.fit_resample(X, y)
adasyn = ADASYN(random_state=42)
X_adasyn, y_adasyn = adasyn.fit_resample(X, y)
borderline_smote = BorderlineSMOTE(random_state=42)
X_borderline_smote, y_borderline_smote = borderline_smote.fit_resample(X, y)
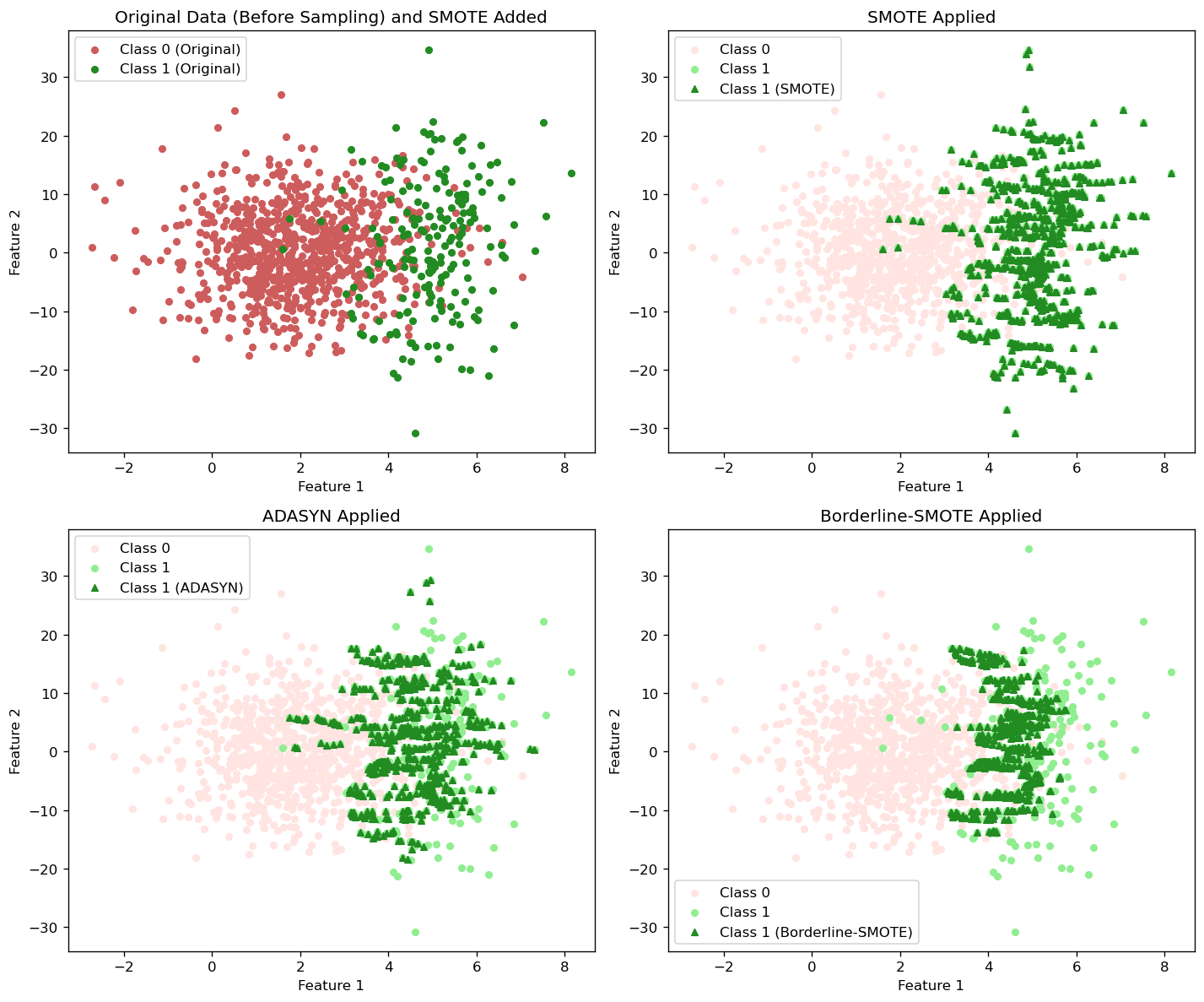
plt.figure(figsize=(12, 10), dpi=120)
smote_class_1_indices = np.where(y_smote == 1)[0]
plt.subplot(2, 2, 1)
plt.scatter(X[y == 0][:, 0], X[y == 0][:, 1], color='indianred', label='Class 0 (Original)', s=20)
plt.scatter(X[y == 1][:, 0], X[y == 1][:, 1], color='forestgreen', label='Class 1 (Original)', s=20)
plt.title("Original Data (Before Sampling) and SMOTE Added")
plt.xlabel("Feature 1")
plt.ylabel("Feature 2")
plt.legend()
plt.subplot(2, 2, 2)
plt.scatter(X_smote[y_smote == 0][:, 0], X_smote[y_smote == 0][:, 1], color='mistyrose', label='Class 0', s=20)
plt.scatter(X_smote[y_smote == 1][:, 0], X_smote[y_smote == 1][:, 1], color='lightgreen', label='Class 1', s=20)
plt.scatter(X_smote[smote_class_1_indices, 0], X_smote[smote_class_1_indices, 1], color='forestgreen', marker='^', label='Class 1 (SMOTE)', s=20)
plt.title("SMOTE Applied")
plt.xlabel("Feature 1")
plt.ylabel("Feature 2")
plt.legend()
plt.subplot(2, 2, 3)
plt.scatter(X_adasyn[y_adasyn == 0][:, 0], X_adasyn[y_adasyn == 0][:, 1], color='mistyrose', label='Class 0', s=20)
plt.scatter(X_adasyn[y_adasyn == 1][:, 0], X_adasyn[y_adasyn == 1][:, 1], color='lightgreen', label='Class 1', s=20)
plt.scatter(X_adasyn[len(X):, 0], X_adasyn[len(X):, 1], color='forestgreen', marker='^', label='Class 1 (ADASYN)', s=20)
plt.title("ADASYN Applied")
plt.xlabel("Feature 1")
plt.ylabel("Feature 2")
plt.legend()
plt.subplot(2, 2, 4)
plt.scatter(X_borderline_smote[y_borderline_smote == 0][:, 0], X_borderline_smote[y_borderline_smote == 0][:, 1], color='mistyrose', label='Class 0', s=20)
plt.scatter(X_borderline_smote[y_borderline_smote == 1][:, 0], X_borderline_smote[y_borderline_smote == 1][:, 1], color='lightgreen', label='Class 1', s=20)
plt.scatter(X_borderline_smote[len(X):, 0], X_borderline_smote[len(X):, 1], color='forestgreen', marker='^', label='Class 1 (Borderline-SMOTE)', s=20)
plt.title("Borderline-SMOTE Applied")
plt.xlabel("Feature 1")
plt.ylabel("Feature 2")
plt.legend()
plt.tight_layout()
plt.show()
metrics_data = {}
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.3, random_state=42)
model = RandomForestClassifier(n_estimators=100, random_state=42)
model.fit(X_train, y_train)
y_pred = model.predict(X_test)
report = classification_report(y_test, y_pred, output_dict=True)
metrics_data["Original"] = {
"precision": [report['0.0']['precision'], report['1.0']['precision']],
"recall": [report['0.0']['recall'], report['1.0']['recall']],
"f1-score": [report['0.0']['f1-score'], report['1.0']['f1-score']],
}
for sampling_name, X_resampled, y_resampled in [
("SMOTE", X_smote, y_smote),
("ADASYN", X_adasyn, y_adasyn),
("Borderline-SMOTE", X_borderline_smote, y_borderline_smote)
]:
X_train, X_test, y_train, y_test = train_test_split(X_resampled, y_resampled, test_size=0.3, random_state=42)
model = RandomForestClassifier(n_estimators=100, random_state=42)
model.fit(X_train, y_train)
y_pred = model.predict(X_test)
report = classification_report(y_test, y_pred, output_dict=True)
metrics_data[sampling_name] = {
"precision": [report['0.0']['precision'], report['1.0']['precision']],
"recall": [report['0.0']['recall'], report['1.0']['recall']],
"f1-score": [report['0.0']['f1-score'], report['1.0']['f1-score']],
}
class_labels = ["Class 0", "Class 1"]
metrics = ["precision", "recall", "f1-score"]
x = np.arange(len(class_labels))
colors = ['b', 'g', 'r', 'c']
line_styles = ['-', '--', '-.', ':']
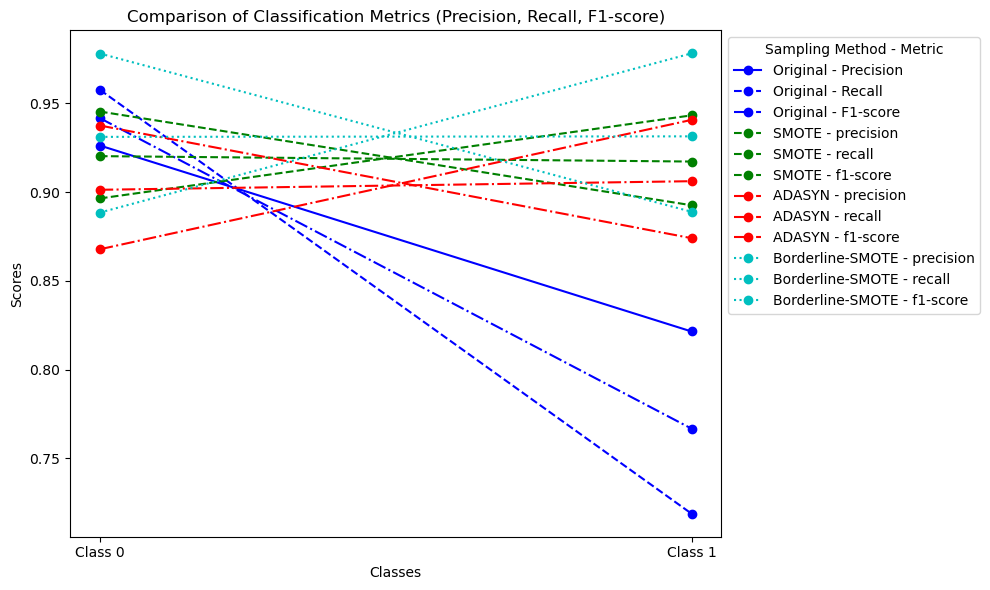
fig, ax = plt.subplots(figsize=(10, 6))
ax.plot(x, metrics_data["Original"]["precision"], label="Original - Precision", color='b', linestyle='-', marker='o')
ax.plot(x, metrics_data["Original"]["recall"], label="Original - Recall", color='b', linestyle='--', marker='o')
ax.plot(x, metrics_data["Original"]["f1-score"], label="Original - F1-score", color='b', linestyle='-.', marker='o')
for i, (model_name, metrics_data_item) in enumerate(metrics_data.items()):
if model_name != "Original":
for j, metric in enumerate(metrics):
ax.plot(x, metrics_data_item[metric], label=f"{model_name} - {metric}", color=colors[i], linestyle=line_styles[i], marker='o')
ax.set_xlabel("Classes")
ax.set_ylabel("Scores")
ax.set_title("Comparison of Classification Metrics (Precision, Recall, F1-score)")
ax.set_xticks(x)
ax.set_xticklabels(class_labels)
ax.legend(title="Sampling Method - Metric", loc="upper left", bbox_to_anchor=(1, 1))
plt.tight_layout()
plt.show()
Sample 2
分布図
性能比較
Original Data Evaluation
precision recall f1-score support
0 0.86 0.96 0.91 470
1 0.73 0.44 0.55 130
accuracy 0.84 600
macro avg 0.80 0.70 0.73 600
weighted avg 0.83 0.84 0.83 600
SMOTE Evaluation
Resampled dataset distribution: Counter({0: 1600, 1: 1600})
precision recall f1-score support
0 0.90 0.89 0.89 498
1 0.88 0.89 0.88 462
accuracy 0.89 960
macro avg 0.89 0.89 0.89 960
weighted avg 0.89 0.89 0.89 960
ADASYN Evaluation
Resampled dataset distribution: Counter({0: 1600, 1: 1560})
precision recall f1-score support
0 0.89 0.89 0.89 492
1 0.88 0.88 0.88 456
accuracy 0.89 948
macro avg 0.89 0.89 0.89 948
weighted avg 0.89 0.89 0.89 948
BorderlineSMOTE Evaluation
Resampled dataset distribution: Counter({0: 1600, 1: 1600})
precision recall f1-score support
0 0.90 0.90 0.90 498
1 0.89 0.89 0.89 462
accuracy 0.89 960
macro avg 0.89 0.89 0.89 960
weighted avg 0.89 0.89 0.89 960
python
# Import necessary libraries
import numpy as np
import matplotlib.pyplot as plt
from imblearn.over_sampling import SMOTE, ADASYN, BorderlineSMOTE
from sklearn.model_selection import train_test_split
from sklearn.ensemble import RandomForestClassifier
from sklearn.metrics import classification_report
from sklearn.datasets import make_moons
from collections import Counter
np.random.seed(42)
X, y = make_moons(n_samples=2000, noise=1.00, random_state=42)
class_0_count = 1600
class_1_count = 400
class_0_indices = np.where(y == 0)[0]
class_1_indices = np.where(y == 1)[0]
sampled_class_0_indices = np.random.choice(class_0_indices, class_0_count, replace=True)
sampled_class_1_indices = np.random.choice(class_1_indices, class_1_count, replace=True)
X_resampled = np.vstack([X[sampled_class_0_indices], X[sampled_class_1_indices]])
y_resampled = np.hstack([y[sampled_class_0_indices], y[sampled_class_1_indices]])
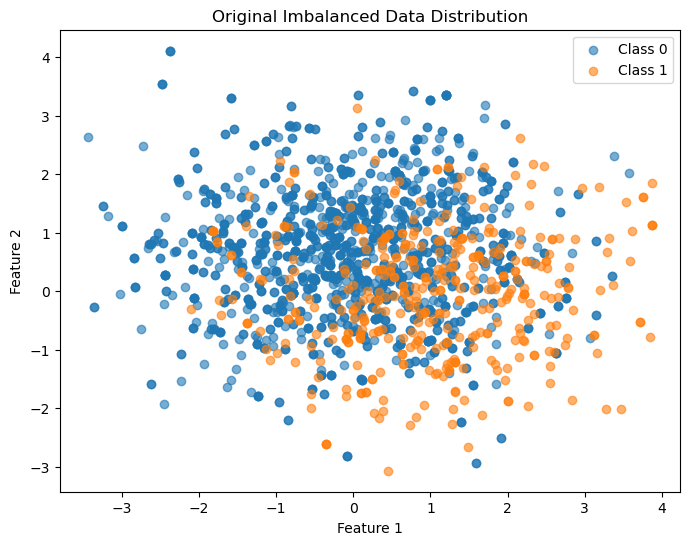
plt.figure(figsize=(8, 6))
plt.scatter(X_resampled[y_resampled == 0][:, 0], X_resampled[y_resampled == 0][:, 1], label="Class 0", alpha=0.6)
plt.scatter(X_resampled[y_resampled == 1][:, 0], X_resampled[y_resampled == 1][:, 1], label="Class 1", alpha=0.6)
plt.title("Original Imbalanced Data Distribution")
plt.xlabel("Feature 1")
plt.ylabel("Feature 2")
plt.legend()
plt.show()
def evaluate_original(X, y):
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.3, random_state=42)
clf = RandomForestClassifier(random_state=42)
clf.fit(X_train, y_train)
y_pred = clf.predict(X_test)
print(classification_report(y_test, y_pred))
print("\nOriginal Data Evaluation")
evaluate_original(X_resampled, y_resampled)
def evaluate_oversampling(method, X, y):
X_res, y_res = method.fit_resample(X, y)
print(f"Resampled dataset distribution: {Counter(y_res)}")
X_train, X_test, y_train, y_test = train_test_split(X_res, y_res, test_size=0.3, random_state=42)
clf = RandomForestClassifier(random_state=42)
clf.fit(X_train, y_train)
y_pred = clf.predict(X_test)
print(classification_report(y_test, y_pred))
print("\nSMOTE Evaluation")
smote = SMOTE(random_state=42)
evaluate_oversampling(smote, X_resampled, y_resampled)
print("\nADASYN Evaluation")
adasyn = ADASYN(random_state=42)
evaluate_oversampling(adasyn, X_resampled, y_resampled)
print("\nBorderlineSMOTE Evaluation")
borderline_smote = BorderlineSMOTE(random_state=42)
evaluate_oversampling(borderline_smote, X_resampled, y_resampled)
Sample 3
分布図
性能比較
Original Data Evaluation
precision recall f1-score support
0.0 0.94 0.96 0.95 294
1.0 0.82 0.74 0.78 66
accuracy 0.92 360
macro avg 0.88 0.85 0.87 360
weighted avg 0.92 0.92 0.92 360
SMOTE Evaluation
Resampled dataset distribution: Counter({0.0: 960, 1.0: 960})
precision recall f1-score support
0.0 0.92 0.94 0.93 280
1.0 0.94 0.92 0.93 296
accuracy 0.93 576
macro avg 0.93 0.93 0.93 576
weighted avg 0.93 0.93 0.93 576
ADASYN Evaluation
Resampled dataset distribution: Counter({1.0: 974, 0.0: 960})
precision recall f1-score support
0.0 0.89 0.94 0.91 286
1.0 0.94 0.88 0.91 295
accuracy 0.91 581
macro avg 0.91 0.91 0.91 581
weighted avg 0.91 0.91 0.91 581
BorderlineSMOTE Evaluation
Resampled dataset distribution: Counter({0.0: 960, 1.0: 960})
precision recall f1-score support
0.0 0.93 0.93 0.93 280
1.0 0.94 0.93 0.93 296
accuracy 0.93 576
macro avg 0.93 0.93 0.93 576
weighted avg 0.93 0.93 0.93 576
python
import numpy as np
import matplotlib.pyplot as plt
from imblearn.over_sampling import SMOTE, ADASYN, BorderlineSMOTE
from sklearn.model_selection import train_test_split
from sklearn.ensemble import RandomForestClassifier
from sklearn.metrics import classification_report
from collections import Counter
r_0 = np.linspace(0.5, 1.5, 600)
theta_0 = np.linspace(0, 2 * np.pi, 600)
x_0 = r_0 * np.cos(theta_0)
y_0 = r_0 * np.sin(theta_0)
r_1 = np.linspace(1.5, 2.5, 600)
theta_1 = np.linspace(0, 2 * np.pi, 600)
x_1 = r_1 * np.cos(theta_1)
y_1 = r_1 * np.sin(theta_1)
x_0 += np.random.normal(0, 0.8, 600)
y_0 += np.random.normal(0, 0.2, 600)
x_1 += np.random.normal(0, 0.4, 600)
y_1 += np.random.normal(0, 0.6, 600)
X = np.vstack([np.column_stack([x_0, y_0]), np.column_stack([x_1, y_1])])
y = np.hstack([np.zeros(600), np.ones(600)])
class_0_count = 960
class_1_count = 240
class_0_indices = np.where(y == 0)[0]
class_1_indices = np.where(y == 1)[0]
sampled_class_0_indices = np.random.choice(class_0_indices, size=class_0_count, replace=True)
sampled_class_1_indices = np.random.choice(class_1_indices, size=class_1_count, replace=True)
X_resampled = np.vstack([X[sampled_class_0_indices], X[sampled_class_1_indices]])
y_resampled = np.hstack([y[sampled_class_0_indices], y[sampled_class_1_indices]])
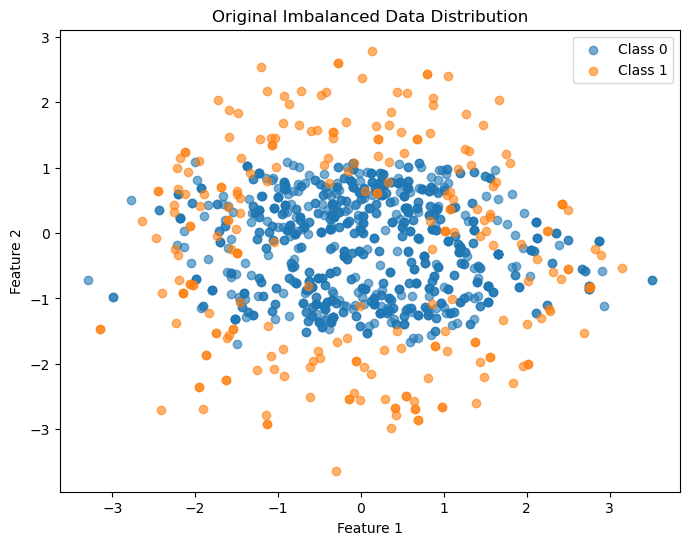
plt.figure(figsize=(8, 6))
plt.scatter(X_resampled[y_resampled == 0][:, 0], X_resampled[y_resampled == 0][:, 1], label="Class 0", alpha=0.6)
plt.scatter(X_resampled[y_resampled == 1][:, 0], X_resampled[y_resampled == 1][:, 1], label="Class 1", alpha=0.6)
plt.title("Original Imbalanced Data Distribution")
plt.xlabel("Feature 1")
plt.ylabel("Feature 2")
plt.legend()
plt.show()
def evaluate_original(X, y):
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.3, random_state=42)
clf = RandomForestClassifier(random_state=42)
clf.fit(X_train, y_train)
y_pred = clf.predict(X_test)
print(classification_report(y_test, y_pred))
print("\nOriginal Data Evaluation")
evaluate_original(X_resampled, y_resampled)
def evaluate_oversampling(method, X, y):
X_res, y_res = method.fit_resample(X, y)
print(f"Resampled dataset distribution: {Counter(y_res)}")
X_train, X_test, y_train, y_test = train_test_split(X_res, y_res, test_size=0.3, random_state=42)
clf = RandomForestClassifier(random_state=42)
clf.fit(X_train, y_train)
y_pred = clf.predict(X_test)
print(classification_report(y_test, y_pred))
print("\nSMOTE Evaluation")
smote = SMOTE(random_state=42)
evaluate_oversampling(smote, X_resampled, y_resampled)
print("\nADASYN Evaluation")
adasyn = ADASYN(random_state=42)
evaluate_oversampling(adasyn, X_resampled, y_resampled)
print("\nBorderlineSMOTE Evaluation")
borderline_smote = BorderlineSMOTE(random_state=42)
evaluate_oversampling(borderline_smote, X_resampled, y_resampled)
5. オーバーサンプリングが適している場合
オーバーサンプリングは、特に以下のような場合に有効である。
-
少数派クラスが重要な場合:
少数派クラスの予測精度が特に重要なタスク(例: 医療診断、詐欺検出)では、オーバーサンプリングが有効である。少数派クラスを増加させることで、モデルがそれに注目し、より正確な予測ができるようになる -
クラス不均衡が大きい場合:
クラス不均衡が極端な場合、モデルは多数派クラスに偏りがちである。オーバーサンプリングによって、少数派クラスのサンプルを増やし、バランスを取ることができる -
過学習に注意が必要な場合:
オーバーサンプリングを行う際は過学習のリスクがあるため、手法によっては、適切な正則化やクロスバリデーションを行うことが重要である
-
Fernández, A., Garcia, S., Herrera, F., & Chawla, N. V. (2018). SMOTE for learning from imbalanced data: progress and challenges, marking the 15-year anniversary. Journal of artificial intelligence research, 61, 863-905. ↩