分子の重ね合わせ表示
有機構造化学は有機物の結晶構造とその物性について検討する学問です。有機構造化学では、結晶中の分子同士の形状を比較したいってことがよくあります。比較したい分子同士の結合長やら二面角やら数値を比較しても良いのですが、二つの分子の三次元画像を重ねて表示しちゃえば直感的にもわかりやすいです。この分子の重ね合わせ表示の図は論文とかではよく見るのですが、自分でやろうとすると思ったような描画ができずなかなか大変です。
そこで、ここでは、有機構造化学の分野でよく用いられる結晶構造データであるcifファイルから分子一個の立体構造データであるmolファイルを取り出しpythonを用いて三次元の重ね合わせ表示を行う方法をまとめます。
本記事の内容は無料でできるのが強みです。また、pythonによる描画なので色、大きさ、表示形式、重ね合わせ方法など設定の自由度が高いです。しかし、初めてpythonを扱う場合、環境構築が恐ろしく大変なのが短所です。
インプットとアウトプット
今回のデモンストレーションにおけるインプットデータとアウトプットデータを示します。
・インプット
→結晶構造データ(.cif)
1)アスピリン1
2)イブプロフェン2
結晶構造はCrystallography Open Database(COD)3から無料でダウンロードできます。ここで、アスピリン、イブプロフェンは共に解熱鎮痛剤の薬効物質です。CODからダウンロードした二つの結晶構造を以下に示します。
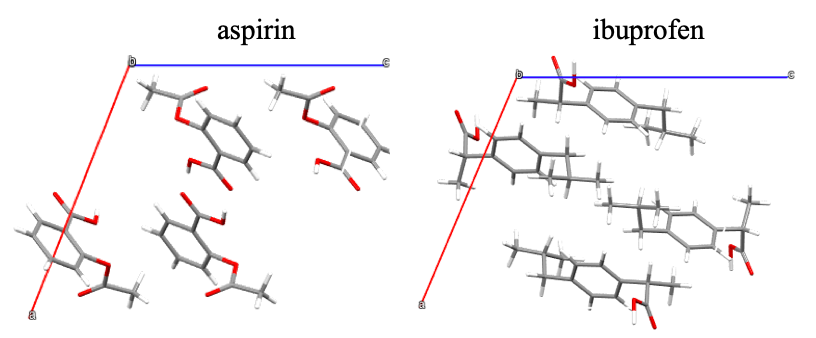
アスピリンとイブプロフェンの重ね合わせ画像はこんな感じです。
使用application
・Mercury
→The Cambridge Crystallographic Data Centre(CCDC)が提供する結晶構造解析アプリ。注釈URLにて無料ダウンロード可能4。
・JupyterLab
→今回pythonを動かす実行環境です。自分はpythonの実行結果がすぐ見れるエディタというくらいの認識しかないです。Anacondaというアプリケーションをダウンロードすると使えるようになります。Anacondaを下記URLにて無料ダウンロード可能5。
準備
・pythonライブラリのインストール
1)Rdkit6
2)py3Dmol7
今回はAnacondaにデフォルトで存在しないライブラリを二つ用います。事前にインストールしてください。
・Mercuryによるcifファイル中の分子のmolファイル抽出
→Mercuryでcifファイルを開きます。
→重ね合わせを行いたい分子のみ表示させます。
→File-Save as...からMOL file形式で保存します。
実行プログラム
準備ができたらANACONDA.NAVIGATORからJupyterLabを起動し次のプログラムを動かします。
動かす際にはinput sectionの情報を実行条件に合わせて適切に変更してください。
ここでは現在のディレクトリにある1515581.mol、2001546.molの二分子を分子中の'OC(O)C'の部分(COOH、カルボキシ基)が重なるように画像を表示します。
本プログラムを作成するにあたって注釈のサイトを参考にさせていただきました。7
from rdkit import Chem
from rdkit.Chem import AllChem
from rdkit.Chem.Draw import IPythonConsole
import py3Dmol
import copy
##input section
#input files
mol0 = Chem.MolFromMolFile("./1515581.mol", removeHs=False)
mol1 = Chem.MolFromMolFile("./2001546.mol", removeHs=False)
#重ね合わせ箇所smiles表記
template_smile = 'OC(O)C'
##calculation section
print(Chem.MolToSmiles(mol0))
print(Chem.MolToSmiles(mol1))
mols = [mol0, mol1]
template = Chem.MolFromSmiles(template_smile)
align_mols = [copy.deepcopy(m) for m in mols]
view = py3Dmol.view(width=400, height=300)
prbMatch = align_mols[0].GetSubstructMatch(template)
refMatch = align_mols[1].GetSubstructMatch(template)
AllChem.AlignMol(prbMol=align_mols[0], refMol=align_mols[1],atomMap=list(zip(prbMatch, refMatch)))
view.addModel(Chem.MolToMolBlock(align_mols[0]), 'sdf')
view.addModel(Chem.MolToMolBlock(align_mols[1]), 'sdf')
view.setStyle({'stick': {}})
view.setBackgroundColor('0xeeeeee')
view.zoomTo()
view.show()
view.png()
実行が完了したら、
1)重ね合わせを行う分子のsmiles表記
2)view.show()で表示される分子の重ね合わせ画像
3)view.png()で表示される分子の重ね合わせ静止画像
がJupyterLab上に表示されます。view.show()で表示された画像はマウスで掴んでぐりぐり回したり拡大縮小できます。view.png()で表示された静止画像はドラックアンドドロップで保存ができます。なので画像の保存はview.show()で表示された画像をスクリーンショットするかview.png()で表示された静止画像のドラックアンドドロップで行ってください。
これで得られた画像は次のようになります。
分子左手前のカルボキシ基が重ねられていることがわかるかと思います。
重ね方を変えて、ベンゼン環が重なる形で表示したいなとなるかもしれません。その時は、input sectionのtemplate_smileを'OC(O)C'から'C1C([H])C([H])C([H])C([H])C1'に変更してください。そうしたら次のような画像が得られます。
今日のところはここまで。
-
COD ID:1515581
アスピリンの結晶構造の文献。
Varughese, Sunil, et al. Chemical Science 2.11 (2011): 2236-2242. ↩ -
COD ID:2001546
イブプロフェンの結晶構造の文献。
Freer, ANDREW A., et al. Acta Crystallographica Section C: Crystal Structure Communications 49.7 (1993): 1378-1380. ↩ -
Crystallography Open Database。
http://www.crystallography.net/cod/ ↩ -
Mercuryのダウンロード。
https://www.ccdc.cam.ac.uk/support-and-resources/downloads/ ↩ -
Anacondaのダウンロード。
https://www.anaconda.com/ ↩ -
Rdkitのcondaによるインストール。
https://anaconda.org/rdkit/rdkit
もしくはhttps://www.rdkit.org/docs_jp/Installation_jp.html ↩ -
py3Dmolのcondaによるインストール。
https://anaconda.org/conda-forge/py3dmol ↩ ↩2


