以前書いた下記の記事では、ひとまずAIアプリを作るための一連の流れを覚えることが主目的だったので、画像判別精度についてはあまり求めていなかったです。
ただ、やはり機械学習エンジニアを名乗る以上は、精度を追求せねば、、ということで、今回はその精度アップの話をしたいと思います。
参考にしたページとやったこと
ここでは、脳のMRI画像のデータセットの「Discussion」ページで人気の高かった下記ページを参考にしました。
精度アップのためにやったことは主に下記2つです。
- 画像の余計な部分を削除
- 画像の水増し
※モデルの構造は前回の記事から変えていません。
それでは順番に説明していきます。
1.画像の余計な部分を削除
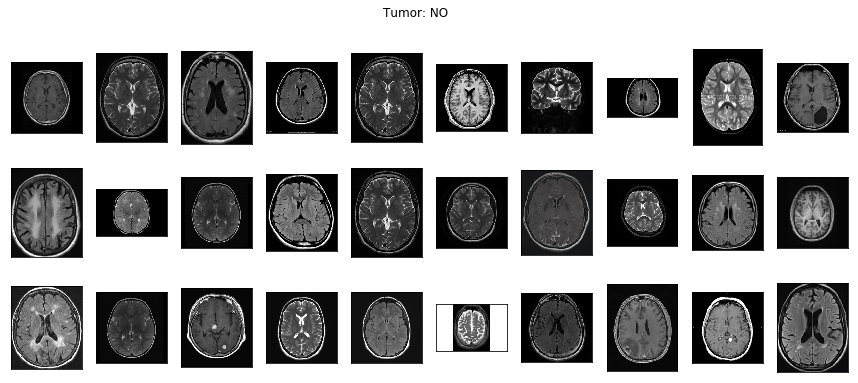
下記画像を見れば分かるように、ここで提供されているデータセットの画像はサイズがバラバラで、しかも脳の周りの黒い空白スペースの面積もバラバラです。
モデルに変な学習をさせないためにも、画像ごとの差はできるだけ小さい方が望ましいので脳の周りの余計な部分は削除します。
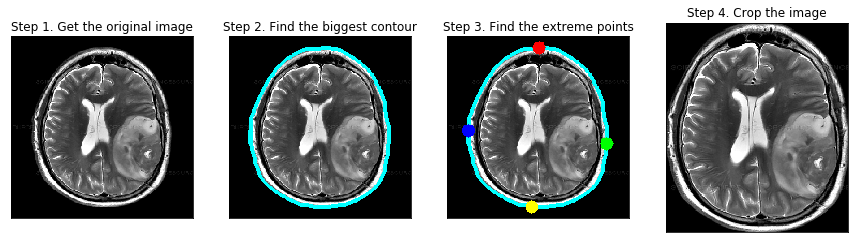
削除の流れとしては、下記にあるように4ステップで進めていきます(Step1は読み込むだけなので処理としては3ステップ)。
やってることとしては、まずはSTEP2で画像の輪郭を取得します。
この時、RGBだと輪郭が取り辛いので一旦グレースケールに変換しています。
ここでは0〜255諧調のうちの45で区切ってますが、どこで白・黒を区切るかはさじ加減ですね。
STEP3では、その輪郭から一番外側のポイントを4つ取得しています。
最後のSTEP4では、その4つのポイントを通る四角形を作り、それ以外の部分を削除することで脳の画像だけが残るようにしています。
def crop_imgs(set_name, add_pixels_value=0):
"""
Finds the extreme points on the image and crops the rectangular out of them
"""
set_new = []
for img in set_name:
gray = cv2.cvtColor(img, cv2.COLOR_RGB2GRAY)
gray = cv2.GaussianBlur(gray, (5, 5), 0)
# threshold the image, then perform a series of erosions +
# dilations to remove any small regions of noise
thresh = cv2.threshold(gray, 45, 255, cv2.THRESH_BINARY)[1]
thresh = cv2.erode(thresh, None, iterations=2)
thresh = cv2.dilate(thresh, None, iterations=2)
# find contours in thresholded image, then grab the largest one
cnts = cv2.findContours(thresh.copy(), cv2.RETR_EXTERNAL, cv2.CHAIN_APPROX_SIMPLE)
cnts = imutils.grab_contours(cnts)
c = max(cnts, key=cv2.contourArea)
# find the extreme points
extLeft = tuple(c[c[:, :, 0].argmin()][0])
extRight = tuple(c[c[:, :, 0].argmax()][0])
extTop = tuple(c[c[:, :, 1].argmin()][0])
extBot = tuple(c[c[:, :, 1].argmax()][0])
ADD_PIXELS = add_pixels_value
new_img = img[extTop[1]-ADD_PIXELS:extBot[1]+ADD_PIXELS, extLeft[0]-ADD_PIXELS:extRight[0]+ADD_PIXELS].copy()
set_new.append(new_img)
return np.array(set_new)
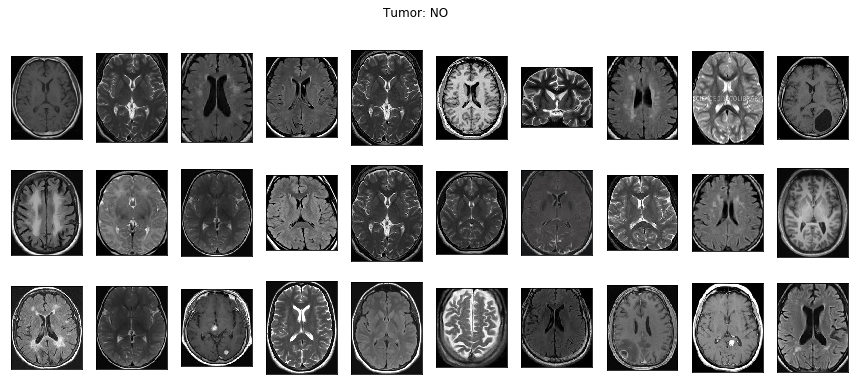
上記で不要な部分を削除すると、下記のように画像に統一感が出てきます。
2.画像の水増し
今回のデータセットの画像は100〜200枚しかなかったので、データを水増しします。
水増しすると悪いことをやっているようですが、機械学習の世界では良い行いとされています。
水増しの手法は色々あって、一般的には下記のようなことが行われます。
- 画像を回転
- 画像を上下左右にずらす(はみ出しても良し)
- 左右上下反転
- 明るさを変える
train_datagen = ImageDataGenerator(
rotation_range=15,
width_shift_range=0.1,
height_shift_range=0.1,
shear_range=0.1,
brightness_range=[0.5, 1.5],
horizontal_flip=True,
vertical_flip=True,
preprocessing_function=preprocess_input
)
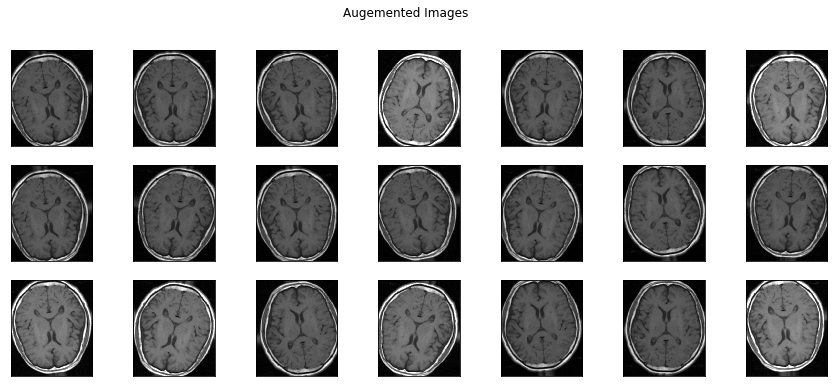
以下は水増しした画像です。微妙に違う画像が並んでいます。
水増しを行う時の注意点としては、水増しをしすぎるとたくさんの似たような画像から学習をすることになってしまうので、過学習が起きやすいということです。
なので水増しはほどほどにしておきましょう。
その他、モデルの構造は大きくは変えていません。
それではこれらの画像を使ってモデルの精度を確認してみましょう。
精度アップした結果
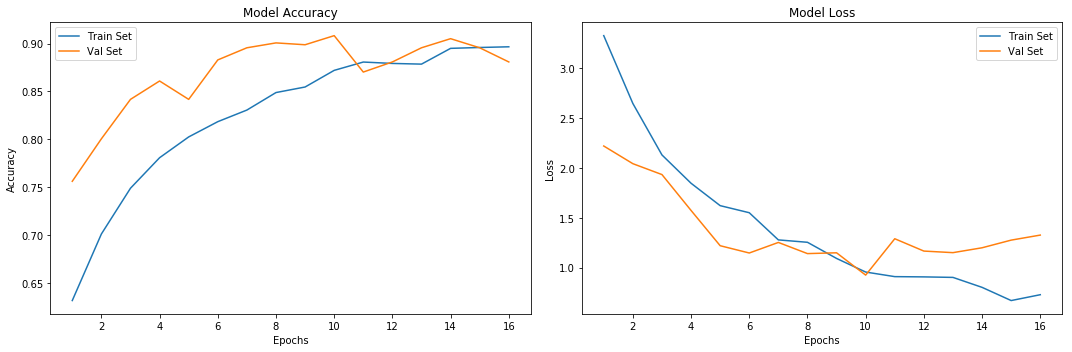
下記が今回のモデルの結果です。
エポック数が増えるにつれ精度は上昇し、最終的には90%近くにまで到達しているのが分かります。
お試しで作ったモデルとしては十分な精度だと思います。
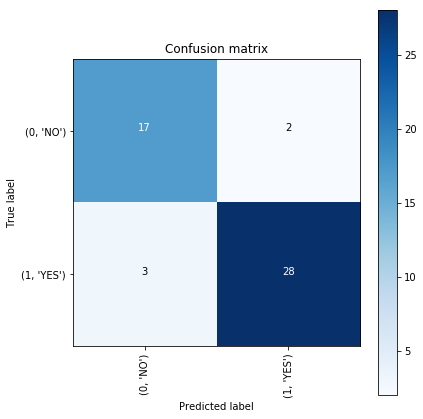
ただし、今回みたいな医療画像を判定する目的だと、間違った時に人の命に関わってしまうので「間違って無しと答えるよりも、間違って有りと診断する方がマシ」という考え方が一般的に取られます。
そういったことを判定するために作られるのが混同行列で、以下のように作ります。
# validate on val set
predictions = model.predict(X_val_prep)
predictions = [1 if x>0.5 else 0 for x in predictions]
accuracy = accuracy_score(y_val, predictions)
print('Val Accuracy = %.2f' % accuracy)
confusion_mtx = confusion_matrix(y_val, predictions)
cm = plot_confusion_matrix(confusion_mtx, classes = list(labels.items()), normalize=False)
まとめ
今回はページに記載されているコードをそのまま借用させてもらっただけですが、今後はオリジナルなアイデアも盛り込んで更なる精度アップをしていきたいと思っています。