【Python初心者】主婦が糖尿病診断の予測をしてみた
【目次】:
- 自己紹介
- 環境
- データ読み込み
- コンテンツ確認
- 考察
- データの可視化
- データの処理
- 学習・評価
- まとめ
1. 自己紹介
40代主婦。Pythonは初心者レベル。
Access(データベース)が好きで、SQLとVBAをちょっぴりかじった程度。
とはいえ、コーディングは殆どしておらず、ちょっぴりカスタマイズ出来る程度・・。
あるあるですが、育児と仕事の両立に悩み、お家でもお仕事出来るようになりたい!と切に願い一念発起。
以前から興味のあったデータ分析を学びたいと思い、Aidemyのデータ分析講座 (3ヶ月)を受講しました。
妊娠糖尿病患者と直接関わる機会があったので、糖尿病患者のデータ分析をしてみました。
2. 環境
環境詳細 :
OS: Windows 10
ブラウザ: Google Chrome
開発環境: Google Colaboratory
Python 3.10.12
Pandas 1.5.3
matplotlib 3.7.1
sklearn 1.2.2
Seaborn 0.12.2
必要なライブラリとバージョン情報:
from platform import python_version
import pandas as pd
import matplotlib
import sklearn
import matplotlib.pyplot as plt
import seaborn as sns
from sklearn.ensemble import RandomForestClassifier
from sklearn.model_selection import train_test_split
from sklearn.metrics import f1_score
from sklearn.metrics import f1_score,recall_score
# 警告が多く表示されるため以下ライブラリと設定により非表示にする
import warnings
warnings.simplefilter('ignore')
#version確認
print(python_version())
print(pd.__version__)
print(matplotlib.__version__)
print(sklearn.__version__)
print(sns.__version__)
3.10.12
1.5.3
3.7.1
1.2.2
0.12.2
3. データの読み込み
手順:
今回のデータセットはKaggleの下記サイトからダウンロード可能です。
https://www.kaggle.com/datasets/akshaydattatraykhare/diabetes-dataset
kaggleからデータセットをダウンロードし読み込みます。
データセットはGoogleドライブ上に保存しています。
Googleドライブ上のデータファイルを読み込むには、事前にdriveのインポートとマウントを行う必要があります。
from google.colab import drive
drive.mount('/content/drive')
Mounted at /content/drive
ドライブのマウントが完了したら、指定したパスのファイル(データ)を読み込みます。
df = pd.read_csv("/content/drive/MyDrive/Datasets/diabetes.csv")
4. コンテンツ確認
データ内容確認
データを読み込んだので、以下の流れでデータの中身を確認します。
- データの先頭10行を表示して、実際のデータの中身を確認する
- データフレームの行数、カラムを確認する
- 各カラムのデータ型を確認する
print("◆先頭10行表示: ")
print(df.head(10))
print()
print("◆行数、カラム数確認: ")
print(df.shape)
print()
print("◆データ型確認: ")
print(df.dtypes)
◆先頭10行表示:
Pregnancies Glucose BloodPressure SkinThickness Insulin BMI \
0 6 148 72 35 0 33.6
1 1 85 66 29 0 26.6
2 8 183 64 0 0 23.3
3 1 89 66 23 94 28.1
4 0 137 40 35 168 43.1
5 5 116 74 0 0 25.6
6 3 78 50 32 88 31.0
7 10 115 0 0 0 35.3
8 2 197 70 45 543 30.5
9 8 125 96 0 0 0.0
DiabetesPedigreeFunction Age Outcome
0 0.627 50 1
1 0.351 31 0
2 0.672 32 1
3 0.167 21 0
4 2.288 33 1
5 0.201 30 0
6 0.248 26 1
7 0.134 29 0
8 0.158 53 1
9 0.232 54 1
◆行数、カラム数確認:
(768, 9)
◆データ型確認:
Pregnancies int64
Glucose int64
BloodPressure int64
SkinThickness int64
Insulin int64
BMI float64
DiabetesPedigreeFunction float64
Age int64
Outcome int64
dtype: object
データフレームの構造は「768行、9列」ありますね。
768件のデータ、被験者がいたということになります。
糖尿病と判定された人、そうでない人の数を確認します。
※(Outcome=1)が陽性を意味します。
df["Outcome"].value_counts()
0 500
1 268
Name: Outcome, dtype: int64
全体で768人、そのうち500人が糖尿病でない、268人が糖尿病と判定されています。
カラム概要
各カラムの意味は次の通りです。
- Pregnancies : 妊娠回数
- Glucose : 血糖値
- BloodPressure : 血圧
- SkinThickness : 皮膚の厚さ
- Insulin : インスリン
- BMI : 肥満度
- DiabetesPedigreeFunction : 糖尿病血統機能
- Age : 年齢
- Outcome : 成果(フラグ)。(糖尿病は1, そうでなければ0と判定)
補足: DiabetesPedigreeFunction、直訳すると「糖尿病血統機能」。なんだか良く分からない言葉ですよね。
勝手なイメージですが、恐らくこんな感じかと思います。
血の繋がりのある家族・親戚で糖尿病発症の要因を遺伝的に持っているかどうか。
例えば、祖母が糖尿病だった、叔父が高血圧だった、姉が妊娠糖尿病だった等、 血族で何らかの生活習慣病や糖尿病発症の病歴があったとします。 血族の病歴から、遺伝的に「糖尿病になる可能性(要因)を「数値化」したもの、と思われます。
要約統計量
次に、データの要約統計量(合計、平均、標準偏差、最小値、最大値等)を確認します。
df.describe()
Pregnancies Glucose BloodPressure SkinThickness Insulin BMI DiabetesPedigreeFunction Age Outcome
count 768.000000 768.000000 768.000000 768.000000 768.000000 768.000000 768.000000 768.000000 768.000000
mean 3.845052 120.894531 69.105469 20.536458 79.799479 31.992578 0.471876 33.240885 0.348958
std 3.369578 31.972618 19.355807 15.952218 115.244002 7.884160 0.331329 11.760232 0.476951
min 0.000000 0.000000 0.000000 0.000000 0.000000 0.000000 0.078000 21.000000 0.000000
25% 1.000000 99.000000 62.000000 0.000000 0.000000 27.300000 0.243750 24.000000 0.000000
50% 3.000000 117.000000 72.000000 23.000000 30.500000 32.000000 0.372500 29.000000 0.000000
75% 6.000000 140.250000 80.000000 32.000000 127.250000 36.600000 0.626250 41.000000 1.000000
max 17.000000 199.000000 122.000000 99.000000 846.000000 67.100000 2.420000 81.000000 1.000000
5. 考察
要約統計量の数値から分かること
-
countの項目で全てのカラムがデータの行数である768を満たしている。(欠損値はない。)
-
minの項目でPregnancies、Glucose、BloodPressure、SkinThickness、Insulin、BMI の最小値が0となってる。
-
Insulin の最大値が平均値から大きく離れており、std(標準偏差::データのばらつき具合)の値が大きい。 データを可視化し外れ値を確認する必要がある。
-
0か1で判定するOutcomeの平均が0.5より小さい。目的変数に偏りがあるので可視化して確認したほうが良さそう。
補足情報:
糖尿病には1型糖尿病、2型糖尿病、妊娠糖尿病の3種類があるが、一般的に知られていて数が多いのが2型糖尿病。
肥満でBMI、血糖値(Glucose)が高い。1型糖尿病は特殊で、遺伝的に体内でインスリンが作られない為、若くてBMI値が低くても糖尿病になる。
もし1型糖尿病に該当するケースがある場合は、数は少ないが、外れ値として「インスリンが極端に低くて若い陽性者」が検出される可能性がある。
仮説
- Glucose、BloodPressure、SkinThickness、 Insulin、BMI の値が0というのは通常は考えられない。
- 上記特徴については、0の値が無効値である可能性が高い。
6. データの可視化
仮説の検証と可視化
仮説を検証する為、Glucose、BloodPressure、SkinThickness、 Insulin、 BMI で「データにどれくらいばらつきがあるか」、可視化したいと思います。
データの可視化
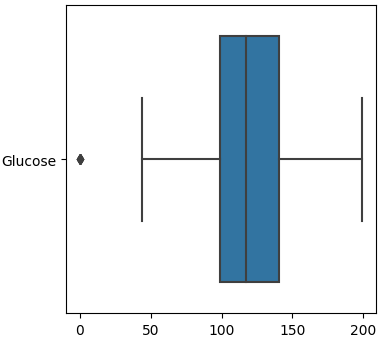
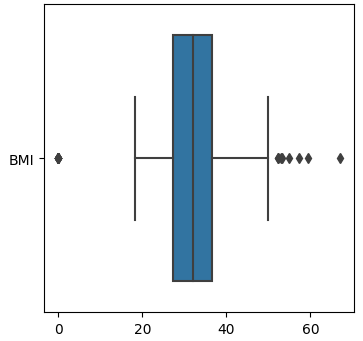
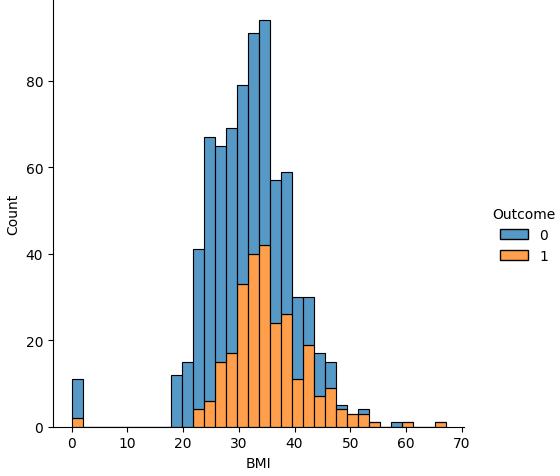
Glucose
Glucoseのデータのばらつきを箱ひげ図で確認します。plt.figure(figsize=(4,4))
sns.boxplot(data=df[["Glucose"]], orient="h")
plt.show()
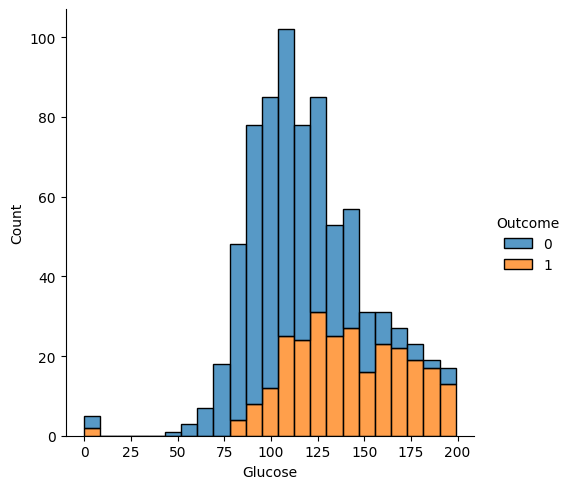
ヒストグラムで確認します。
sns.displot(data=df, x="Glucose", hue="Outcome", multiple="stack")
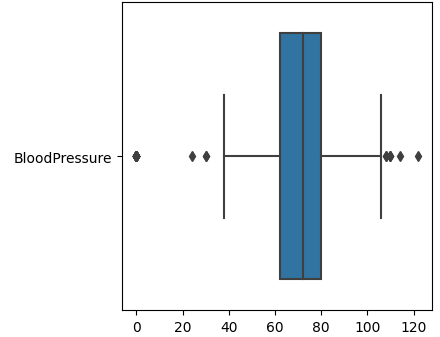
BloodPressure
BloodPressureの箱ひげ図を見てみます。
sns.boxplot(data=df[["BloodPressure"]], orient="h")
plt.show()
こちらも0値は明らかに外れ値で少なそうです。
BloodPressureのヒストグラムを見てみます
sns.displot(data=df, x="BloodPressure", hue="Outcome", multiple="stack")
BloodPressureの0値の個数は40個未満、全体の5%程度ですね。
BloodPressureの0値はやや多い、という印象です。
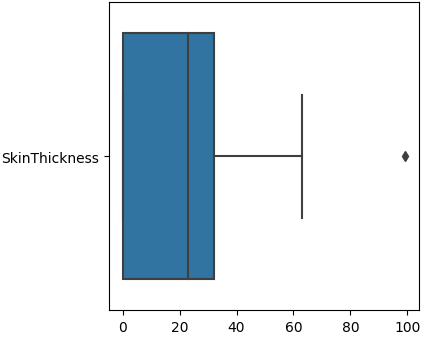
SkinThickness
SkinThicknessの箱ひげ図を見てみます。
plt.figure(figsize=(4,4))
sns.boxplot(data=df[["SkinThickness"]], orient="h")
plt.show()
100付近のデータは明らかな外れ値ですね。
0値が多そうです。
ヒストグラムで確認します。
sns.displot(data=df, x="SkinThickness", hue="Outcome", multiple="stack")
SkinThicknessの0値が230個近くあります。
また、60以上の外れ値は極端に少なくなっています。
Insulin
Insulinの箱ひげ図を見てみます。
plt.figure(figsize=(4,4))
sns.boxplot(data=df[["Insulin"]], orient="h")
plt.show()
ヒストグラムで確認します。
sns.displot(data=df, x="Insulin", hue="Outcome", multiple="stack")
0値が380ほどありますね。
0値が多すぎるのと、最小値と最大値の幅があり過ぎるのが気になります。
適切なデータが取得出来ていないのでは、という印象を持ちました。
検証結果:
「Glucose、 BloodPressure、 SkinThickness,、Insulin、 BMIの値が0というのは通常は考えられない」と仮説を立てました。
可視化した結果、0値が明らかに外れ値で、行を削除したほうが良さそうな特徴と、0値以外にも問題がある特徴が分かりました。
各特徴への対応方針:
-
BloodPressure:0値のデータは少なめ、5%程度。今回は削除せずに中央値で補填する
-
Glucose と BMI:0値が明らかに外れ値で個数も少ないので削除する。
-
SkinThickness: 0値の個数が多く、最大値、最小値の幅が大きい。その為、精度に与える影響大。相関関係も不明。今回は特徴として使わない。
-
Insullin:インスリン0値が有りうるのか不明。0値が全体の半分近くのウェイトを占めている。0値を補正するか、インスリン自体を特徴量として扱わないほうが良いかもしれない。今回は特徴量として使わない。
-
Pregnancies、DiabetesPedigreeFunction、Age については、補正は行わずに特徴量としてそのまま利用する。
以上から、下記流れでデータの前処理を行います。
- BloodPressureの0値を中央値で埋める
- GlucoseとBMIの0値を削除する
- SkinThickness と Insullin の列を削除する
7. データ前処理
データを前処理する
1. BloodPressureの0値を中央値で埋める
BloodPressureの中央値を計算します。
print(df['BloodPressure'].median())
72.0
BloodPresureが0のデータを中央値で埋めます。
df['BloodPressure'] = df['BloodPressure'].replace(0, df['BloodPressure'].median())
データの要約統計量を再確認します。
print(df.describe())
Pregnancies Glucose BloodPressure SkinThickness Insulin \
count 768.000000 768.000000 768.000000 768.000000 768.000000
mean 3.845052 120.894531 72.386719 20.536458 79.799479
std 3.369578 31.972618 12.096642 15.952218 115.244002
min 0.000000 0.000000 24.000000 0.000000 0.000000
25% 1.000000 99.000000 64.000000 0.000000 0.000000
50% 3.000000 117.000000 72.000000 23.000000 30.500000
75% 6.000000 140.250000 80.000000 32.000000 127.250000
max 17.000000 199.000000 122.000000 99.000000 846.000000
BMI DiabetesPedigreeFunction Age Outcome
count 768.000000 768.000000 768.000000 768.000000
mean 31.992578 0.471876 33.240885 0.348958
std 7.884160 0.331329 11.760232 0.476951
min 0.000000 0.078000 21.000000 0.000000
25% 27.300000 0.243750 24.000000 0.000000
50% 32.000000 0.372500 29.000000 0.000000
75% 36.600000 0.626250 41.000000 1.000000
max 67.100000 2.420000 81.000000 1.000000
BloodPresure の最小値が0値でなく、平均に中央値:72が表示されていることが確認できました
2. GlucoseとBMIの0値を削除する
最初にGlucoseが0の行を削除します。
コードとしては、dfをGlucose=0でないものに上書きするすることで、0の行を削除したものと一緒になります。
df=df[df['Glucose']!=0] # dfをGlucose=0でない行に上書きする
df.describe() # 要約統計量表示
Pregnancies Glucose BloodPressure SkinThickness Insulin BMI DiabetesPedigreeFunction Age Outcome
count 763.000000 763.000000 763.000000 763.000000 763.000000 763.000000 763.000000 763.000000 763.000000
mean 3.851900 121.686763 72.418087 20.477064 80.292267 31.986763 0.472477 33.271298 0.348624
std 3.374729 30.535641 12.098656 15.973171 115.457448 7.893221 0.331963 11.772161 0.476847
min 0.000000 44.000000 24.000000 0.000000 0.000000 0.000000 0.078000 21.000000 0.000000
25% 1.000000 99.000000 64.000000 0.000000 0.000000 27.300000 0.243500 24.000000 0.000000
50% 3.000000 117.000000 72.000000 23.000000 36.000000 32.000000 0.374000 29.000000 0.000000
75% 6.000000 141.000000 80.000000 32.000000 128.500000 36.550000 0.626500 41.000000 1.000000
max 17.000000 199.000000 122.000000 99.000000 846.000000 67.100000 2.420000 81.000000 1.000000
Glucoseの最小値が44.000000になり、0が無くなったことが確認できました。
次に、BMIが0の値を削除します。
df=df[df['BMI']!=0] # dfをBMI=0でない行に上書きする
df.describe() # 要約統計量表示
Pregnancies Glucose BloodPressure SkinThickness Insulin BMI DiabetesPedigreeFunction Age Outcome
count 752.000000 752.000000 752.000000 752.000000 752.000000 752.000000 752.000000 752.000000 752.000000
mean 3.851064 121.941489 72.385638 20.715426 81.348404 32.454654 0.473051 33.312500 0.351064
std 3.375189 30.601198 12.147132 15.930487 115.925034 6.928926 0.330108 11.709395 0.477621
min 0.000000 44.000000 24.000000 0.000000 0.000000 18.200000 0.078000 21.000000 0.000000
25% 1.000000 99.750000 64.000000 0.000000 0.000000 27.500000 0.244000 24.000000 0.000000
50% 3.000000 117.000000 72.000000 23.000000 39.000000 32.300000 0.377000 29.000000 0.000000
75% 6.000000 141.000000 80.000000 32.000000 130.000000 36.600000 0.627500 41.000000 1.000000
max 17.000000 199.000000 122.000000 99.000000 846.000000 67.100000 2.420000 81.000000 1.000000
BMIの最小値が18.200000になり、0が無くなったことが確認できました。
SkinThickness と Insullin の列を削除する
SkinThickness と Insullin の列を削除します。
df=df.drop(['SkinThickness', 'Insulin'], axis=1) # SkinThicknessとInsullin の列削除
df
Pregnancies Glucose BloodPressure BMI DiabetesPedigreeFunction Age Outcome
count 752.000000 752.000000 752.000000 752.000000 752.000000 752.000000 752.000000
mean 3.851064 121.941489 72.385638 32.454654 0.473051 33.312500 0.351064
std 3.375189 30.601198 12.147132 6.928926 0.330108 11.709395 0.477621
min 0.000000 44.000000 24.000000 18.200000 0.078000 21.000000 0.000000
25% 1.000000 99.750000 64.000000 27.500000 0.244000 24.000000 0.000000
50% 3.000000 117.000000 72.000000 32.300000 0.377000 29.000000 0.000000
75% 6.000000 141.000000 80.000000 36.600000 0.627500 41.000000 1.000000
max 17.000000 199.000000 122.000000 67.100000 2.420000 81.000000 1.000000
SkinThicknessとInsullinの列が削除されていることが確認できました。
データの前処理が終わったので、次は成形したデータを「教師データ」と「訓練データ」に分割し、モデルの学習を行います。
8. 学習・評価
モデルとスコア
今回はランダムフォレストで学習を行います。
また、「F値」と「再現率」でスコアを出してみたいと思います。
1 .F値 (score: 0.711864406779661)
バランスよく陽性、陰性の判定を行いたい場合は、F値でスコアを算出します。
# F値
import pandas as pd
import numpy as np
import matplotlib
import matplotlib.pyplot as plt
from sklearn.ensemble import RandomForestClassifier
from sklearn.model_selection import train_test_split
from sklearn.datasets import make_classification
from sklearn.metrics import f1_score
X = df.drop(["Outcome"], axis = 1) # 説明変数(糖尿病判定以外の特徴量使用)
y = df["Outcome"] # 目的変数(糖尿病判定フラグ)
# trainデータ、testデータの分割
train_X, test_X, train_y, test_y = train_test_split(X, y, test_size=0.2, random_state=42)
# 標準化の実施。※標準化はtrainデータ、testデータの分割後に行う
train_std = train_X.std(axis=0) #標準偏差値
train_mean = train_X.mean(axis=0) #平均値
std_train_X = (train_X-train_mean)/train_std #訓練データを標準化
std_test_X = (test_X-train_mean)/train_std # 検証データを標準化
# ランダムフォレスト
model = RandomForestClassifier(max_depth=10, n_estimators=20,random_state=0)
# モデルの学習
model.fit(std_train_X,train_y)
# test_Xに対するモデルの分類予測結果
y_pred = model.predict(std_test_X)
#print(y_pred)
#モデルのF値を計算する
score = f1_score(test_y, y_pred)
print("F値:" + str(score))
F値:0.711864406779661
2. 再現率 (score: 0.7)
今度は、再現率で評価を行います。
再現率は陽性判定を取りこぼしがないよう判定するので、糖尿病の可能性を見逃したくない場合に使用します。
# 再現率(recall)
import pandas as pd
import numpy as np
import matplotlib
import matplotlib.pyplot as plt
from sklearn.ensemble import RandomForestClassifier
from sklearn.model_selection import train_test_split
from sklearn.datasets import make_classification
from sklearn.metrics import f1_score,recall_score
X = df.drop(["Outcome"], axis = 1) # 説明変数(糖尿病判定以外の特徴量使用)
y = df["Outcome"] # 目的変数(糖尿病判定フラグ)
# trainデータ、testデータの分割
train_X, test_X, train_y, test_y = train_test_split(X, y, test_size=0.2, random_state=42)
# 標準化の実施
train_std = train_X.std(axis=0) #標準偏差値
train_mean = train_X.mean(axis=0) #平均値
std_train_X = (train_X-train_mean)/train_std #訓練データを標準化
std_test_X = (test_X-train_mean)/train_std # 検証データを標準化
# ランダムフォレスト
model = RandomForestClassifier(max_depth=10, n_estimators=20,random_state=0)
# モデルの学習
model.fit(std_train_X,train_y)
# test_Xに対するモデルの分類予測結果
y_pred = model.predict(std_test_X) #標準化した検証データで予測
#print(y_pred)
#モデルのスコア(再現率)を計算する
score = recall_score(test_y, y_pred)
print("再現率:" + str(score))
再現率:0.7
学習結果とまとめ
今回、F値と再現率で評価を行った結果、以下の結果となりました。
- F値 : score: 0.711864406779661
- 再現率 : score: 0.7
学習結果の精度はあまり高くありませんでしたが、課題もいくつか見えてきました。
SkinThickness とInsulin の列を安易に削除してしまったので、ヒートマップで各特徴の相関関係を可視化し、閾値を設けて外れ値を除去する手法も取り入れたいと思いました。
また、上記を行った上で、複数モデルを使ったランダムリサーチ、グリッドリサーチの両方でクロスバリデーションを行い、最適なハイパーパラメータを算出する流れを構築したいと思いました。
(偉そうに書いていますが、まだまだ理解と実力が伴っていません・・。精進あるのみです!)
今回のケースだと、糖尿病であるかないか、バランスよく判定したい場合はF値を使います。
糖尿病の可能性を見逃したくない、取りこぼしがないよう陽性判定をしたい場合は、再現率を使います。
上記を踏まえると、病院の診断等、陽性判定を見逃したく場合は再現率を使ったほうが良いかもしれません。
9. まとめ
Aidemyのデータ分析講座を終えて
今回、Aidemyで三カ月間、データ分析講座を受講し、(講師の方やネットの先人たちの知恵を拝借しつつ)データ分析の流れを掴めたことは、大変良い経験でした。 (正直、全然コードが書けなくて、いかに基礎が大事か思い知らされました。)専門用語が分からない、コーディングがうまくいかない等、試行錯誤で時間ばかり経過する辛さも経験しましたが、それでも分かった瞬間は、感慨もひとしおでした。
統計学の知識や数式に頭がフリーズすることもしばしばですが、改めて数学や統計学に興味を持つきっかけにもなりました。
(統計検定、G検定、e-資格など、いつかチャレンジしたいです!)
今後の課題:
-
アウトプット:
今後は学んだことをいかにアウトプットして、自身の強みに繋げていくかが課題だと思いました。 -
時間の使い方:
分からないところを延々と悩むよりも、疑問点を明確にして誰かに教えを乞うことが、レベルアップの近道!と思いました。 -
写経:
理解するという意味では(お手本となる)コードを写経(?)するのも大事だと痛感しました。
写経して理解してからコピペ、という流れが自分には合っていそうです。 -
チートシート:
理解してからのコピペ、のコピペの部分ですが、情報の取り纏めと迅速なアウトプットの為、必須だと思いました。
散らばった情報をどうやって集約し、必要な時に迅速にアウトップと出来るか。
迅速に、というのがミソですが・・・方法は模索中です。
課題はてんこ盛りですが、これからもプログラミングを楽しみつつ、実務に繋げていけるよう勉強を継続したいと思います。
今回のデータ分析の内容に更なるブラッシュアップをかけて、より汎用性のあるモデルを構築するのが、目下の目標です。)
最後までお読みいただき、有難うございました!