海外youtubeで、Pythonを使って、Covid-19(コロナ)の感染者数などのデータを使ってデータ分析をするレクチャー動画があったのでやってみました。
出典動画はこちら↓
Analyzing Coronavirus with Python (COVID-19) by NeuralNine on Youtube
対象
Pythonの初心者で、Pandasに慣れたいデータ分析をしてみたい方に特におすすめです。
レクチャーは英語ですが、とても平易かつ丁寧な解説なので是非見てみて下さい。
本記事について
- Jupyter notebook上で日本語でコメントを入れながら、当該動画の演習を行いました。実際に動画を見ながら演習を行うのがおすすめですが、英語が苦手な方やデータ分析の流れを把握したい方が、ざっとこの記事を読むだけでもイメージをつかめるように書いています。
- リアルデータ(※出典は後述)を利用していますが、データ分析プロセスとしては特にシャープな分析結果を目的としているものではなく、(Pandasライブラリ等の)演習に重きを置いたものになります。
- 2020/5/1までのデータを利用しています。(※レクチャー動画は、撮影時の3/22までのデータ)
演習
Analyzing Coronavirus with Python (COVID-19) by NeuralNine on Youtube
データセットは以下のサイトからダウンロード
HDX(HUMANITARIAN DATA EXCHANGE)
リンク先のデータセットの以下のものを使用
- time_series_covid19_confirmed_global.csv
- time_series_covid19_deaths_global.csv
- time_series_covid19_recovered_global.csv
【感染(確認)者,死者数,回復者数】 のデータです。
データの名前が長いので、以下のように変更します。
- time_series_covid19_confirmed_global.csv → covid_confirmed.csv
- time_series_covid19_deaths_global.csv → covid_deaths.csv
- time_series_covid19_recovered_global.csv → covid_recovered.csv
ライブラリをインポート
import pandas as pd
import matplotlib.pyplot as plt
%matplotlib inline
データを読み込みます
confirmed = pd.read_csv("covid19_confirmed.csv")
deaths = pd.read_csv("covid19_deaths.csv")
recovered = pd.read_csv("covid19_recovered.csv")
試しに感染者のデータを表示させます(※ 元の動画では、撮影時時点の3/22のデータですが、以下は5/1まで)
confirmed.head()
| Province/State | Country/Region | Lat | Long | 1/22/20 | 1/23/20 | 1/24/20 | 1/25/20 | 1/26/20 | 1/27/20 | ... | 4/22/20 | 4/23/20 | 4/24/20 | 4/25/20 | 4/26/20 | 4/27/20 | 4/28/20 | 4/29/20 | 4/30/20 | 5/1/20 | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | NaN | Afghanistan | 33.0000 | 65.0000 | 0 | 0 | 0 | 0 | 0 | 0 | ... | 1176 | 1279 | 1351 | 1463 | 1531 | 1703 | 1828 | 1939 | 2171 | 2335 |
| 1 | NaN | Albania | 41.1533 | 20.1683 | 0 | 0 | 0 | 0 | 0 | 0 | ... | 634 | 663 | 678 | 712 | 726 | 736 | 750 | 766 | 773 | 782 |
| 2 | NaN | Algeria | 28.0339 | 1.6596 | 0 | 0 | 0 | 0 | 0 | 0 | ... | 2910 | 3007 | 3127 | 3256 | 3382 | 3517 | 3649 | 3848 | 4006 | 4154 |
| 3 | NaN | Andorra | 42.5063 | 1.5218 | 0 | 0 | 0 | 0 | 0 | 0 | ... | 723 | 723 | 731 | 738 | 738 | 743 | 743 | 743 | 745 | 745 |
| 4 | NaN | Angola | -11.2027 | 17.8739 | 0 | 0 | 0 | 0 | 0 | 0 | ... | 25 | 25 | 25 | 25 | 26 | 27 | 27 | 27 | 27 | 30 |
5 rows × 105 columns
※ 緯度(Lat),経度(Long)
今回は、ProvinceとLat/Longはあまり必要でないので、列ごと消します
confirmed = confirmed.drop(['Province/State','Lat','Long'],axis=1)
deaths = deaths.drop(['Province/State','Lat','Long'],axis=1)
recovered = recovered.drop(['Province/State','Lat','Long'],axis=1)
このデータを、Country/Regionごとに集計してみましょう
confirmed = confirmed.groupby(confirmed["Country/Region"]).aggregate("sum")
deaths = deaths.groupby(deaths["Country/Region"]).aggregate("sum")
recovered = recovered.groupby(recovered["Country/Region"]).aggregate("sum")
confirmed.head()
| 1/22/20 | 1/23/20 | 1/24/20 | 1/25/20 | 1/26/20 | 1/27/20 | 1/28/20 | 1/29/20 | 1/30/20 | 1/31/20 | ... | 4/22/20 | 4/23/20 | 4/24/20 | 4/25/20 | 4/26/20 | 4/27/20 | 4/28/20 | 4/29/20 | 4/30/20 | 5/1/20 | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Country/Region | |||||||||||||||||||||
| Afghanistan | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | ... | 1176 | 1279 | 1351 | 1463 | 1531 | 1703 | 1828 | 1939 | 2171 | 2335 |
| Albania | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | ... | 634 | 663 | 678 | 712 | 726 | 736 | 750 | 766 | 773 | 782 |
| Algeria | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | ... | 2910 | 3007 | 3127 | 3256 | 3382 | 3517 | 3649 | 3848 | 4006 | 4154 |
| Andorra | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | ... | 723 | 723 | 731 | 738 | 738 | 743 | 743 | 743 | 745 | 745 |
| Angola | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | ... | 25 | 25 | 25 | 25 | 26 | 27 | 27 | 27 | 27 | 30 |
5 rows × 101 columns
そして、次に、日付が特徴量となっていますが、今回は国を特徴量にしたいため、データを転置(行列入れ替え)します。
confirmed = confirmed.T
deaths = deaths.T
recovered = recovered.T
confirmed.head()
| Country/Region | Afghanistan | Albania | Algeria | Andorra | Angola | Antigua and Barbuda | Argentina | Armenia | Australia | Austria | ... | United Kingdom | Uruguay | Uzbekistan | Venezuela | Vietnam | West Bank and Gaza | Western Sahara | Yemen | Zambia | Zimbabwe |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1/22/20 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | ... | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| 1/23/20 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | ... | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 |
| 1/24/20 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | ... | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 |
| 1/25/20 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | ... | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 |
| 1/26/20 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 4 | 0 | ... | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 |
5 rows × 187 columns
ここまでで、データの下準備は一旦整いました。計算に移っていきます。
まず、感染者数の増加数の推移をみていきましょう
ここで必要になってくるデータは当該日とその前日の感染者数の差です。
new_cases = confirmed.copy()
for day in range(1,len(confirmed)):
new_cases.iloc[day] = confirmed.iloc[day] - confirmed.iloc[day - 1]
直近の10日分のデータを見ます
new_cases.tail(10)
| Country/Region | Afghanistan | Albania | Algeria | Andorra | Angola | Antigua and Barbuda | Argentina | Armenia | Australia | Austria | ... | United Kingdom | Uruguay | Uzbekistan | Venezuela | Vietnam | West Bank and Gaza | Western Sahara | Yemen | Zambia | Zimbabwe |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 4/22/20 | 84 | 25 | 99 | 6 | 1 | 1 | 113 | 72 | 7 | 52 | ... | 4466 | 8 | 38 | 3 | 0 | 8 | 0 | 0 | 4 | 0 |
| 4/23/20 | 103 | 29 | 97 | 0 | 0 | 0 | 291 | 50 | 10 | 77 | ... | 4608 | 14 | 42 | 23 | 0 | 6 | 0 | 0 | 2 | 0 |
| 4/24/20 | 72 | 15 | 120 | 8 | 0 | 0 | 172 | 73 | 15 | 69 | ... | 5394 | 6 | 46 | 7 | 2 | 4 | 0 | 0 | 8 | 1 |
| 4/25/20 | 112 | 34 | 129 | 7 | 0 | 0 | 173 | 81 | 17 | 77 | ... | 4929 | 33 | 58 | 5 | 0 | -142 | 0 | 0 | 0 | 2 |
| 4/26/20 | 68 | 14 | 126 | 0 | 1 | 0 | 112 | 69 | 20 | 77 | ... | 4468 | 10 | 7 | 2 | 0 | 0 | 0 | 0 | 4 | 0 |
| 4/27/20 | 172 | 10 | 135 | 5 | 1 | 0 | 111 | 62 | 7 | 49 | ... | 4311 | 14 | 35 | 4 | 0 | 0 | 0 | 0 | 0 | 1 |
| 4/28/20 | 125 | 14 | 132 | 0 | 0 | 0 | 124 | 59 | 23 | 83 | ... | 4002 | 5 | 35 | 0 | 0 | 1 | 0 | 0 | 7 | 0 |
| 4/29/20 | 111 | 16 | 199 | 0 | 0 | 0 | 158 | 65 | 8 | 45 | ... | 4091 | 5 | 63 | 2 | 0 | 1 | 0 | 5 | 2 | 0 |
| 4/30/20 | 232 | 7 | 158 | 2 | 0 | 0 | 143 | 134 | 14 | 50 | ... | 6040 | 13 | 37 | 2 | 0 | 0 | 0 | 0 | 9 | 8 |
| 5/1/20 | 164 | 9 | 148 | 0 | 3 | 1 | 104 | 82 | 12 | 79 | ... | 6204 | 5 | 47 | 2 | 0 | 9 | 0 | 1 | 3 | 0 |
10 rows × 187 columns
感染者データと比べてみましょう
confirmed.tail(10)
| Country/Region | Afghanistan | Albania | Algeria | Andorra | Angola | Antigua and Barbuda | Argentina | Armenia | Australia | Austria | ... | United Kingdom | Uruguay | Uzbekistan | Venezuela | Vietnam | West Bank and Gaza | Western Sahara | Yemen | Zambia | Zimbabwe |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 4/22/20 | 1176 | 634 | 2910 | 723 | 25 | 24 | 3144 | 1473 | 6652 | 14925 | ... | 134638 | 543 | 1716 | 288 | 268 | 474 | 6 | 1 | 74 | 28 |
| 4/23/20 | 1279 | 663 | 3007 | 723 | 25 | 24 | 3435 | 1523 | 6662 | 15002 | ... | 139246 | 557 | 1758 | 311 | 268 | 480 | 6 | 1 | 76 | 28 |
| 4/24/20 | 1351 | 678 | 3127 | 731 | 25 | 24 | 3607 | 1596 | 6677 | 15071 | ... | 144640 | 563 | 1804 | 318 | 270 | 484 | 6 | 1 | 84 | 29 |
| 4/25/20 | 1463 | 712 | 3256 | 738 | 25 | 24 | 3780 | 1677 | 6694 | 15148 | ... | 149569 | 596 | 1862 | 323 | 270 | 342 | 6 | 1 | 84 | 31 |
| 4/26/20 | 1531 | 726 | 3382 | 738 | 26 | 24 | 3892 | 1746 | 6714 | 15225 | ... | 154037 | 606 | 1869 | 325 | 270 | 342 | 6 | 1 | 88 | 31 |
| 4/27/20 | 1703 | 736 | 3517 | 743 | 27 | 24 | 4003 | 1808 | 6721 | 15274 | ... | 158348 | 620 | 1904 | 329 | 270 | 342 | 6 | 1 | 88 | 32 |
| 4/28/20 | 1828 | 750 | 3649 | 743 | 27 | 24 | 4127 | 1867 | 6744 | 15357 | ... | 162350 | 625 | 1939 | 329 | 270 | 343 | 6 | 1 | 95 | 32 |
| 4/29/20 | 1939 | 766 | 3848 | 743 | 27 | 24 | 4285 | 1932 | 6752 | 15402 | ... | 166441 | 630 | 2002 | 331 | 270 | 344 | 6 | 6 | 97 | 32 |
| 4/30/20 | 2171 | 773 | 4006 | 745 | 27 | 24 | 4428 | 2066 | 6766 | 15452 | ... | 172481 | 643 | 2039 | 333 | 270 | 344 | 6 | 6 | 106 | 40 |
| 5/1/20 | 2335 | 782 | 4154 | 745 | 30 | 25 | 4532 | 2148 | 6778 | 15531 | ... | 178685 | 648 | 2086 | 335 | 270 | 353 | 6 | 7 | 109 | 40 |
10 rows × 187 columns
例えば、AfghanistanやAlgeriaやArgentinaやUnited Kingdomは、感染者数の絶対数も多いですが、新規感染者数も依然多いということが分かります。
new_casesでは、感染者数の日別の"増加数"を見ましたが、次に"増加率"を見ましょう。
(当該日の増加数 / 前日の感染者数) * 100 で増加率を出すことが出来ます
growth_rate = confirmed.copy()
for day in range(1,len(growth_rate)):
growth_rate.iloc[day] = ( new_cases.iloc[day] / confirmed.iloc[day-1] ) * 100
growth_rate.tail(10)
| Country/Region | Afghanistan | Albania | Algeria | Andorra | Angola | Antigua and Barbuda | Argentina | Armenia | Australia | Austria | ... | United Kingdom | Uruguay | Uzbekistan | Venezuela | Vietnam | West Bank and Gaza | Western Sahara | Yemen | Zambia | Zimbabwe |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 4/22/20 | 7.692308 | 4.105090 | 3.521878 | 0.836820 | 4.166667 | 4.347826 | 3.728143 | 5.139186 | 0.105342 | 0.349627 | ... | 3.430845 | 1.495327 | 2.264601 | 1.052632 | 0.000000 | 1.716738 | 0.0 | 0.000000 | 5.714286 | 0.000000 |
| 4/23/20 | 8.758503 | 4.574132 | 3.333333 | 0.000000 | 0.000000 | 0.000000 | 9.255725 | 3.394433 | 0.150331 | 0.515913 | ... | 3.422511 | 2.578269 | 2.447552 | 7.986111 | 0.000000 | 1.265823 | 0.0 | 0.000000 | 2.702703 | 0.000000 |
| 4/24/20 | 5.629398 | 2.262443 | 3.990688 | 1.106501 | 0.000000 | 0.000000 | 5.007278 | 4.793171 | 0.225158 | 0.459939 | ... | 3.873720 | 1.077199 | 2.616610 | 2.250804 | 0.746269 | 0.833333 | 0.0 | 0.000000 | 10.526316 | 3.571429 |
| 4/25/20 | 8.290155 | 5.014749 | 4.125360 | 0.957592 | 0.000000 | 0.000000 | 4.796230 | 5.075188 | 0.254605 | 0.510915 | ... | 3.407771 | 5.861456 | 3.215078 | 1.572327 | 0.000000 | -29.338843 | 0.0 | 0.000000 | 0.000000 | 6.896552 |
| 4/26/20 | 4.647984 | 1.966292 | 3.869779 | 0.000000 | 4.000000 | 0.000000 | 2.962963 | 4.114490 | 0.298775 | 0.508318 | ... | 2.987250 | 1.677852 | 0.375940 | 0.619195 | 0.000000 | 0.000000 | 0.0 | 0.000000 | 4.761905 | 0.000000 |
| 4/27/20 | 11.234487 | 1.377410 | 3.991721 | 0.677507 | 3.846154 | 0.000000 | 2.852004 | 3.550974 | 0.104260 | 0.321839 | ... | 2.798678 | 2.310231 | 1.872659 | 1.230769 | 0.000000 | 0.000000 | 0.0 | 0.000000 | 0.000000 | 3.225806 |
| 4/28/20 | 7.339988 | 1.902174 | 3.753199 | 0.000000 | 0.000000 | 0.000000 | 3.097677 | 3.263274 | 0.342211 | 0.543407 | ... | 2.527345 | 0.806452 | 1.838235 | 0.000000 | 0.000000 | 0.292398 | 0.0 | 0.000000 | 7.954545 | 0.000000 |
| 4/29/20 | 6.072210 | 2.133333 | 5.453549 | 0.000000 | 0.000000 | 0.000000 | 3.828447 | 3.481521 | 0.118624 | 0.293026 | ... | 2.519864 | 0.800000 | 3.249097 | 0.607903 | 0.000000 | 0.291545 | 0.0 | 500.000000 | 2.105263 | 0.000000 |
| 4/30/20 | 11.964930 | 0.913838 | 4.106029 | 0.269179 | 0.000000 | 0.000000 | 3.337223 | 6.935818 | 0.207346 | 0.324633 | ... | 3.628914 | 2.063492 | 1.848152 | 0.604230 | 0.000000 | 0.000000 | 0.0 | 0.000000 | 9.278351 | 25.000000 |
| 5/1/20 | 7.554123 | 1.164295 | 3.694458 | 0.000000 | 11.111111 | 4.166667 | 2.348690 | 3.969022 | 0.177357 | 0.511261 | ... | 3.596918 | 0.777605 | 2.305051 | 0.600601 | 0.000000 | 2.616279 | 0.0 | 16.666667 | 2.830189 | 0.000000 |
10 rows × 187 columns
さて、感染者数(confirmed)はいわゆる累計の数なので、一旦ここで、現在進行形の感染者数(Active)を出しましょう。
【感染者数(confirmed)】から【死者数(deaths)と回復者数(recovered)】を引くと、【現在進行形の感染者数(Active)】が計算できそうです。
active_cases = confirmed.copy()
for day in range(0,len(confirmed)):
active_cases.iloc[day] = confirmed.iloc[day] - deaths.iloc[day] - recovered.iloc[day]
そして、この現在進行形の感染者数active_casesのデータを利用して、再度、現在進行形の感染者数の増加率を調べましょう。
これを調べることによって、収束していそうかどうかが分かりそうです。
overall_growth_rate = confirmed.copy()
for day in range(0,len(confirmed)):
overall_growth_rate.iloc[day] = ((active_cases.iloc[day] - active_cases.iloc[day-1]) / active_cases.iloc[day-1]) * 100
overall_growth_rate.tail(10)
| Country/Region | Afghanistan | Albania | Algeria | Andorra | Angola | Antigua and Barbuda | Argentina | Armenia | Australia | Austria | ... | United Kingdom | Uruguay | Uzbekistan | Venezuela | Vietnam | West Bank and Gaza | Western Sahara | Yemen | Zambia | Zimbabwe |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 4/22/20 | 7.064018 | 5.462185 | 2.920284 | -5.276382 | 6.250000 | -15.384615 | 3.718200 | 6.250000 | -12.214551 | -9.498681 | ... | 3.270797 | -1.895735 | -4.258555 | -1.265823 | -13.461538 | 2.046036 | 0.000000 | 0.000000 | 12.500000 | -4.347826 |
| 4/23/20 | 9.072165 | 0.000000 | -4.524540 | -6.366048 | 0.000000 | 0.000000 | 10.896226 | 2.941176 | -6.836056 | -9.750567 | ... | 3.411790 | -7.729469 | -5.480540 | 12.179487 | -2.222222 | -3.759398 | -83.333333 | 0.000000 | 0.000000 | 0.000000 |
| 4/24/20 | 5.860113 | 2.390438 | 4.738956 | -1.699717 | 0.000000 | -9.090909 | 4.423650 | 0.119048 | -5.064935 | -4.199569 | ... | 3.743980 | -4.712042 | -1.260504 | 0.571429 | 13.636364 | 1.041667 | 0.000000 | -100.000000 | 22.222222 | 4.545455 |
| 4/25/20 | 9.642857 | 9.727626 | 4.141104 | 2.017291 | 0.000000 | 0.000000 | 4.480652 | 0.594530 | -15.321477 | -5.994755 | ... | 3.332975 | 16.483516 | -2.382979 | 2.840909 | -10.000000 | -36.082474 | 0.000000 | NaN | 0.000000 | 8.695652 |
| 4/26/20 | 3.745928 | 2.127660 | 6.701031 | 0.000000 | 5.882353 | 0.000000 | 1.091618 | 4.609929 | -11.954766 | -4.304504 | ... | 3.232666 | 1.886792 | -6.538797 | -1.657459 | 0.000000 | 3.629032 | 0.000000 | NaN | -2.272727 | 0.000000 |
| 4/27/20 | 11.930926 | -0.694444 | 5.383023 | -10.169492 | 5.555556 | 0.000000 | 2.815272 | 5.197740 | -3.669725 | -1.582674 | ... | 3.051680 | 1.388889 | -6.343284 | -0.561798 | 0.000000 | 0.000000 | 0.000000 | NaN | 0.000000 | -8.000000 |
| 4/28/20 | 8.134642 | 1.048951 | 2.226588 | -4.402516 | 0.000000 | 0.000000 | 3.450863 | 4.296455 | -5.714286 | -6.559458 | ... | 2.318102 | -1.369863 | -6.474104 | 0.000000 | 6.666667 | 5.058366 | 0.000000 | NaN | 16.279070 | 0.000000 |
| 4/29/20 | 5.512322 | -2.768166 | 9.032671 | -8.552632 | -5.263158 | 0.000000 | 4.387237 | 3.192585 | -4.444444 | -7.472826 | ... | 2.386758 | -6.018519 | -4.472843 | 1.129944 | 0.000000 | 0.370370 | 0.000000 | inf | -20.000000 | 0.000000 |
| 4/30/20 | 13.521819 | -3.202847 | 4.406580 | -15.467626 | 0.000000 | 0.000000 | 2.605071 | 10.279441 | -1.585624 | -4.013705 | ... | 3.845988 | 2.955665 | 0.000000 | -2.234637 | 6.250000 | -1.845018 | 0.000000 | -40.000000 | 20.000000 | 34.782609 |
| 5/1/20 | 5.955604 | -3.308824 | 5.796286 | -0.425532 | -5.555556 | -30.000000 | 2.064997 | 2.986425 | -2.255639 | -6.578276 | ... | 3.750518 | -6.220096 | -3.567447 | 1.142857 | 0.000000 | 3.383459 | 0.000000 | 33.333333 | -33.333333 | 0.000000 |
10 rows × 187 columns
直近10日(2020/04/22-05/01)の現在進行形の感染者数の増加率 (中国・イタリア・アメリカ・日本)
※ ちょこっと、ここの部分にオリジナルの加えています。
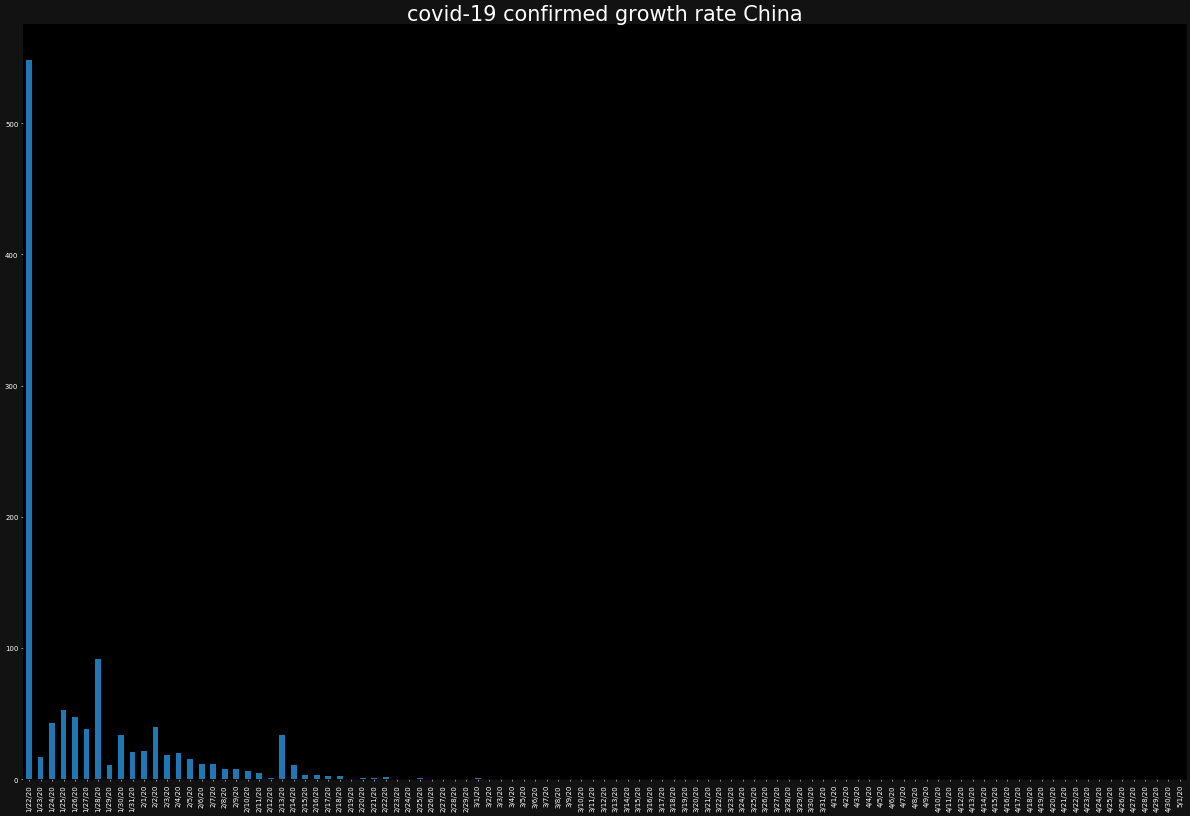
まず、コロナ発症国と考えられている中国は、最近収束しているみたいなので、中国の直近10日のデータを見てみましょう
overall_growth_rate['China'].tail(10)
4/22/20 -3.314528
4/23/20 -7.731583
4/24/20 -8.774704
4/25/20 -4.852686
4/26/20 -9.107468
4/27/20 -9.118236
4/28/20 -2.866593
4/29/20 -5.448354
4/30/20 -4.441777
5/1/20 -5.904523
Name: China, dtype: float64
増加率マイナス、つまり現在進行形の感染者数が減り続けている → 収束に向かっていることが分かります。
次に、イタリアはどうでしょうか。
overall_growth_rate['Italy'].tail(10)
4/22/20 -0.009284
4/23/20 -0.790165
4/24/20 -0.300427
4/25/20 -0.638336
4/26/20 0.241859
4/27/20 -0.273319
4/28/20 -0.574599
4/29/20 -0.520888
4/30/20 -2.967790
5/1/20 -0.598714
Name: Italy, dtype: float64
1%未満の減少率なので微減ですが、増えてはいません。
overall_growth_rate['US'].tail(10)
4/22/20 3.470050
4/23/20 3.307839
4/24/20 2.102556
4/25/20 3.874078
4/26/20 2.536775
4/27/20 2.064644
4/28/20 2.166569
4/29/20 2.377575
4/30/20 -0.668941
5/1/20 2.583283
Name: US, dtype: float64
アメリカは、まだ数%ずつ増加しています。
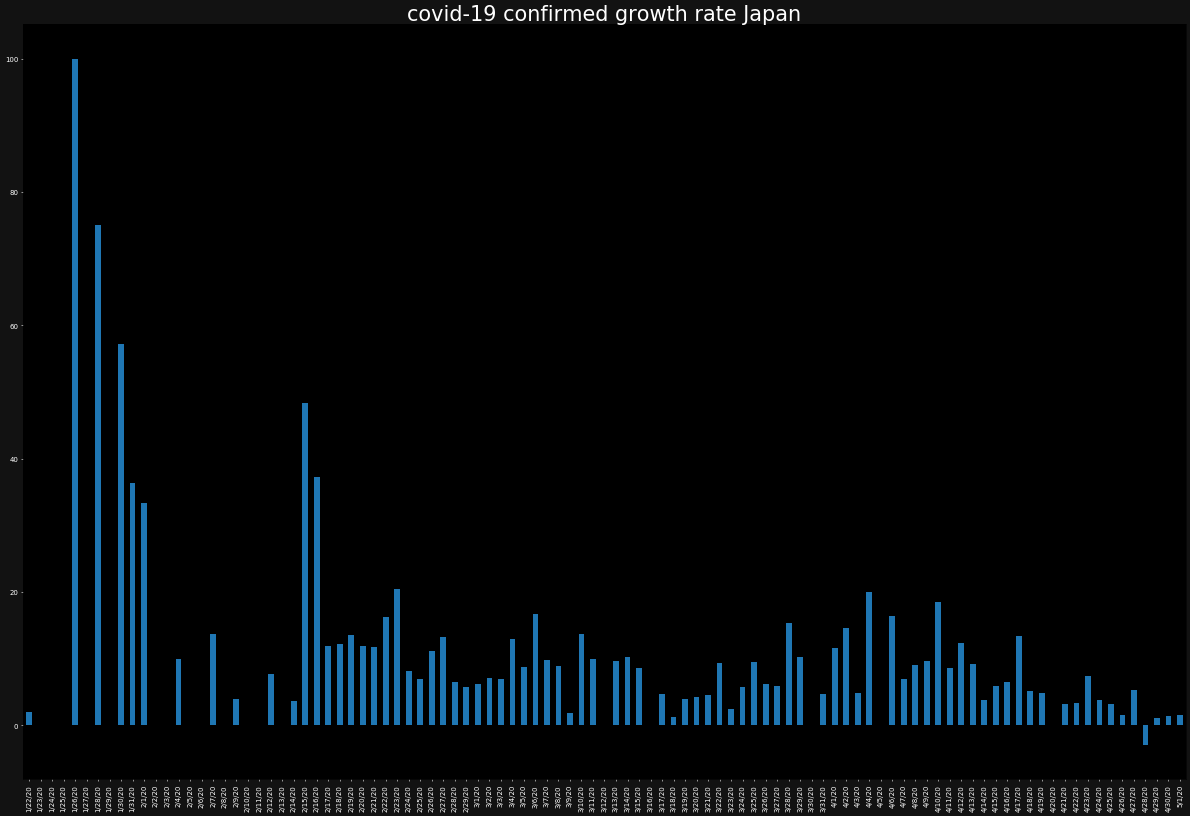
overall_growth_rate['Japan'].tail(10)
4/22/20 2.512198
4/23/20 6.794937
4/24/20 3.868765
4/25/20 2.382691
4/26/20 0.401248
4/27/20 5.408526
4/28/20 -3.589182
4/29/20 -2.875120
4/30/20 0.755803
5/1/20 -2.884444
Name: Japan, dtype: float64
日本も、ちょっとずつ直近で減ってきた感じはありますが、増加傾向にあります。
日本は、せっかくなので、直近10日の増加率の平均を見てみましょう。
overall_growth_rate['Japan'].tail(10).mean()
1.277542288600591
1%程度ですが、増加傾向にある感じです。まぁ、日本はまだ感染者数も少ないので、比較的抑えられているといえるかもしれません。
ここから可視化(visualization)を交えていきます。
最初に死亡率を見ていきましょう。死亡率は、地域ごとのコロナの深刻さを表す指標となるので重要です。
まず、死亡率のデータフレームは、先ほどの手順とほぼ同様です。死亡率は、死亡者/感染者 で表されます。
death_rate = confirmed.copy()
for day in range(0,len(confirmed)):
death_rate.iloc[day] = (deaths.iloc[day] / confirmed.iloc[day]) * 100
次に、必要な必要なベッド台数(逆に、どれだけ不足が生じそうか)を算出します。
感染者のうち病院が必要な人の割合である入院率"hospitalization"を用います。これは、正しい数値は、分からないので、一旦仮の数値(ここでは、0.05)を用います。好きな数字に変えても大丈夫です。このレクチャーでは、分析・計算手法にフォーカスするため、一旦正確さは置いておきます。
※ちなみに、入院率は、コロナ陽性かつ入院が必要な人のことで、残りの95%は陽性であっても、入院(ベッド)は必要ではないとみなしています。
hospitalization_rate_estimate = 0.05
hospitalization_needed = confirmed.copy()
for day in range(0,len(confirmed)):
hospitalization_needed.iloc[day] = active_cases.iloc[day] * hospitalization_rate_estimate
hospitalization_needed.tail()
| Country/Region | Afghanistan | Albania | Algeria | Andorra | Angola | Antigua and Barbuda | Argentina | Armenia | Australia | Austria | ... | United Kingdom | Uruguay | Uzbekistan | Venezuela | Vietnam | West Bank and Gaza | Western Sahara | Yemen | Zambia | Zimbabwe |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 4/27/20 | 71.30 | 14.30 | 76.35 | 15.90 | 0.95 | 0.50 | 133.30 | 46.55 | 52.50 | 118.15 | ... | 6654.15 | 10.95 | 50.20 | 8.85 | 2.25 | 12.85 | 0.05 | 0.00 | 2.15 | 1.15 |
| 4/28/20 | 77.10 | 14.45 | 78.05 | 15.20 | 0.95 | 0.50 | 137.90 | 48.55 | 49.50 | 110.40 | ... | 6808.40 | 10.80 | 46.95 | 8.85 | 2.40 | 13.50 | 0.05 | 0.00 | 2.50 | 1.15 |
| 4/29/20 | 81.35 | 14.05 | 85.10 | 13.90 | 0.90 | 0.50 | 143.95 | 50.10 | 47.30 | 102.15 | ... | 6970.90 | 10.15 | 44.85 | 8.95 | 2.40 | 13.55 | 0.05 | 0.25 | 2.00 | 1.15 |
| 4/30/20 | 92.35 | 13.60 | 88.85 | 11.75 | 0.90 | 0.50 | 147.70 | 55.25 | 46.55 | 98.05 | ... | 7239.00 | 10.45 | 44.85 | 8.75 | 2.55 | 13.30 | 0.05 | 0.15 | 2.40 | 1.55 |
| 5/1/20 | 97.85 | 13.15 | 94.00 | 11.70 | 0.85 | 0.35 | 150.75 | 56.90 | 45.50 | 91.60 | ... | 7510.50 | 9.80 | 43.25 | 8.85 | 2.55 | 13.75 | 0.05 | 0.20 | 1.60 | 1.55 |
5 rows × 187 columns
一旦、ここで国をひとつ取り出しても、どれだけ深刻かわかりづらいため、一旦直近5日の平均を見ます。
hospitalization_needed.tail().mean().mean()
532.5691978609626
直近5日のすべての国の必要ベッド数の平均です。ばらつきも当然あるため、あまり理想的な参考値ではないですが、一旦これを参考とします。
イタリアの直近5日の平均を見てみましょう。
hospitalization_needed['Italy'].tail().mean()
5181.6900000000005
つまり、ざっと世界中の平均の10倍という数値が出ました。かなり深刻です。
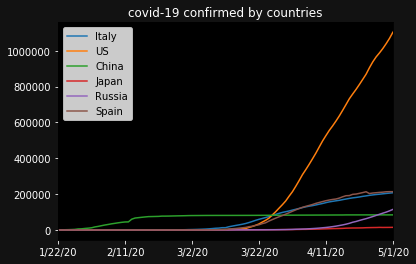
可視化します。ただ、国が多すぎるので、今回は、いくつか任意の国を選びましょう。ここでは、イタリア、アメリカ、中国、日本、ロシア スペインを選びます。
countries = ['Italy','US',"China","Japan","Russia","Spain"]
ax = plt.subplot()
ax.set_facecolor("black")
ax.figure.set_facecolor("#121212")
ax.tick_params(axis="x",colors="white")
ax.tick_params(axis="y",colors="white")
ax.set_title("covid-19 confirmed by countries",color="white")
for country in countries:
confirmed[country].plot(label=country)
plt.legend(loc="upper left")
plt.show()
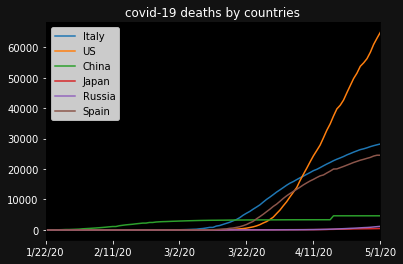
USが3月末ぐらいから著しく感染者が増えていますね。
死者数を見ましょう。
グラフの形はあまり変わらず、感染者数に伴っているような感じです。
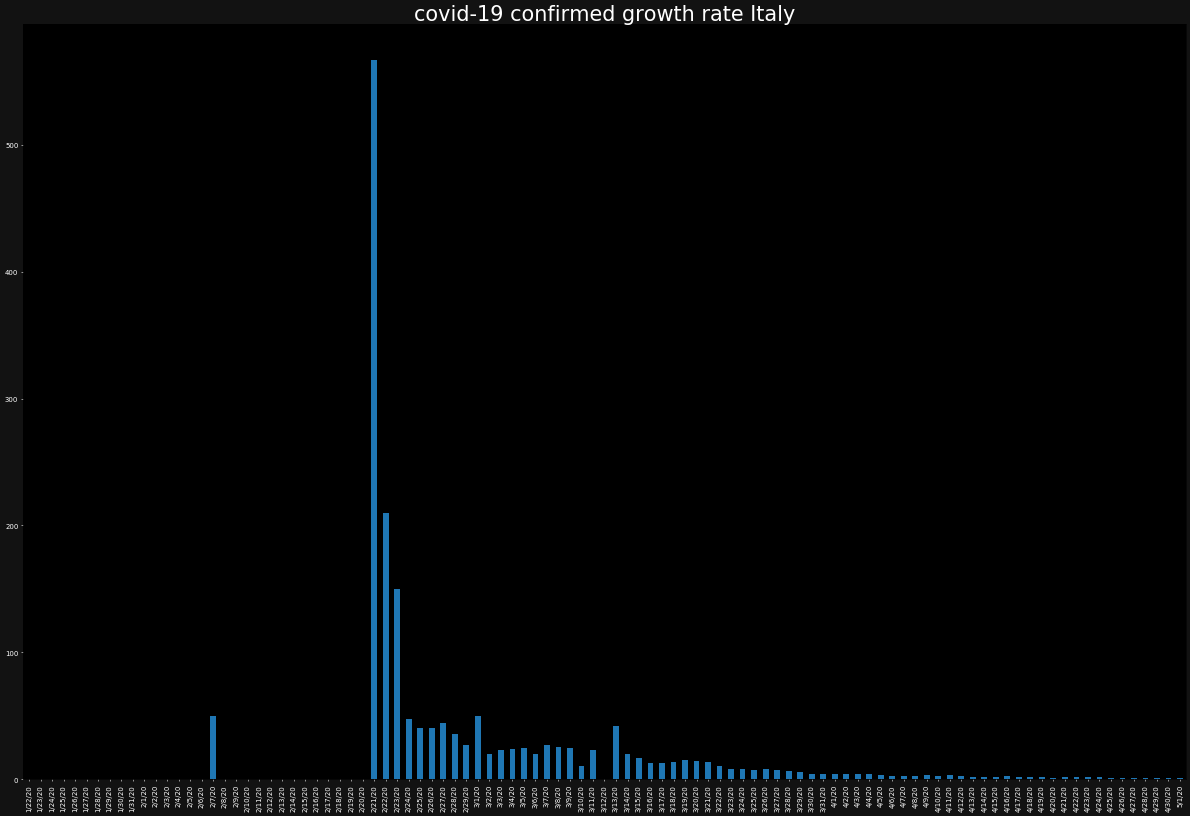
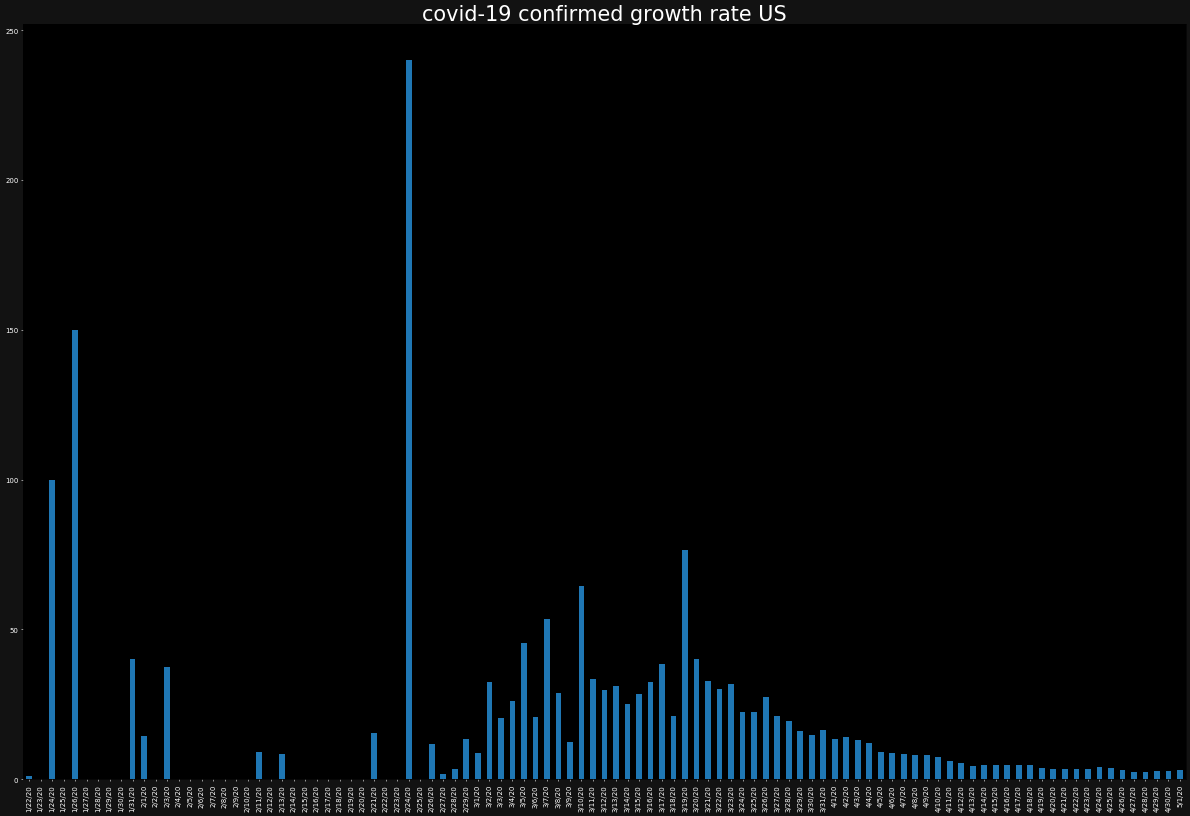
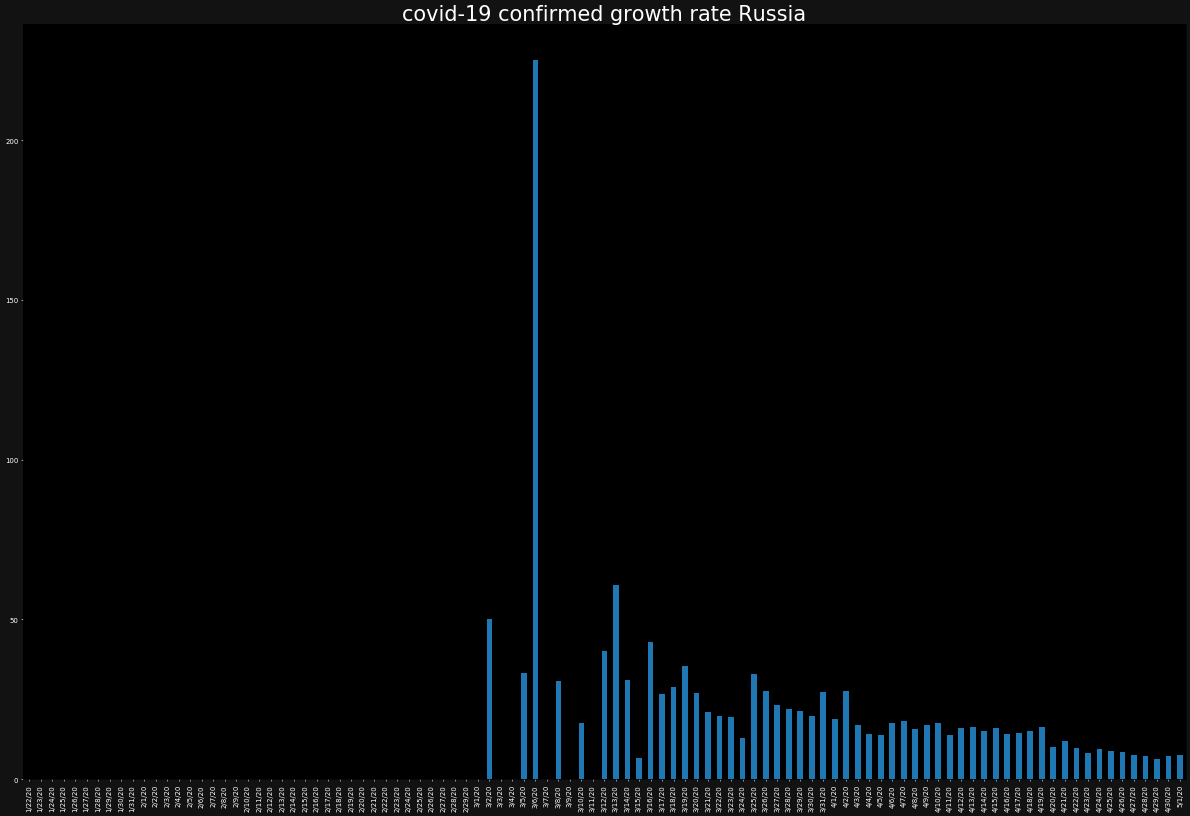
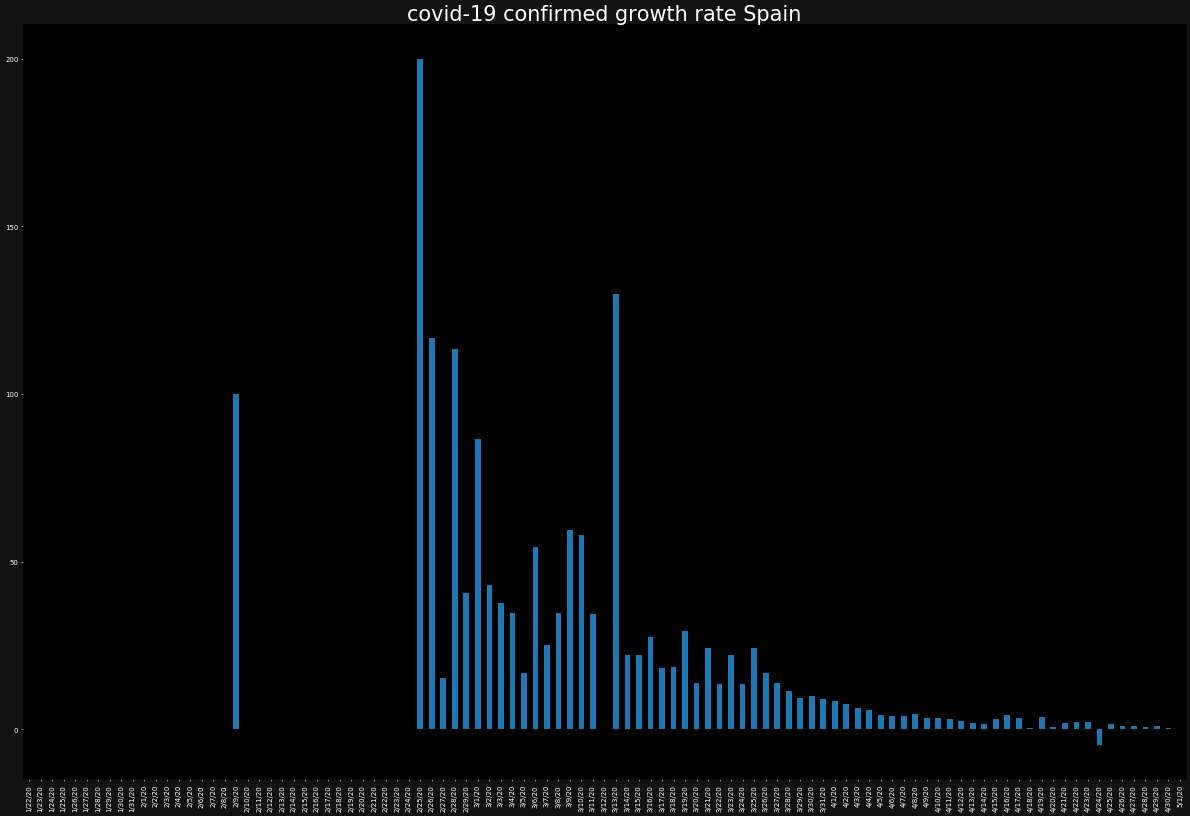
次に、感染者の増加率をプロットしてみましょう。
ただし、今度は、バーチャートでプロットします。また、バーチャートではあまりひとつの図にグラフが重なりすぎると、可視性が低くなるため別々に表示します。
for country in countries:
ax = plt.subplot()
ax.set_facecolor("black")
ax.figure.set_facecolor("#121212")
ax.tick_params(axis="x",colors="white")
ax.tick_params(axis="y",colors="white")
ax.set_title(f"covid-19 confirmed growth rate {country}",color="white")
growth_rate[country].plot.bar()
plt.show()
同じ要領で死者数の感染数、死亡率(※上記は、"感染者の増加率"でこちらは"死亡率"です)も見ましょう。
ax = plt.subplot()
ax.set_facecolor("black")
ax.figure.set_facecolor("#121212")
ax.tick_params(axis="x",colors="white")
ax.tick_params(axis="y",colors="white")
ax.set_title("covid-19 deaths by countries",color="white")
for country in countries:
deaths[country].plot(label=country)
plt.legend(loc="upper left")
plt.show()
for country in countries:
ax = plt.subplot()
ax.set_facecolor("black")
ax.figure.set_facecolor("#121212")
ax.tick_params(axis="x",colors="white")
ax.tick_params(axis="y",colors="white")
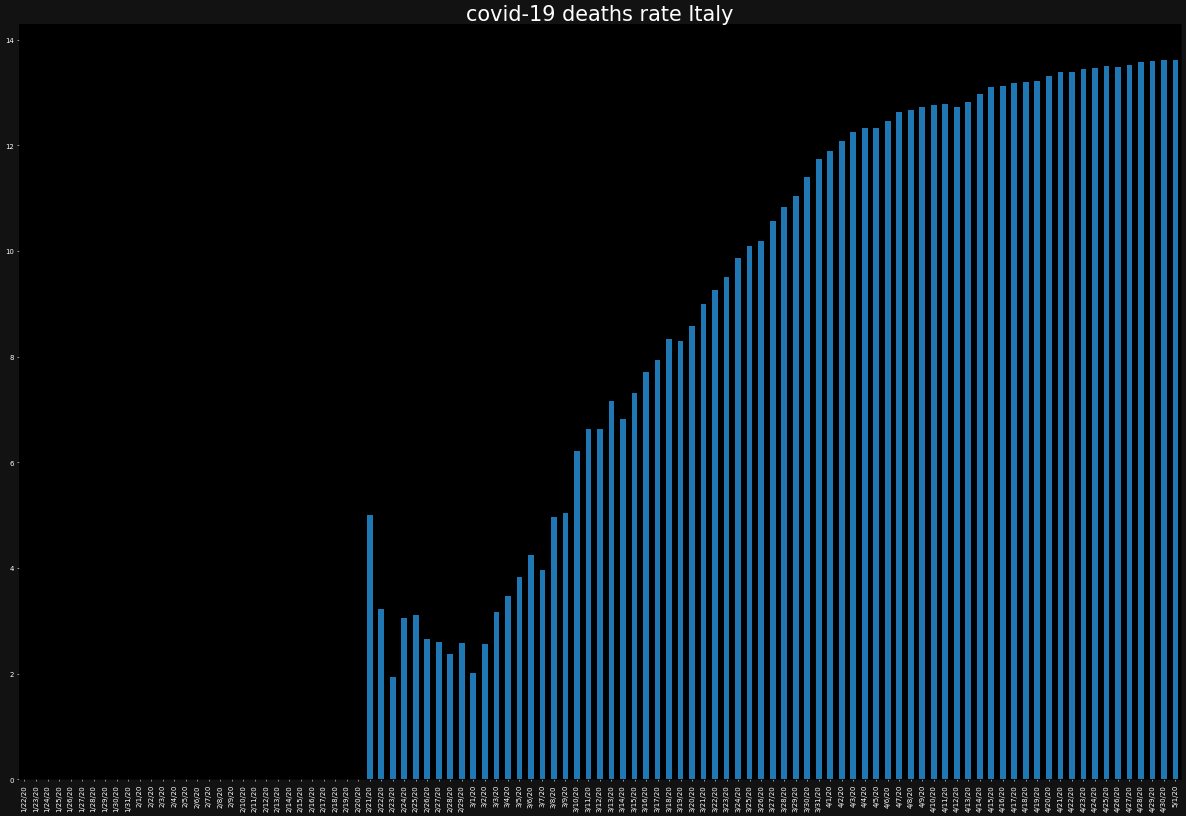
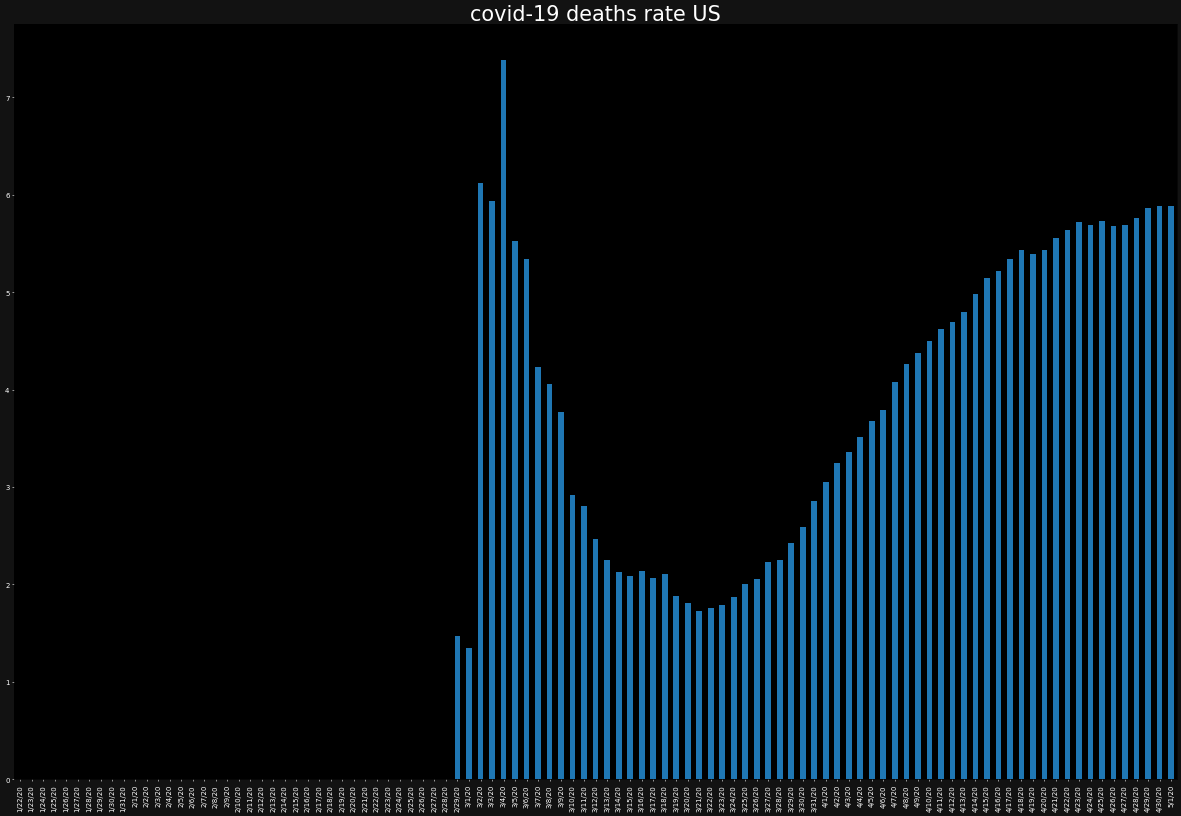
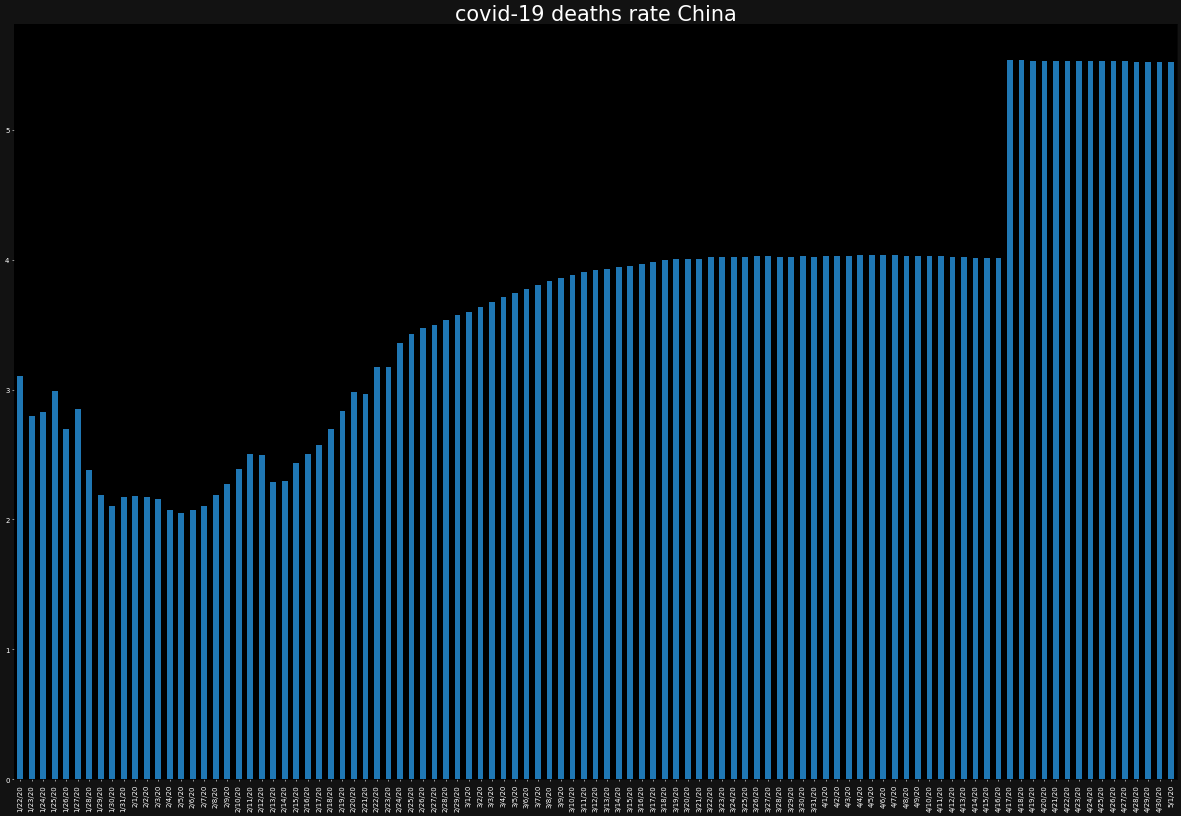
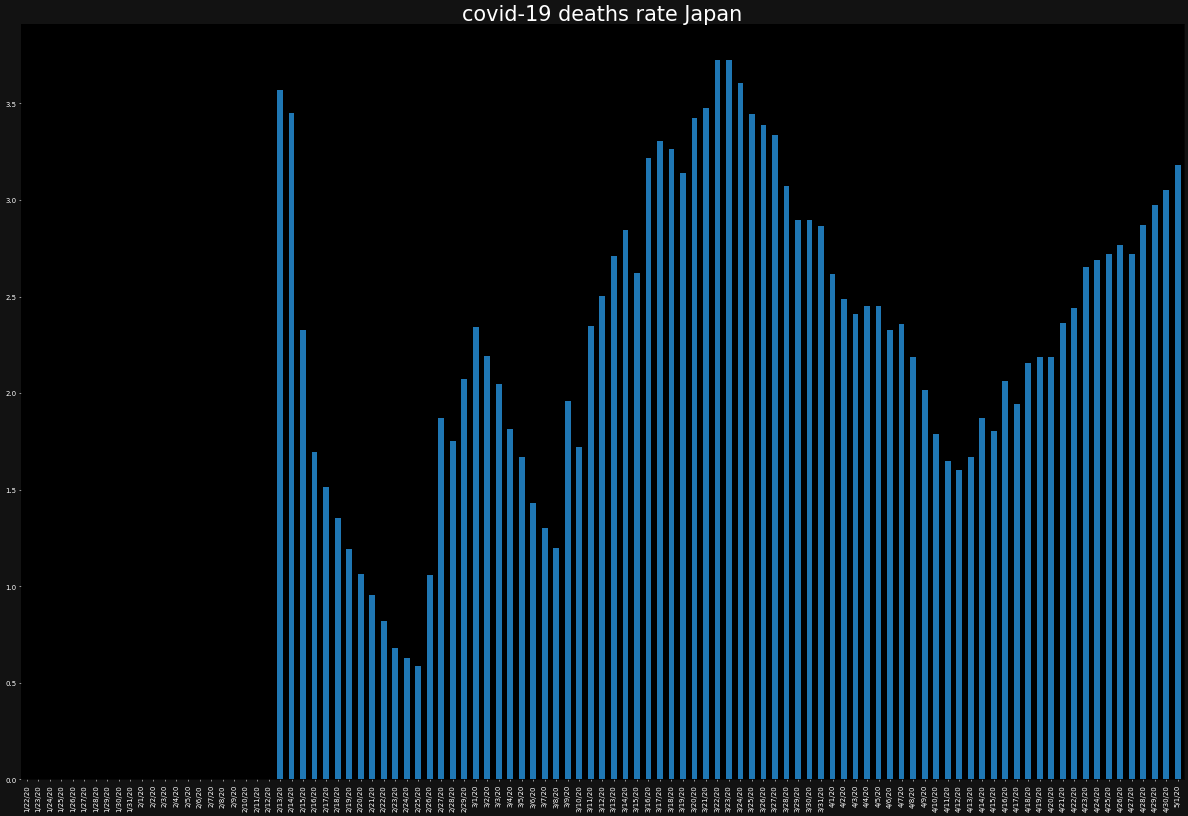
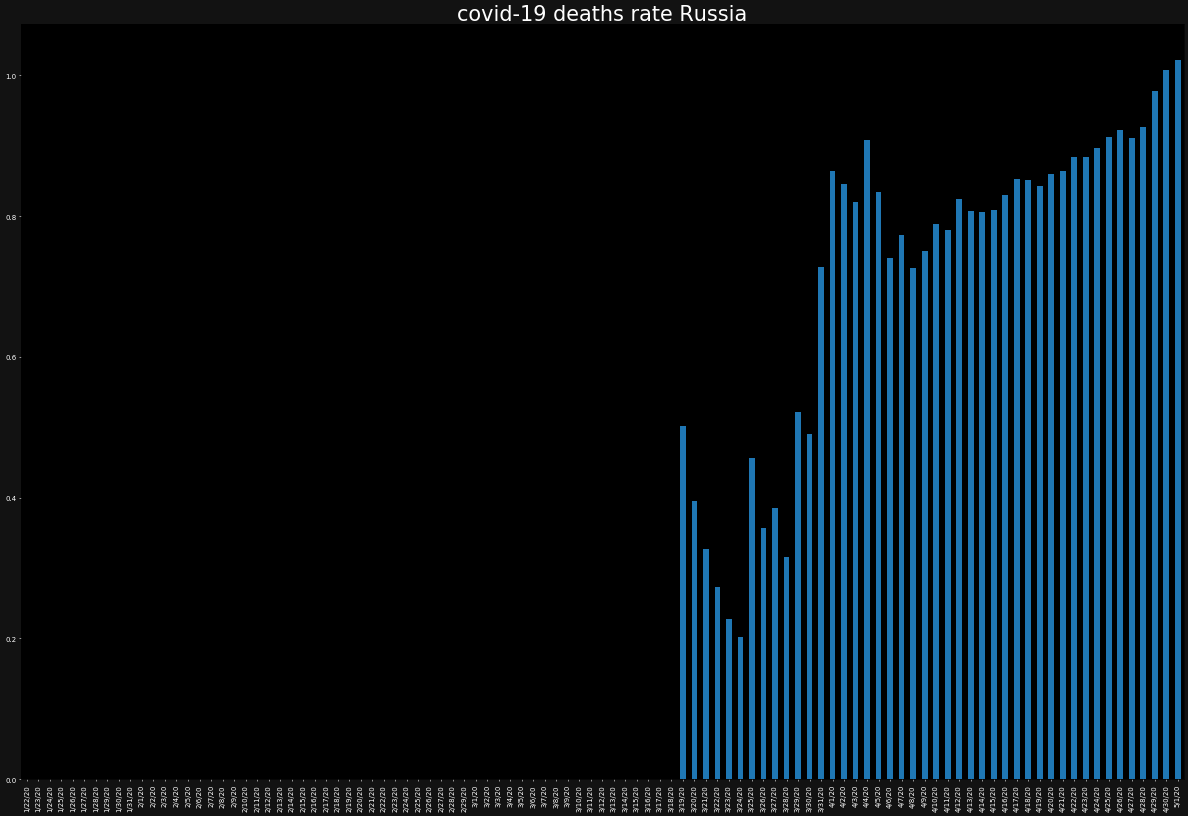
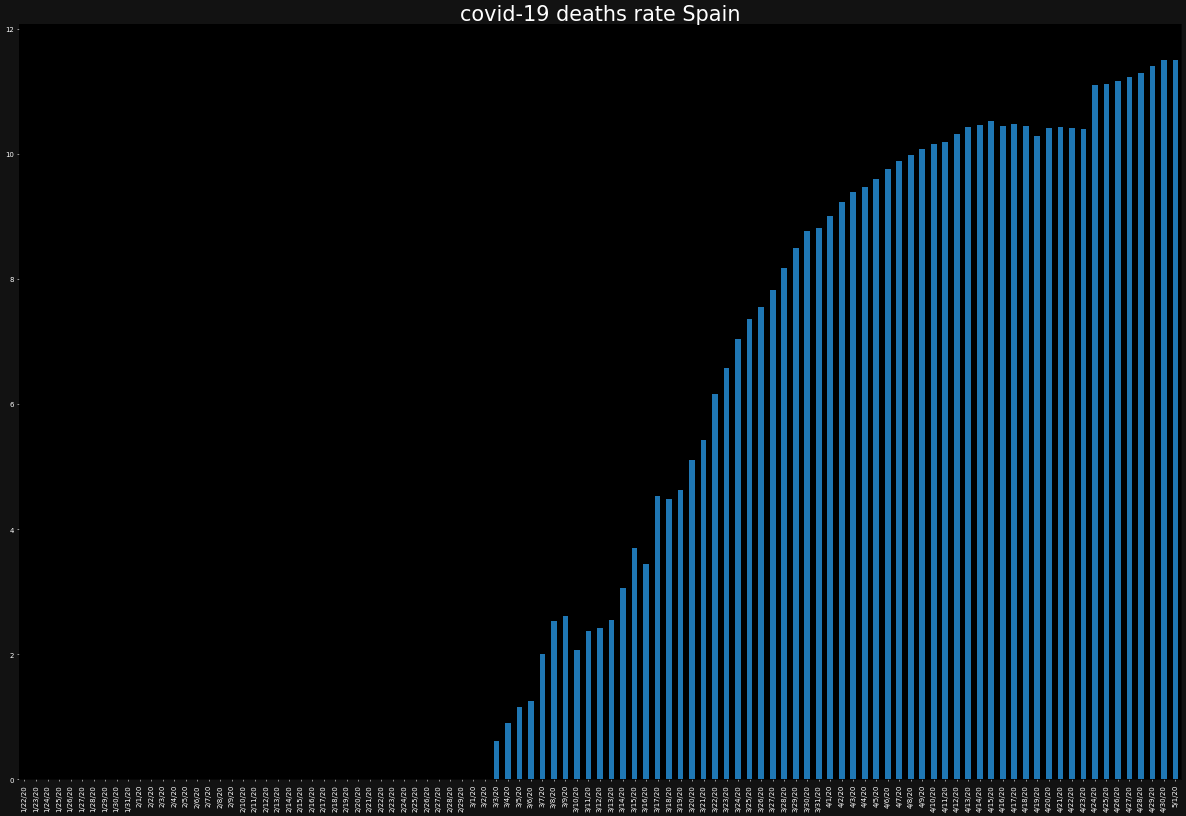
ax.set_title(f"covid-19 deaths rate {country}",color="white")
death_rate[country].plot.bar()
plt.show()
死亡率に国別で差があることが分かります。
さて、最後に今後のコロナウイルスの影響のシミュレーションに移ります。
一旦、今後、仮値として、日次で1%の感染者が増えるとしましょう。
simulated_growth_rate = 0.01
ここで、予測用のこれからの新しい日付データを追加します。
範囲を指定して、日付データを生成できるdate_rangeメソッドを利用します。
今回用いているデータは、05/01/20が最後なので、その次の日から40日間。
dates = pd.date_range(start="05/02/2020",periods=40,freq='D')
dates = pd.Series(dates)
dates = dates.dt.strftime("%m/%d/%Y")
simulated = confirmed.copy()
simulated = simulated.append(pd.DataFrame(index=dates))
for day in range(len(confirmed),len(confirmed)+40):
simulated.iloc[day] = simulated.iloc[day-1] * (1 + simulated_growth_rate)
ax = plt.subplot()
ax.set_facecolor("black")
ax.figure.set_facecolor("#121212")
ax.tick_params(axis="x",colors="white")
ax.tick_params(axis="y",colors="white")
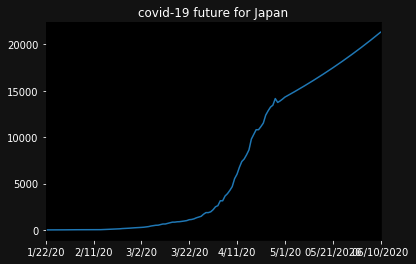
ax.set_title(f"covid-19 future for Japan",color="white")
simulated['Japan'].plot()
plt.show()
※ 念押しですが、これは仮の成長率(※ 日次1%ずつ増え続ける)に基づいた数字です。厳密なシミュレーションとして、受け止める必要はありません。
以上です。ちょくちょく引用元の動画と違う部分はありますが、ざっとデータ分析の流れをつかめたかと思います。
是非、元動画をご覧ください。
投稿者のNeuralNine氏は、動画の最後に、この分析は仮の数値などはあるものだから、真剣に受け止めなくていいことは強調したうえで、事実をデータをもとに把握し、自分が意識すべきことは大事であることを伝えてくれていました。