作業環境
Jupyter Notebook(6.1.4)を用いて作業を進めた。
各versionはpandas(1.1.3), scikit-learn(0.23.2), statsmodels(0.12.0)である。
やりたいこと
最小二乗法で最適化された線形回帰モデルについて学習し、そのモデルを評価する指標として決定係数や自由度調整済み決定係数を学んだ。学習時には手作業で係数を追加したり削除したりしながら決定係数を比較していたが、これを自動探索するプログラムを練習として組んでみたいと考えた。
つまりやりたいことは、データ全体を渡したら最も自由度調整済み決定係数が高くなる説明変数の組み合わせとその自由度調整済み決定係数を返してくれるプログラムを考えて作ってみたい。
なお、その説明変数が説明変数として適切かどうかは特に考慮せず、単純に決定係数を比較して説明変数を選択するのが今回の目的である。
前回は以下の記事で全探索の方法を取り扱った。
そのときの課題として、全探索は時間がかかってしまうという問題があったので、探索方法を工夫して時間短縮を目指したい。今回は増加法・減少法で探索してみる。これは必ずしも最大ではないかもしれないが、工夫によってはそれに近い組み合わせを求められることが期待できる。
探索の方針を考える
今回は説明変数を1つずつ増加(減少)させて自由度調整済み決定係数を計算し、増加(減少)前に比べて大きくなればその自由度調整済み決定係数とそれを与える説明変数を保存していく。増加(減少)する順番については、回帰係数の検定のp値を指標にしたい。これは帰無仮説が"その説明変数の回帰係数が$0$である"としたときのt検定で考えるp値であり、つまりその説明変数が回帰モデルに影響を与えている可能性を考えることができる指標である。このp値が小さいほど回帰モデルに与える影響が大きいと考えて、p値が小さい順(大きい)に説明変数を増加(減少)させていく。
前回はsklearn.linear_modelを用いた実装とstatsmodelsを用いた実装を考えたが、今回はstatsmodelsを使った実装を考えていきたい。理由は以下の2点である。
- 全探索のとき、
sklearn.linear_modelに比べて実行時間が短かった - p値を簡単に得ることができる
特にp値の取り出し方について、後の節で取り扱う。
検証に使用するデータ
scikit-learnのdatasetsの中のボストン住宅データ(説明変数13個)を使って検証していく。以下のように説明変数データセットと目的変数データセットを作成した。
import pandas as pd
from sklearn.datasets import load_boston
X = pd.DataFrame(load_boston().data, columns=load_boston().feature_names)
y = pd.DataFrame(load_boston().target, columns=['target'])
statsmodels.apiを用いてp値を取得する
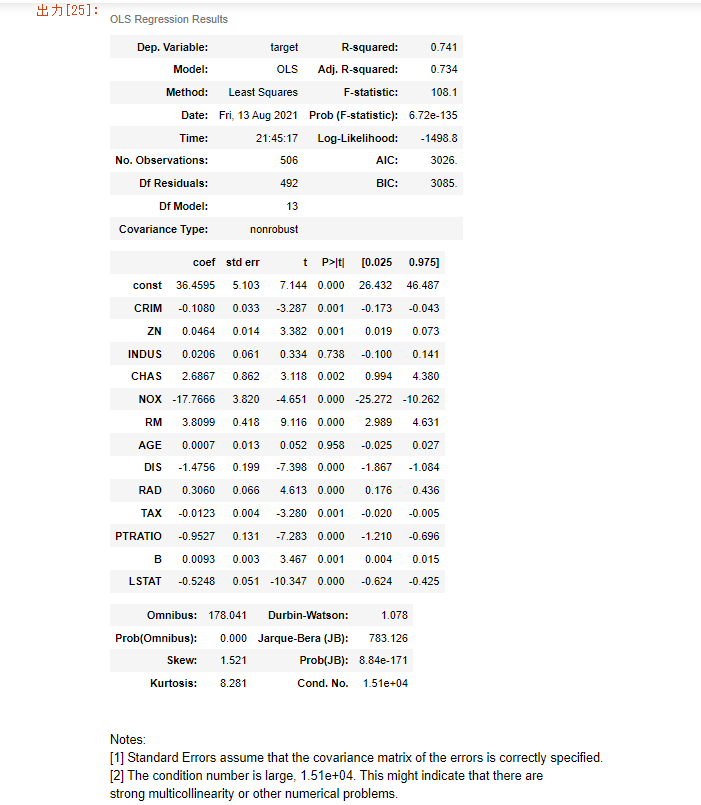
statsmodels.apiでは'summary`を使うことで基礎統計量を一括で取得することができる。
import statsmodels.api as sm
X1 = sm.add_constant(X)
m = sm.OLS(y, X1)
result = m.fit()
result.summary()
この中でも各説明変数の$P>|t|$列に表示されている値を使って説明変数を並べ替えたい。p値の取得は以下で行うことができる。
result.pvalues
# --->
# const 3.283438e-12
# CRIM 1.086810e-03
# ZN 7.781097e-04
# INDUS 7.382881e-01
# CHAS 1.925030e-03
# NOX 4.245644e-06
# RM 1.979441e-18
# AGE 9.582293e-01
# DIS 6.013491e-13
# RAD 5.070529e-06
# TAX 1.111637e-03
# PTRATIO 1.308835e-12
# B 5.728592e-04
# LSTAT 7.776912e-23
# dtype: float64
それぞれの値はresult.pvalues[i]のようにインデックスを指定して得ることができる。
for i in range(X.shape[1]):
print('{} : {:.3f}'.format(X1.columns[i], result.pvalues[i]))
# --->
# const : 0.000
# CRIM : 0.001
# ZN : 0.001
# INDUS : 0.738
# CHAS : 0.002
# NOX : 0.000
# RM : 0.000
# AGE : 0.958
# DIS : 0.000
# RAD : 0.000
# TAX : 0.001
# PTRATIO : 0.000
# B : 0.001
# LSTAT : 0.000
結果を見てみるとほかの説明変数に比べてINDUS列とAGE列のp値が大きい。これらは全探索のときに選ばれなかった特徴量である。
増加法による探索
全探索探索を参考に以下のように実装した。
def R2search_up(X, y):
"""自動調整済み決定係数が最も大きい説明変数を探索する(増加法)
パラメータ
-----------
X : 説明変数
y : 目的変数
戻り値
-----------
best_r2, best_col : 最も大きい自由度調整済み決定係数とそれを与える説明変数のリスト
"""
x_col = X.columns
#まずは説明変数全体でモデルを作成し、p値が小さい順に説明変数を並び替える
#モデルの作成
X = sm.add_constant(X)
model = sm.OLS(y, X)
result = model.fit()
#説明変数とそのp値のペアのリストを作成
list = [[x_col[i], result.pvalues[i+1]] for i in range(len(x_col))]
#ペアの2番目の要素について昇順に並び替え
list.sort(key = lambda x:x[1])
#ペアの1番目の要素を取り出してx_colを更新する
x_col = [list[i][0] for i in range(len(x_col))]
#最大値を保存するオブジェクトの作成
best_col = []
best_r2 = 0
col = []
for i in range(len(x_col)):
#x_colから特徴量をp値が小さい順に追加
col.append(x_col[i])
X_train = X[col]
X_train = sm.add_constant(X_train)
model = sm.OLS(y, X_train)
result = model.fit()
r2 = result.rsquared_adj
#r2が今までのものより大きいのであれば更新
if r2 >= best_r2:
best_r2 = r2
best_col = col.copy()
return best_r2, best_col
検証データで動作を確かめてみたところ、次のような結果が得られた。
print(R2search_up(X, y))
# ---> (0.7348057723274566, ['LSTAT', 'RM', 'DIS', 'PTRATIO', 'NOX', 'RAD', 'B', 'ZN', 'CRIM', 'TAX', 'CHAS'])
全探索のときと同じ結果を得ることができた。実行速度を%%timeitで確認すると以下の通りであった。
%%timeit
print(R2search_up(X, y))
# ---> 45.5 ms ± 1.63 ms per loop (mean ± std. dev. of 7 runs, 10 loops each)
# 前回定義したstatsmodelsを使用した全探索R2seach2での結果を表示する
%%timeit
print(R2search2(X, y))
# ---> 39.2 s ± 3.62 s per loop (mean ± std. dev. of 7 runs, 1 loop each)
比較してみると、かなり時間を短縮して求めることができたことがわかる。
減少法による探索
同様にして減少法による実装を行う。
def R2search_down(X, y):
"""自動調整済み決定係数が最も大きい説明変数を探索する(減少法)
パラメータ
-----------
X : 説明変数
y : 目的変数
戻り値
-----------
best_r2, best_col : 最も大きい自由度調整済み決定係数とそれを与える説明変数のリスト
"""
x_col = X.columns
#まずは説明変数全体でモデルを作成し、p値が小さい順に説明変数を並び替える
#モデルの作成
X = sm.add_constant(X)
model = sm.OLS(y, X)
result = model.fit()
#説明変数とそのp値のペアのリストを作成
list = [[x_col[i], result.pvalues[i+1]] for i in range(len(x_col))]
#ペアの2番目の要素について降順に並び替え
list.sort(key = lambda x:x[1], reverse=True)
#ペアの1番目の要素を取り出してx_colを更新する
x_col = [list[i][0] for i in range(len(x_col))]
#最大値を保存するオブジェクトの作成
best_col = []
best_r2 = 0
for i in range(len(x_col)-1):
X_train = X[x_col]
X_train = sm.add_constant(X_train)
model = sm.OLS(y, X_train)
result = model.fit()
r2 = result.rsquared_adj
#r2が今までのものより大きいのであれば更新
if r2 >= best_r2:
best_r2 = r2
best_col = x_col.copy()
#p値の大きいほうから説明変数を減らす
x_col.pop(0)
return best_r2, best_col
検証データで動作を確かめてみたところ、次のような結果が得られた。
print(R2search_down(X, y))
# ---> (0.7348057723274566, ['CHAS', 'TAX', 'CRIM', 'ZN', 'B', 'RAD', 'NOX', 'PTRATIO', 'DIS', 'RM', 'LSTAT'])
こちらでも全探索のときと同じ結果を得ることができた。実行速度を%%timeitで確認すると以下の通りであった。
%%timeit
print(R2search_down(X, y))
# ---> 42.6 ms ± 1.7 ms per loop (mean ± std. dev. of 7 runs, 10 loops each)
こちらでもやはり全探索よりも早く探索することができている。
課題
次のような課題が考えられる。
- 最初にも書いたが、戻り値が自由度調整済み決定係数の最大である保証はない
- 自由度調整済み決定係数がどのくらい特徴量増加の影響を抑えられているのかが(知識不足で)わからない
一番目について、検証データでは補って余りある実行時間の差があった。ただ、別のデータを使った時に本当の最大値とどのくらい差がでるのか確証がもてない。
二番目について、例えば増加法では特徴量が毎回増加するので決定係数は必然的に増加する。私に統計学の知識が足りないので、本当に特徴量増加の影響を抑えられているのかを論じることができない。ここをもう少し調べてみたい。