目標・目的
SDFファイルから記述子を求め、csvファイルとして扱えるようにする。記述子ってなに?
記述子とは、分子構造から数値化できるものを表したものである。(csvファイルの形にするようなもの) >引用元(https://www.yodosha.co.jp/jikkenigaku/keyword/4091.html) >> 化合物の化学的特徴を,部分構造の特性や物理化学的性質など数値化してあらわした特徴ベクトルを意味する >引用元2(https://future-chem.com/rdkit-descriptors/) >>記述子(デスクリプター)とは分子の性質を決める指標のこと記述子が必要な理由
SDFファイルとは、複数の分子を1つのファイルに納めたファイルのことである。(創薬などでは、データの受け渡しがSDFファイルを使っているらしい) SDFファイルの中身は、取得するDB([pubchem](https://pubchem.ncbi.nlm.nih.gov/)や[PDB](https://pdbj.org/))によっては、カラム名(変数名)がかなり変わってくるが、ほとんどは、「分子量」や「SMILES」や「モル質量」などの基本情報しか載ってないため、記述子は計算などをして、導かないといけない。 しかし、この記述子は数値となっていることから、機械学習やDeepLearningに使うことができる。現在では、スクリーニング(特徴抽出)やたんぱく質とリガンドとの相互作用分析など、さまざまなところで、最先端の技術が使われている。 例)https://www.jstage.jst.go.jp/article/tjsai/29/1/29_D-TS13_12/_pdf/-char/ja (2014年) https://www.jstage.jst.go.jp/article/jccj/2/4/2_4_119/_pdf実装
https://datachemeng.com/pythonassignment/ ここのサイトからダウンロードしたSDFファイルを使って、csvファイルを作成していきたいと思います。(ちなみに、上のサイトのコードも見本にさせていただきました。ありがとうございます)rdkit.py
from rdkit.Chem import Draw, PandasTools
from rdkit import Chem
import pandas as pd
from rdkit.Chem import Descriptors
from rdkit.ML.Descriptors import MoleculeDescriptors
これらのライブラリをインポートします。
rdkit.py
mols = Chem.SDMolSupplier('logSdataset1290_2d.sdf')
mols #=> rdkit.Chem.rdmolfiles.SDMolSupplier....
まずは、SDMolSupplierで、SDFファイルから取得。
しかし、変数molsの中身が見えないため、見たい人は、PandasToolsを使うことで、DataFrameで見ることができる。
rdkit.py
df = PandasTools.LoadSDF('logSdataset1290_2d.sdf')
df
データ整理している
rdkit.py
y_name = 'logS'
mols = [mol for mol in mols if mol is not None ] #=> SDMolSupplierは入力構造に不備があるとNoneになるため、
len(mols) #=>1290
rdkit.py
descriptor_names = [descriptor_name[0] for descriptor_name in Descriptors._descList]
len(descriptor_names) #=> 200
Descriptors._descListは、記述子とその計算メソッドがある。200個もの記述子があるが、それは一部分らしい。
rdkit.py
descriptors = pd.DataFrame([descriptor_calculation.CalcDescriptors(mol) for mol in mols])#for文で一つ一つ分子を計算している
そして、DataFrameに計算された数値を入れていく。
rdkit.py
descriptors.columns = descriptor_names
smile = [Chem.MolToSmiles(mol) for mol in mols] #=> smileを取得する
descriptors.index = smile
y = pd.DataFrame([float(mol.GetProp(y_name)) for mol in mols]) #分子のlogSを取得
y.index = smile
y.columns = [y_name]
dataset = pd.concat([descriptors, y], axis=1)
dataset.to_csv('dataset.csv') #=> dataset.csvが出力された
yにlogsを取得しているが、logsは化合物の水に対する溶解度の指標であり、 高いと水に対する溶解度は高いことが言える。創薬などでは重要な指標となっている。
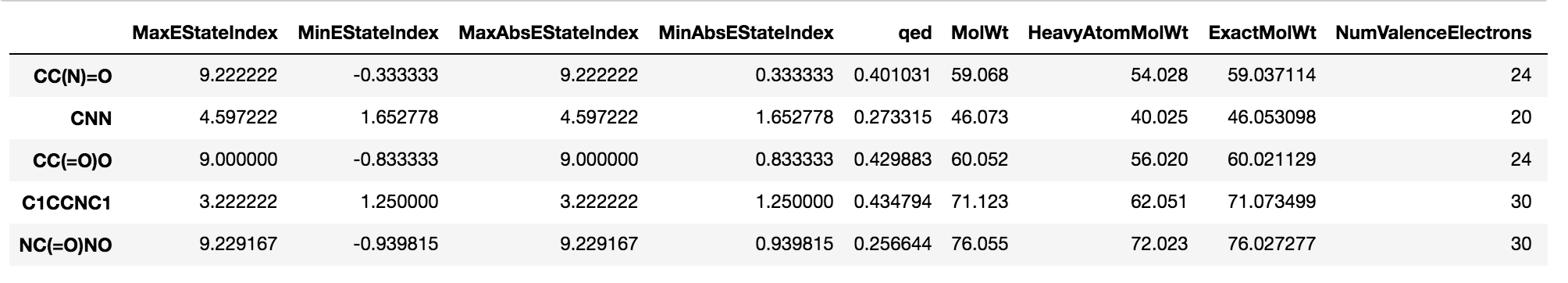
上の写真が、csvファイルにした時の数値である。
これで、構造分子を数値化になったので、ここから、PCA(主成分分析)や深層学習に当てはめていき、類似度の評価や相互作用分析に使えるようにしていきたい。
※これを書いた人は、バイオ・ケモインフォマティクスをほとんどわかっていないため、間違いがあれば指摘していただきたいです。