Circlatorって?

細菌ゲノムファイルの環状化の処理ツールです.細菌ゲノムは染色体とプラスミドに分けられ、一般的にどちらも環状化構造を取ります。
ナノポアリード(ONT配列)をアセンブリすると、ゲノムファイル(FASTA配列)として,染色体とプラスミドの配列が別々に取得できますが、これら配列両端に重複配列や足りない配列が含まれる可能性があります。環状ゲノムとして扱うには、重複配列の削除や不足配列の補填などの環状化処理をしなきゃいけないです.その時にCirclatorを使います.
本記事ではCirclatorの機能を紹介します!また,実際に筆者が使ってみた記録も残します.読者の方々には,Circlatorをどのような時に使うのか参考になれば幸いです.なお,説明はバイオインフォマティクス初学者に対応したものにしました.
インストールしよう!

インストールはconda create -n circlator -c bioconda -c conda-forge circlatorって感じにCondaで行けます.OSに関して,Linuxではインストールできました.ただ,M1 Macではインストールできるもののプログラムをランする時エラーが出てしまいました.使用の際にOSを検討すべきだと考えてます.
以下に論文と公式サイトを提示します.
Circlatorの論文
GitHub
環状化させるってどういうこと?
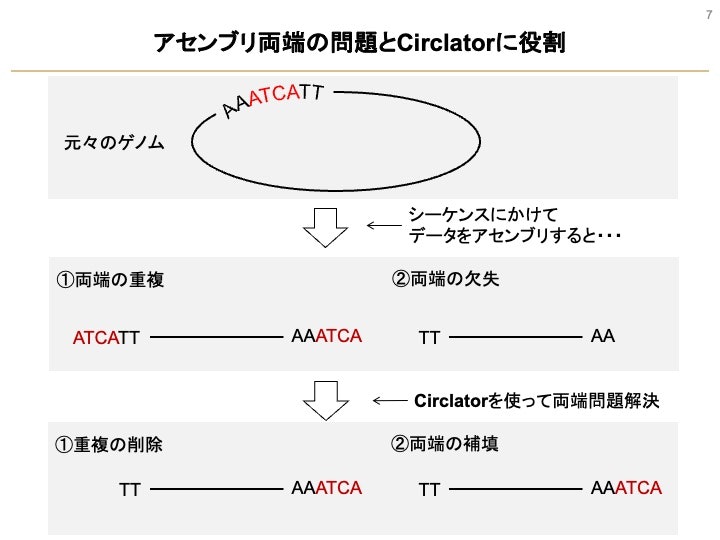
ONT配列からアセンブリして得られたFASTA配列はそのまま環状ゲノムとしては使えないです.なぜなら,配列の両端に重複や欠失がある可能性が高いためです.
の重複の削除た欠失の補填を行うことを環状化させる定義されます.この作業はマニュアルでやるとなると,FASTA配列の両端を切って,BLASTとして,重複部分を消して・・・.と言う感じにめちゃくちゃ面倒です.絶対ミスがでます.
Circlatorはこれら作業を自動化してくれた長所です.
Circlatorは何ができるの?
Circlatorが何をやっているか図で説明します.

①まず,ONT配列をFASTA配列にマッピングします.マッピングは,どのリードがFASTA配列のどの場所に対応しているか解析する方法です.
②マッピングの結果から,FASTA配列の両末端からCutoff値の幅に存在するリードを抽出する.
③抽出したリードでアセンブリする.

④アセンブリをFASTA配列にアライメントする.アセンブリが両末端と高い相同性を示せば,末端配列はアセンブリの配列に置換される.
⑤最後に,片方の配列がトリムされて,環状化が完了する.
成功の鍵は,④のアライメントがうまくいくかです.アセンブリがFASTA配列の両末端と相同性が高くなければ,環状化されないと出力されます.
早速使ってみよう!

このコマンドで一発です.
circlator all genome.fasta ONT.fastq.gz output_dir
出力として,なんか色々でますが重要なのは最後です.
Circularized 3 of 3 contigs
これは3つのコンティグ中3つとも環状化できたことを示します.これが出てきたら万々歳です.
やったね!

そうです.実際にはエラーがバンバン出ます.
そんなうまくいきませんよね.ここからが本番です.
筆者の例
色々苦戦したので,その記録を載せます.トラブルシューティングの参考になれば幸いです.
筆者の例で説明します.
まず,前提です.
- 菌株XのFASTA配列(コンティグ3つ)とONT配列を使う.
- FASTA配列は染色体1本(5.0Mbp)とプラスミド2本(200kbpと50kbp)で構成されることがわかっている.
この前提をもとにやっていきます
まずcirclator allした結果
Circulaized 1 of 3 contigs
となりました.記録を見るとプラスミド一本だけが環状化していました.
色々解析したところ,以下の手順を踏むとうまくいきました.
- 3つのコンティグを別々のファイルに分ける(
chr.fasta,plasmid_1.fasta,plasmid_2.fasta) - ONT配列をFASTA配列にマッピングして,どのリードがどのコンティグに対応しているか解析し,コンティグ毎のリードにファイルを分ける(
chr.fastq.gz,plasmid_1.fastq.gz,plasmid_2.fastq.gz) - 各コンティグFASTAとONTを一対一でCirclatorにかけた.この時Cutoff値を調節すると,全てのファイルが環状化できた.
これに至った経緯と実際どのように分けたかについては,長いので別記事にまとめようと思ってます.
最後に
今回はCirclatorの紹介をしました.
みなさんも,バクテリアでナノポアする場合,ぜひ参考にしてください!!
