FASTQって?
ゲノムシーケンスデータの形式です。イルミナなどによって読まれた塩基配列とその**クオリティスコアがテキストベースで記載されています。クオリティスコアはASCII文字で表されます。そのため、基本的に内容は見れないのですが、「何が書かれているか」を知っておく事は必要です。
今回はその構成を、自身の備忘録的な役割を兼ねて紹介します。
FASTQの内容を知ろう
どんな構成?
1リード分の構成は以下の様になっている。
1行目 ID(ヘッダー)
2行目 塩基配列
3行目 IDまたは改行
4行目 信頼値
1行目のIDは配列に紐付けされる名前みたいなものです。ゲノムがペアエンドで呼ばれた場合、ペア同士を対応させるためにも必要です。
2行目の配列の長さは、シーケンサーによって変わります。例えば、リード長が100baseであれば、2行目には100個分の塩基配列が記載されます。
3行目では、IDが再度記載されたり、改行されたりします。行頭に+と記されます。
4行目では、各塩基単位の信頼値が記載されます。
このセットが、幾つも一つのファイルにまとめられます。
例えば以下のような記載です。こちら
から引用。
@SRR1170086.1 HWI-ST845:120525:D10G7ACXX:8:1101:9728:1999 length=50
GTTTTTAAAATGAGTTTGCAAATATTACTGTATTTTTNTCCCATGCTTTT
+SRR1170086.1 HWI-ST845:120525:D10G7ACXX:8:1101:9728:1999 length=50
11=DDFFFFHHGFIGGIIJJJJIJJJJJJIIIIIJJJ#1?DHIJJIJIII
@SRR1170086.2 HWI-ST845:120525:D10G7ACXX:8:2316:19897:100799 length=50
CGATGGAATAGATTTCTCCAAGTTAGTTGGAGGCTAGTCTTCTCTACATA
+SRR1170086.2 HWI-ST845:120525:D10G7ACXX:8:2316:19897:100799 length=50
???DADDA:++22C::4:::AA33C,,,2EFECB;ED9D4?4*0?D0B9B
クオリティスコアの意味は?
Fastq形式4行目の信頼値はASCII文字で記載されます。ASCII文字から数値(ASCIIcode)に変換し、数式に代入することでクオリティスコア(Q)が出せます。さらにその値を数式に代入することで、シークエンシングエラー(Perror)を導出できます。シークエンシングエラーは、対応する塩基がエラーである確率を意味します。
以下数式です。
Q=(ASCIIcode)-33\\
P_{error}=10^{\frac{-Q}{10}}
クオリティスコアを出してみよう
上記のFASTQの例を用います。
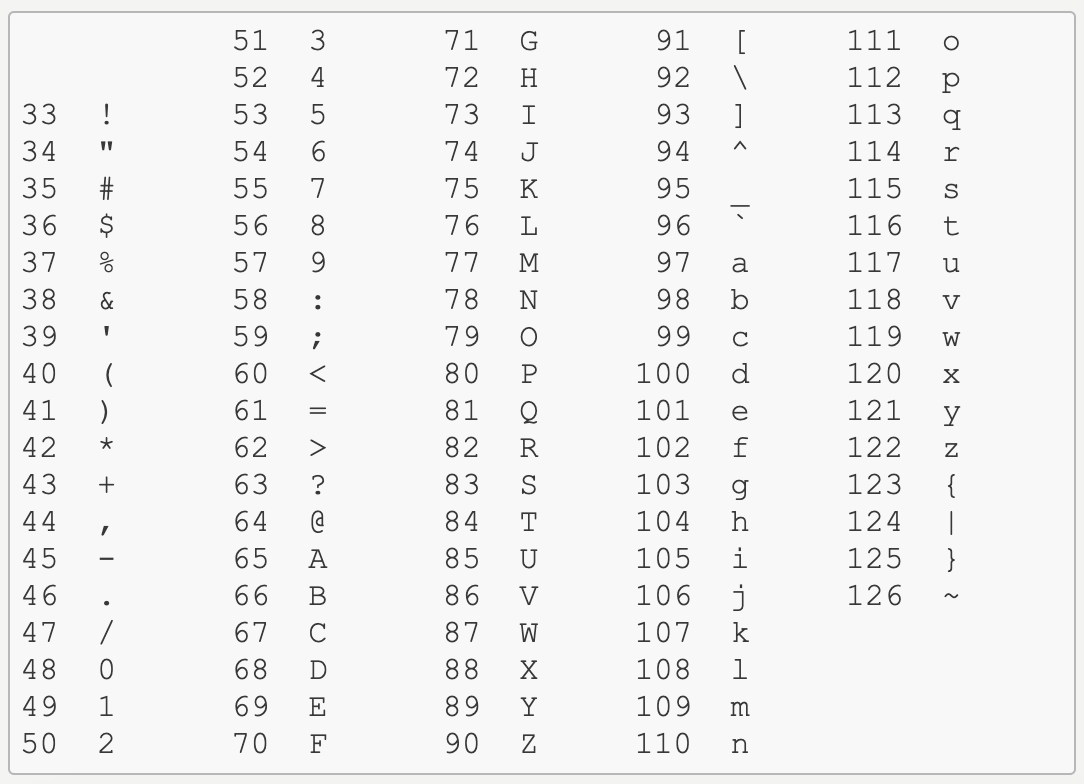
ID:SRR1170086.1の4行目左から3個目の「=」で考えます。
ASCII表をみると=は61なので、*Perror*は以下のように導出できます。
ASCIIcode=61\\
Q=61-33=28\\
P_{error}=10^{\frac{-28}{10}}=0.0015848
すなわち、『3個目のTがエラーである確率は0.0015848』という事です。
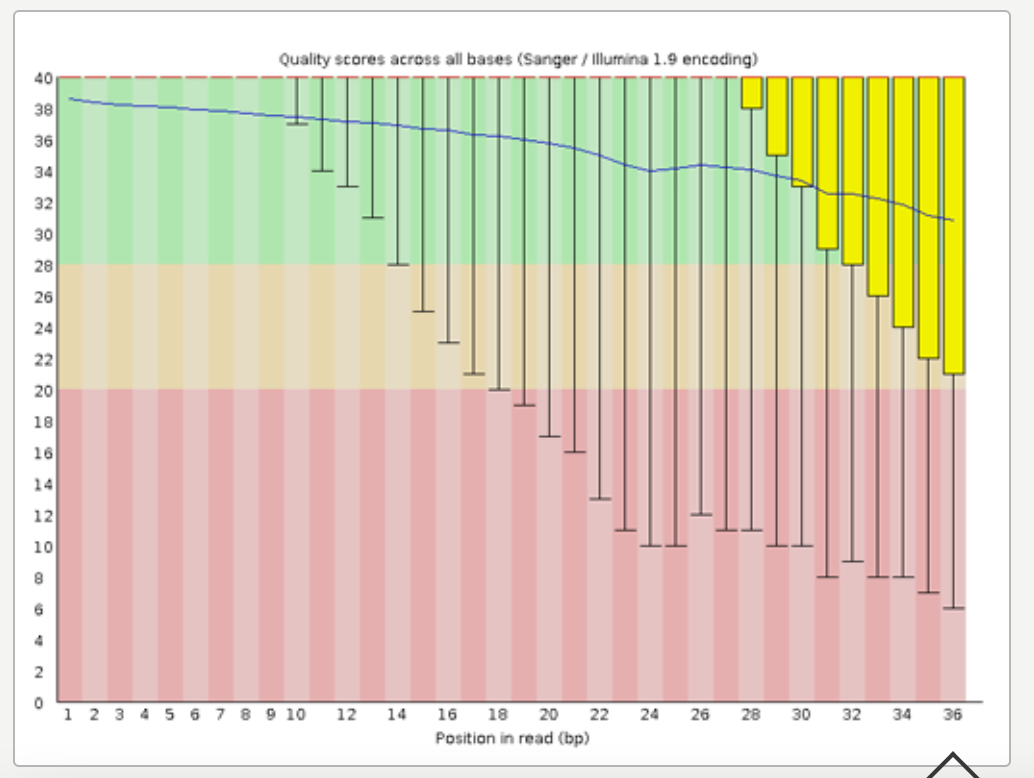
余談 エラーの確率は後配列になるほど高い?
下図は配列ごとのクオリティスコアをグラフしたものです。配列が右(後ろ)になるにつれ、スコアが低下していることがわかります。上述した数式を見ればわかるのですが、スコアが低いほどエラーは大きくなります。
実際にFASTQを用いる際は、クオリティチェックとトリミングが欠かせません。これに関しては別記事にてまとめます。
終わりに
FASTQを用いる際は、内容を把握することが大切です。