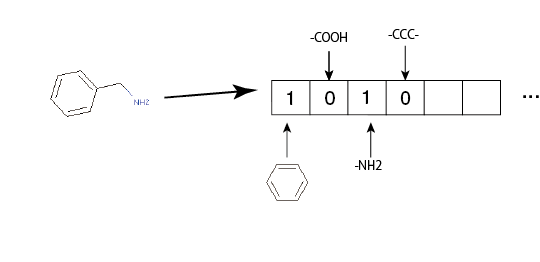
概略
David Duvenaud et al, "Convolutional Networks on Graphs
for Learning Molecular Fingerprints", NIPS 2016(https://arxiv.org/abs/1509.09292) をKerasを用いて実装しました
ソースコード[https://github.com/mpozpnd/NeuralFingerprint-Keras]
化合物のベクトル化
機械学習を用いて、化合物のデータから化合物のなんらの物性を予測をしたいということがあります。
たとえば創薬において、ウェットな実験によって既に所望の薬剤としての性質の有無が分かっている化合物のデータによって学習モデルを構築し、新規の化合物について薬剤としての性質を持つかどうか予測をする、というタスクがあります。
なぜ学習ができるのかというと、「†似た†1構造をもつ化合物は似たような性質をもつ」という傾向があるためです。
たとえば、(わりと極端な例ですが)
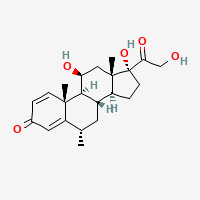
プレドニゾロン
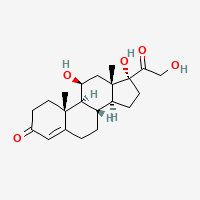
ヒドロコルチゾン
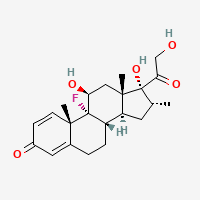
デキサメタゾン
はそれぞれ販売されているステロイド抗炎症薬で、体内で似たような働きをするのですが、なんとなく似ているor似た部分構造を持っていることがわかります
そこで、化合物の構造を表現するような特徴を用いて学習モデルを構築することになります。
ここで問題が二つあります。
-
二つ目として、例えば画像であればリサイズにより同じ長さのデータに揃えることができますが、化合物は一つ一つ大きさ(この場合、含まれる原子の数など)が異なるということです。
このため、何らかの方法で、化合物をその構造をよく表すような固定長の特徴量ベクトルに変換する、という手法が必要になります。
基本的によく使われる考え方としては、
- ある決まった長さのビットのベクトルを用意します
- ビットごとに、特定の部分構造を割り当てる。たとえば、ベクトルを$v$として、$v[0]$はベンゼン環に、$v[1]$はアミノ基に...というような感じです
- ある化合物について、それぞれの部分構造を持っていれば該当するビットを1に、存在しなければ0にします
こうすることで、グラフ構造を持つ化合物を固定長の特徴ベクトルに変換することができます。このうち2.のわり当てる方法について、様々な手法が提案されています。
- daylight
- maccskey
- ECFP
- etc.
ECFP
Extended Connectivity Fingerprint(ECFP)はよく使われる化合物の特徴量化手法です。
アルゴリズムとしては、
- 化合物に含まれるそれぞれの原子について、そこから距離1,距離2,距離3...の部分構造をすべて取り出す
- 1.で得た全ての部分構造について、Morgan法により一意の整数に変換する(ハッシュ化)
- 2.を規定のビット数(長さ2048がよく使われます)でmodをとって得られた番地のビットを1にする
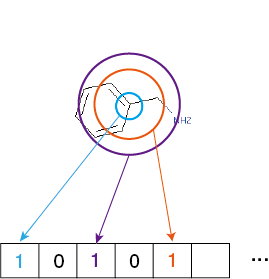
という感じです。あらかじめ割り当てのための部分構造を人手で用意する必要がなく、それゆえに多くの部分構造を特徴量に含めることができます。
NeuralFingerprint
NeuralFingerprint(以下、NFP)NIPS2016にて提案された手法です。NFPは、ECFPのアルゴリズムの一部をニューラルネットに置き換えたものになります。
ECFPにおけるハッシュ関数はsigmoidなどの活性化関数に置き換わり、modがsoftmaxに置き換わっています
ここで注目するべきは、仮にネットワークが持つ重みの絶対値が非常に大きい場合、NFPはECFPにほぼ近似できるという点です。
この場合、NFP9行目のσは出力が1か0の二値関数に近似され、これを$r_a$の次元で結合したものはハッシュ関数とみなすことができます。
またNFP10行目のsoftmax関数は、出力のどこか一つだけ1であとは0になるargmaxと同じ挙動になり、結果的にmodと同じ役割を果たすことになります。
このことから、ECFPはNFPの特別な場合である、言い換えればNFPはECFPの一般化であると言えます。
実装
レイヤーのソースコードを載せます
どうしてもレイヤーの計算の中で化合物ごとに異なる原子数の情報が必要になってしまうので、入力ベクトルの最初の要素にこれを持たせ、内部で無理やり取り出して利用しています。
class NFPLayer(Layer):
'''
NeuralFingerprint implementation with keras
# Arguments
N_dim_atom: dimension of atom_feature
N_dim_bond: dimension of bond_feature
radius: radius od NFP(default: 4)
large_weights: if True, NFP layer has big random weight(to compare with ECFP)
'''
def __init__(self, output_dim, N_dim_atom, N_dim_bond, radius=4,
init='glorot_normal', large_weights=False, H_regularizer=None,
W_regularizer=None, **kwargs):
self.output_dim = output_dim
self.N_dim_atom = N_dim_atom
self.N_dim_bond = N_dim_bond
self.radius = radius
self.init = initializations.get(init)
self.H_regularizer = regularizers.get(H_regularizer)
self.W_regularizer = regularizers.get(W_regularizer)
self.large_weights = large_weights
super(NFPLayer, self).__init__(**kwargs)
def build(self, input_shape):
h_len = self.N_dim_atom + self.N_dim_bond
self.H = [self.init((h_len, h_len), name='{}_H{}'.format(self.name, i)) for i in range(self.radius)]
self.W = [self.init((h_len, self.output_dim), name='{}_W{}'.format(self.name, i)) for i in range(self.radius)]
if self.large_weights:
self.H = [h * 1000000 for h in self.H]
self.W = [w * 1000000 for w in self.W]
self.regularizers = []
if self.H_regularizer:
for h in self.H:
self.H_regularizer.set_param(h)
self.regularizers.append(self.H_regularizer)
if self.W_regularizer:
for w in self.W:
self.W_regularizer.set_param(w)
self.regularizers.append(self.W_regularizer)
self.trainable_weights = self.H + self.W
def call(self, x, mask=None):
atom_num = x[0].astype("int32")[0]
output = K.variable(np.zeros([self.output_dim]))
atom_feature_len = atom_num * self.N_dim_atom
adj_mat_len = atom_num * atom_num
bond_atom_len = atom_num * atom_num * self.N_dim_bond
# reshape 1-d vector to 2-d and 3-d matrix
feature = K.theano.tensor.reshape(x[:, 1:1 + atom_feature_len], [atom_num, self.N_dim_atom])
adj_mat = K.theano.tensor.reshape(x[:, 1 + atom_feature_len: 1 + atom_feature_len + adj_mat_len], [atom_num, atom_num])
bond_feature = K.theano.tensor.reshape(x[:, 1 + atom_feature_len + adj_mat_len: 1 + atom_feature_len + adj_mat_len + bond_atom_len], [atom_num, atom_num, self.N_dim_bond])
for i in range(self.radius):
bond_ = K.theano.tensor.diagonal(K.dot(adj_mat, bond_feature), 0, 1, 0).T
atom_ = K.dot(adj_mat, feature[:, :self.N_dim_atom])
v = K.theano.tensor.concatenate([bond_, atom_], axis=1)
feature = K.sigmoid(K.dot(v, self.H[i]))
i = K.softmax(K.dot(feature, self.W[i]))
output += K.sum(i, axis=0)
return output.dimshuffle(['x', 0])
def get_output_shape_for(self, input_shape):
return (input_shape[0], self.output_dim)
実験
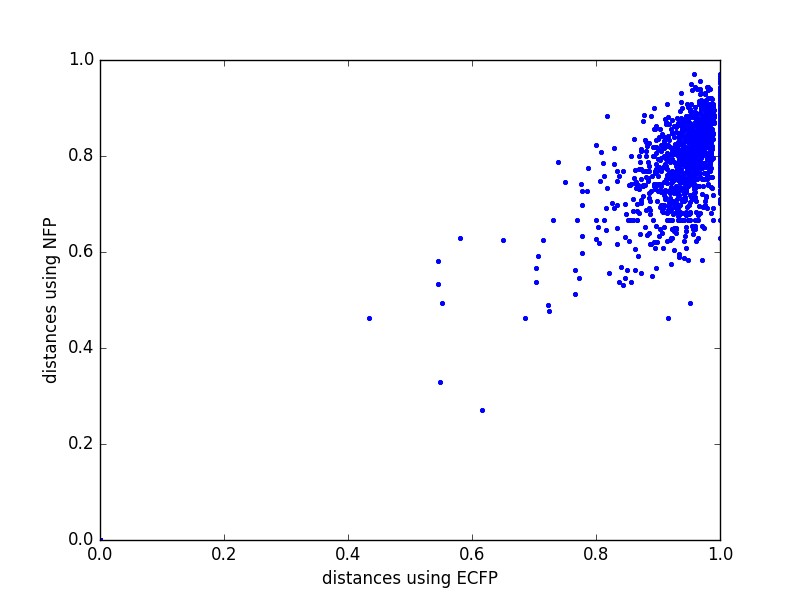
まず、大きな重みで初期化したネットワークがECFPと(ほぼ)等価であることを確認します。
比較としては、
- 複数の化合物をECFPにより変換し、それぞれのベクトル同士の距離を計算します
- 同じ化合物をNFPにより変換し、それぞれのベクトル同士の距離を計算します
- 1.と2.の相関係数を計算します
二つのベクトルの距離は以下のように定義します(tanimoto係数を入力が実数でも計算できるように一般化したもの)
distance(x, y) = 1 - \sum min(x_i, y_i) / \sum max(x_i, y_i)
import os
import sys
sys.path.append(os.curdir)
import numpy as np
import matplotlib.pyplot as plt
from keras import backend as K
from nfp_keras.nfplayer import NFPLayer
import argparse
ps = argparse.ArgumentParser()
ps.add_argument('input_data', type=str)
ps.add_argument('ecfp4_data', type=str)
args = ps.parse_args()
def distance(x, y):
tmp = np.vstack([x, y])
return 1 - tmp.min(axis=0).sum() / tmp.max(axis=0).sum()
ecfp = np.load(args.ecfp4_data)[:50]
data_x = np.load(args.input_data)[:50]
layer = NFPLayer(2048, 62, 5, large_weights=True) # 重みを全て100万倍させます
layer.build(input_shape=24586)
nfp = np.vstack([layer.call(K.variable(x.reshape([1,x.shape[0]]))).eval() for x in data_x])
dists_ecfp = np.hstack([distance(x, y) for x in ecfp for y in ecfp])
dists_nfp = np.hstack([distance(x, y) for x in nfp for y in nfp])
print(dists_ecfp.sum())
print(dists_nfp.sum())
plt.plot(dists_ecfp, dists_nfp, '.')
plt.savefig('compare.png')
print('correlation of 2 FP:%f' % np.corrcoef(dists_ecfp, dists_nfp)[0, 1])
correlation between two FP:0.866761
実際に高い相関係数が得られることがわかります
次に、元の実装 論文で使われているデータセットと同じものを使って溶解度の予測(回帰)をしてみます。
論文と同じように、NFP側はnfpレイヤーの上に1024unitの線形レイヤ載せたモデル、ECFP側は入力が2048次元、1024unitの線形レイヤを使います。
なんかNFP側の方がパラメータがはるかに多いのでフェアじゃない気がしますが。
- NFP
import os
import sys
sys.path.append(os.curdir)
from keras.layers import Dense, Activation
from keras import regularizers
from keras.models import Sequential
from nfp_keras.nfplayer import NFPLayer
import numpy as np
import argparse
ps = argparse.ArgumentParser()
ps.add_argument('input_data_x', type=str)
ps.add_argument('input_data_y', type=str)
args = ps.parse_args()
model = Sequential()
model.add(NFPLayer(2048, 62, 5, input_shape=(24586, ) ))
model.add(Dense(1024, input_dim=2048))
model.add(Activation('relu'))
model.add(Dense(1))
model.compile(loss='mean_squared_error', optimizer='adam')
data_x = np.load(args.input_data_x)
data_y = np.load(args.input_data_y)
n_tr = 800
n_va = 150
n_te = 150
batch_size = 1
train_x = data_x[:n_tr]
valid_x = data_x[n_tr: n_tr + n_va]
test_x = data_x[n_tr + n_va: n_tr + n_va + n_te]
train_y = data_y[:n_tr]
valid_y = data_y[n_tr: n_tr + n_va]
test_y = data_y[n_tr + n_va: n_tr + n_va + n_te]
model.fit(train_x, train_y, batch_size=batch_size, nb_epoch=20,
validation_data=(valid_x, valid_y))
print(model.evaluate(test_x, test_y, batch_size=batch_size))
Train on 800 samples, validate on 150 samples
Epoch 1/20
800/800 [==============================] - 9s - loss: 3.6319 - val_loss: 3.4409
Epoch 2/20
800/800 [==============================] - 10s - loss: 2.6539 - val_loss: 1.3685
Epoch 3/20
800/800 [==============================] - 10s - loss: 1.3447 - val_loss: 1.2521
Epoch 4/20
800/800 [==============================] - 10s - loss: 1.1422 - val_loss: 1.1911
Epoch 5/20
800/800 [==============================] - 10s - loss: 1.0701 - val_loss: 1.6120
Epoch 6/20
800/800 [==============================] - 10s - loss: 1.0350 - val_loss: 1.0238
Epoch 7/20
800/800 [==============================] - 10s - loss: 0.9673 - val_loss: 1.0466
Epoch 8/20
800/800 [==============================] - 10s - loss: 0.9121 - val_loss: 1.0618
Epoch 9/20
800/800 [==============================] - 10s - loss: 0.8887 - val_loss: 0.9444
Epoch 10/20
800/800 [==============================] - 10s - loss: 0.7899 - val_loss: 1.1227
Epoch 11/20
800/800 [==============================] - 10s - loss: 0.7504 - val_loss: 1.1302
Epoch 12/20
800/800 [==============================] - 10s - loss: 0.7085 - val_loss: 0.8317
Epoch 13/20
800/800 [==============================] - 10s - loss: 0.7234 - val_loss: 0.9745
Epoch 14/20
800/800 [==============================] - 10s - loss: 0.6905 - val_loss: 0.8158
Epoch 15/20
800/800 [==============================] - 10s - loss: 0.6744 - val_loss: 0.8983
Epoch 16/20
800/800 [==============================] - 10s - loss: 0.6715 - val_loss: 0.9090
Epoch 17/20
800/800 [==============================] - 10s - loss: 0.6406 - val_loss: 1.6293
Epoch 18/20
800/800 [==============================] - 10s - loss: 0.6107 - val_loss: 0.8702
Epoch 19/20
800/800 [==============================] - 10s - loss: 0.6401 - val_loss: 0.8961
Epoch 20/20
800/800 [==============================] - 10s - loss: 0.5986 - val_loss: 0.8993
149/150 [============================>.] - ETA: 0s0.688796548107
- ECFP
model = Sequential()
model.add(Dense(output_dim=1024, input_dim=2048))
model.add(Activation('relu'))
model.add(Dense(1))
model.compile(loss='mean_squared_error', optimizer='adam')
Train on 800 samples, validate on 150 samples
Epoch 1/20
800/800 [==============================] - 3s - loss: 2.8745 - val_loss: 1.6488
Epoch 2/20
800/800 [==============================] - 3s - loss: 1.2366 - val_loss: 2.0804
Epoch 3/20
800/800 [==============================] - 3s - loss: 0.7095 - val_loss: 1.5705
Epoch 4/20
800/800 [==============================] - 3s - loss: 0.5783 - val_loss: 1.2935
Epoch 5/20
800/800 [==============================] - 3s - loss: 0.5029 - val_loss: 1.3650
Epoch 6/20
800/800 [==============================] - 3s - loss: 0.4190 - val_loss: 1.1915
Epoch 7/20
800/800 [==============================] - 3s - loss: 0.4148 - val_loss: 1.2134
Epoch 8/20
800/800 [==============================] - 3s - loss: 0.3698 - val_loss: 1.1759
Epoch 9/20
800/800 [==============================] - 3s - loss: 0.3246 - val_loss: 1.1978
Epoch 10/20
800/800 [==============================] - 3s - loss: 0.2402 - val_loss: 1.0753
Epoch 11/20
800/800 [==============================] - 3s - loss: 0.2474 - val_loss: 1.4807
Epoch 12/20
800/800 [==============================] - 3s - loss: 0.2079 - val_loss: 1.2086
Epoch 13/20
800/800 [==============================] - 3s - loss: 0.1916 - val_loss: 1.2955
Epoch 14/20
800/800 [==============================] - 3s - loss: 0.2605 - val_loss: 1.0776
Epoch 15/20
800/800 [==============================] - 3s - loss: 0.2289 - val_loss: 1.1403
Epoch 16/20
800/800 [==============================] - 3s - loss: 0.1803 - val_loss: 1.1627
Epoch 17/20
800/800 [==============================] - 3s - loss: 0.1709 - val_loss: 1.2304
Epoch 18/20
800/800 [==============================] - 3s - loss: 0.1724 - val_loss: 1.2065
Epoch 19/20
800/800 [==============================] - 3s - loss: 0.1638 - val_loss: 1.2290
Epoch 20/20
800/800 [==============================] - 3s - loss: 0.1488 - val_loss: 1.4525
32/150 [=====>........................] - ETA: 0s1.38019320567
テストデータに対して、NFPを用いた方がMean square Errorが低くなることがわかります
TODO
- batchへの対応
- 可視化機能
-
この†似ている†は何か定量的な定義があるわけではなく、「見た目なんとなく似ている」というような、わりとふんわりとした概念だと思ってください。 ↩