LightGBMのRのライブラリがCRANではなく、インストールから予測実施までがそれほどわかりやすくはなかったので、以下にまとめました。
セットアップ
Windows 10 で GPU(CUDA)を利用するLightGBM のR-packageをセットアップする
を参考にさせていただいたが、私の環境は
Windows10にCUDA9.0とPyCUDAをインストールしてテストが動きcupyインストールに苦労してChainerのMNISTチュートリアルでGPU使用で速度が約6倍となるのを確かめるまで
を実施済みだったので重複する箇所は端折った。
で、Windows 10 で GPU(CUDA)を利用するLightGBM のR-packageをセットアップするにある動作チェック
library(lightgbm)
## Loading required package: R6
data(agaricus.train, package='lightgbm')
train <- agaricus.train
dtrain <- lgb.Dataset(train$data, label=train$label)
## Loading required package: Matrix
params <- list(objective="regression", metric="l2")
model <- lgb.cv(params, dtrain, 10, nfold=5, min_data=1, learning_rate=1, early_stopping_rounds=10)
公式LightGBM R-package READMEも同じ。
は正常稼働したのだが、予測は?ってなって簡易チュートリアルが見つかりにくかったので調べた。
判別
2値
c.f. LightGBM/R-package/demo/basic_walkthrough.R
data(agaricus.train, package = "lightgbm")
data(agaricus.test, package = "lightgbm")
train <- agaricus.train
test <- agaricus.test
train, testともlist型。train$dataがmatrix型の説明変数。train$labelがnumeric型の目的変数。
なので、他のデータを使うときは0/1のnumeric型の目的変数と、matrix型の説明変数を用意すればよい。
2値でも0/1ではなく、1/2では駄目なようだ。
bst <- lightgbm(data = train$data,
label = train$label,
num_leaves = 4,
learning_rate = 1,
nrounds = 2,
objective = "binary")
でモデル構築。
dtrain <- lgb.Dataset(data = train$data,
label = train$label)
bst <- lightgbm(data = dtrain,
num_leaves = 4,
learning_rate = 1,
nrounds = 2,
objective = "binary")
と、dgb.Dtasetを作成して投入する方法もある。
pred <- predict(bst, test$data)
# plot(pred, test$label)
pred[pred >=0.5]<-1; pred[pred < 0.5]<-0
table(test$label, pred)
pred
0 1
0 813 22
1 13 763
で予測が可能。
マルチ
c.f. LightGBM/R-package/demo/multiclass.R
iris$Species<-as.numeric(as.factor(iris$Species)) -1
マルチクラスの場合も0, 1, 2, ... というような0始まりのnumerical型である必要がある。
library(lightgbm)
train <- as.matrix(iris[c(1:40, 51:90, 101:140), ])
test <- as.matrix(iris[c(41:50, 91:100, 141:150), ])
dtrain <- lgb.Dataset(data = train[, 1:4], label = train[, 5])
dtest <- lgb.Dataset.create.valid(dtrain, data = test[, 1:4], label = test[, 5])
valids <- list(test = dtest)
params <- list(objective = "multiclass", metric = "multi_error", num_class = 3)
model <- lgb.train(params,
dtrain,
100,
valids,
min_data = 1,
learning_rate = 1,
early_stopping_rounds = 10)
my_preds <- predict(model, test[, 1:4], reshape = T)
予測結果は、1行目のデータのclass1の確率、class2の確率、class3の確率、2行目のデータの……と出力されてしまうので、行毎クラス毎の行列で出す場合は reshape = T が必要。
table(test[, 5], max.col(my_preds))
1 2 3
0 9 1 0
1 0 10 0
2 0 0 10
確率の一番高い列を取る max.col() でどのクラスを予測したかが分かる。
回帰
下記は予測までのデモが見つからず、下記で試してみた。
library(lightgbm)
train <- as.matrix(iris[c(1:40, 51:90, 101:140), 1:4])
test <- as.matrix(iris[c(41:50, 91:100, 141:150), 1:4])
dtrain <- lgb.Dataset(data = train[, 1:3], label = train[, 4])
dtest <- lgb.Dataset.create.valid(dtrain, data = test[, 1:3], label = test[, 4])
params <- list(objective="regression", metric="l2")
model <- lgb.train(params, dtrain, 10, nfold=5, min_data=1, learning_rate=1)
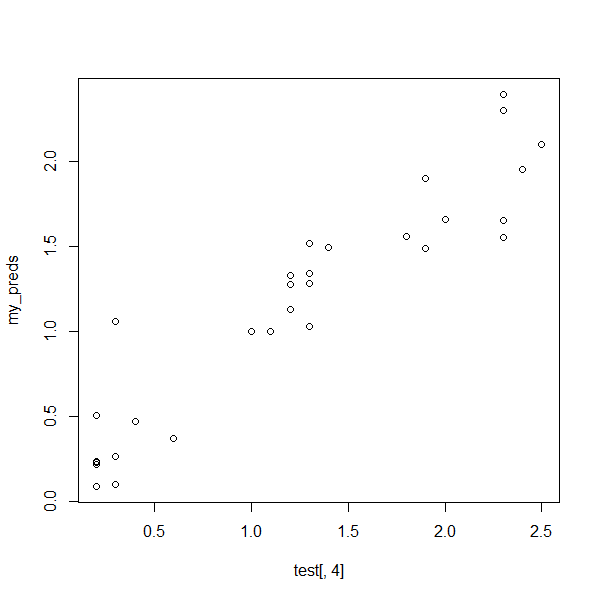
my_preds <- predict(model, test)
plot(test[, 4], my_preds)
予測出来ている。
参考
日本語
- 毎度分かりやすい、Rで学ぶ統計学
日本語だがPython
- Pythonだが丁寧な解説R化も楽そう
英語
- KAGGLERの解説
- RPubsにあった解説
で本記事が日本語でRかつ判別、回帰とも簡易チュートリアル