『Rによるデータサイエンス-データ解析の基礎から最新手法まで』
にある各実行をRのcaretパッケージで試します。
対象外
- caretは教師あり学習の判別と予測がメインなので、教師なし学習や生存分析、時系列モデルなどは対象外。
- 対象外:第1-6章、第11-12章、第17章
全体のメモ
- lmなど通常の関数で作成したモデルに対応するcaret使用のモデルは $finalModel となる。
- ただ一部項目は異なるようです。
- 例 lm
- cars.lm<-lm(dist ~ speed, data = cars)
- cars.caret.lm<-train(dist ~ speed, data = cars, method = 'lm')
- cars.lm に対応するのは cars.caret.lm$finalModel
- 予想結果等は一致する
- モデルの統計値等の項目が異なる場合があるので、図示や統計値抽出の場合は要注意
- 例 lm
第7章 線形回帰分析
7.2 線形単回帰分析 lm
library(caret)
# モデル作成
cars.lm<-train(dist ~ speed, data = cars, method = 'lm')
summary(cars.lm)
# 残差
residuals(cars.lm)
cars.lm$finalModel$residuals
# 回帰係数
round(coefficients(cars.lm$finalModel), 2)
round(cars.lm$finalModel$coefficients, 2)
# 予測
予測値<-predict(cars.lm)
残差<-residuals(cars.lm)
data.frame(cars, 予測値, 残差)
# グラフ
plot(cars)
abline(cars.lm$finalModel, lwd = 2)
par(oma = c(0, 0, 4, 0))
par(mfrow = c(2, 2))
plot(cars.lm$finalModel)
par(mfrow = c(1, 1))
par(oma = c(0, 0, 0, 0))
7.3 線形重回帰分析 lm
# モデル作成
air.lm<-train(Ozone ~ Solar.R + Wind + Temp, data = airquality, method = 'lm')
summary(air.lm)
# 回帰係数
round(coefficients(air.lm$finalModel), 2)
# 相互作用モデル
air.lm2<-train(Ozone ~ (Solar.R + Wind + Temp)^2, data = airquality, method = 'lm')
summary(air.lm2)
# 変数・モデルの選択
# air.lm3<-step(air.lm2$finalModel) はエラーとなる
第8章 非線形回帰分析
8.2 ロジスティック回帰、8.3 多項式回帰
- 非線形回帰nlsは対応なし。
8.4 一般化線形モデル
air.glm1<-train(Ozone ~ Solar.R + Wind + Temp, data = airquality, method = 'glm', family = 'gaussian')
air.glm2<-train(Ozone ~ Solar.R + Wind + Temp, data = airquality, method = 'glm', family = 'Gamma')
AIC(air.glm1$finalModel)
AIC(air.glm2$finalModel)
- 一般化線形モデルによるロジスティック回帰
年度<-c(1966:1984)
普及率<-c(0.003, 0.016, 0.054, 0.139, 0.263, 0.423, 0.611, 0.758, 0.859, 0.903, 0.937, 0.954, 0.978, 0.978, 0.982, 0.985, 0.989, 0.988, 0.992)
TV<-data.frame(年度, 普及率)
# モデル作成
fm5<-train(普及率 ~ 年度, data = TV, method = 'glm', family = 'binomial')
plot(年度, 普及率, type = 'l')
lines(年度, fitted(fm5), lty = 2, col = 'red', lwd = 2)
legend(locator(1), c('実測値', '予測値'), col = 1:2, lty = 1:2)
# 予測
predict(fm5$finalModel, type = 'response')
names(fm5); names(fm5$finalModel) # どちらもglmで作成したモデルとは少し異なる
予測結果に直接影響ないが、caretを用いているため、fm5もfm5$finalModelもglmから直接求めたモデルとは結果の項目が異なっている。
8.5 平滑化回帰と加法モデル
- 平滑化回帰と加法モデル
比較のための多項式モデルは下記で実行可能だが、平滑化回帰は対応なし。
df<-data.frame(x1, y1)
lmp2<-fitted(train(y1 ~ poly(x1, 2), data = df, method = 'lm'))
lines(x1, lmp2, lty = 4, col = 4, lwd = 2)
lmp5<-fitted(train(y1 ~ poly(x1, 5), data = df, method = 'lm'))
lines(x1, lmp5, lty = 2, col = 2, lwd = 2)
lmp8<-fitted(train(y1 ~ poly(x1, 8), data = df, method = 'lm'))
lines(x1, lmp8)
legend(locator(1), c('poly(x1, 2)', 'poly(x1, 5)', 'poly(x1, 8)'), col = c(4, 2, 1), lty = c(4, 2, 1), lwd = 2)
- 加法モデル
- 平滑化スプラインの対応がないので、gamの値がテキストと異なる。
airq2<-na.exclude(airquality[, 1:4])
airq.gam<-train(Ozone ~ Solar.R + Wind + Temp, data = airq2, method = 'gam')
AIC(airq.gam$finalModel)
airq.glm<-train(Ozone ~ Solar.R + Wind + Temp, data = airq2, method = 'glm')
AIC(airq.glm$finalModel)
第9章 線形判別分析
9.3 ケーススタディ(lda)
iris.lab<-c(rep('S', 50), rep('C', 50), rep('V', 50))
iris1<-data.frame(iris[, 1:4], Species = iris.lab)
even.n<-2 * (1:75) - 1
iris.train<-iris1[even.n, ]
iris.test<-iris1[-even.n, ]
Z.lda<-train(Species ~., data = iris.train, method = 'lda')
Z.lda$finalModel
apply(Z.lda$finalModel$means%*%Z.lda$finalModel$scaling, 2, mean)
table(iris.train[, 5], predict(Z.lda))
# plot(Z.lda$finalModel$fitted, dimen = 1) プロットできない
# plot(Z.lda$finalModel$fitted, dimen = 2) プロットできない
Z<-predict(Z.lda$finalModel, iris.train[, -5])
plot(Z$x, type = 'n', ylim = c(-8, 8))
text(Z$x, labels = iris.train$Species)
Y<-predict(Z.lda$finalModel, iris.test[, -5])
table(iris.test[, 5], Y$class)
plot(Y$x, type = 'n')
text(Y$x, labels = iris.test$Species)
図9.2に対応するものはプロット出来ない。ldaとcaret/ldaで出力形式が違うためで、探せば見つかると思うが分からず
図9.3も同様に直接はプロットできなかった。図9.3(a)はpredictを用いてほぼ同等の図がプロット出来た。
9.4 交差確認
iris.CV<-train(Species ~., data = iris, method = 'lda', trControl = trainControl(method = 'LOOCV'))
(lda.tab<-table(iris[, 5], predict(iris.CV)))
sum(lda.tab[row(lda.tab) == col(lda.tab)]) / sum(lda.tab)
sum(lda.tab[row(lda.tab) != col(lda.tab)]) / sum(lda.tab)
第10章 非線形判別分析
10.2 判別関数による判別分析 (qda)
前章で作成したiris.train, iris.testデータを用いる
Z.qda<-train(Species ~., data = iris.train, method = 'qda')
table(iris.train[, 5], predict(Z.qda))
Y.qda<-predict(Z.qda, iris.test[, -5])
table(iris.test[, 5], Y.qda)
10.3 距離による判別分析
- 対応なし
10.4 多数決による判別分析 (k最近傍法 k-Nearest Neighbor)
- K-Nearest-Neighbor & Tuning By Caretを参考にさせていただいた。
iris.knn<-train(Species ~., data = iris.train, method = 'knn', tuneGrid = expand.grid(k = 5))
iris.test.knn<-predict(iris.knn, iris.test[, -5])
table(iris.test.knn, iris.test[, 5])
# 図示
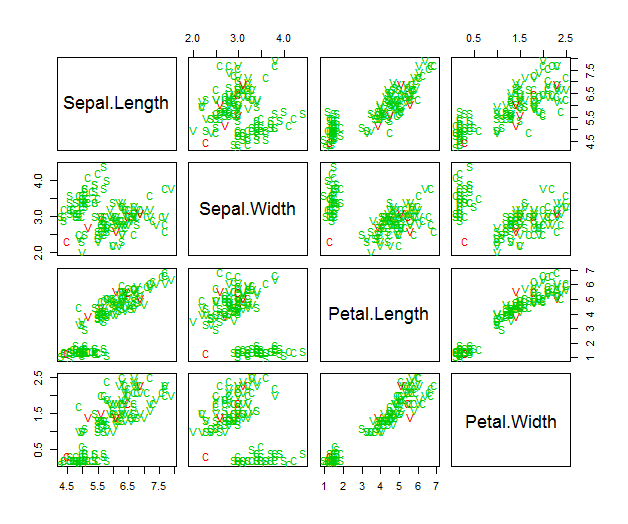
pairs(iris[, 1:4], pch = as.character(iris.test[ ,5]), col = c(3, 2)[(iris.test$Species != iris.test.knn) + 1])
# 交差確認
iris.cv<-train(Species ~., data = iris, method = 'knn', trControl = trainControl(method = 'LOOCV'), tuneGrid = expand.grid(k = 5))
table(iris[, 5], predict(iris.cv))
- 今回はk=5と指定されたが、kの最適値を求めるには下記のような感じ(1<=k<=10の範囲で探している)
knnGrid <- expand.grid(k = c(1:10))
iris.knn<-train(Species ~., data = iris.train, method = 'knn', tuneGrid = knnGrid)
iris.test.knn<-predict(iris.knn, iris.test[, -5])
table(iris.test.knn, iris.test[, 5])
10.5 ベイズ判定法
library(mlbench); data(Glass); G<-Glass[, c(1:5, 10)]
# NaiveBayesはデフォルトではカーネル法を用いないがcaretのnbはカーネル法を用いるのでオプションでFALSEにする
m1<-train(Type ~., data = G, method = 'nb', tuneGrid = data.frame(fL = 0, usekernel = F))
m1.p<-predict(m1)
tem1<-table(G$Type, m1.p)
1 - sum(diag(tem1)) / sum(tem1)
# カーネル法を用いる
m1<-train(Type ~., data = G, method = 'nb')
m1.p<-predict(m1)
tem1<-table(G$Type, m1.p)
1 - sum(diag(tem1)) / sum(tem1)
第13章 樹木モデル
分類木
set.seed(20)
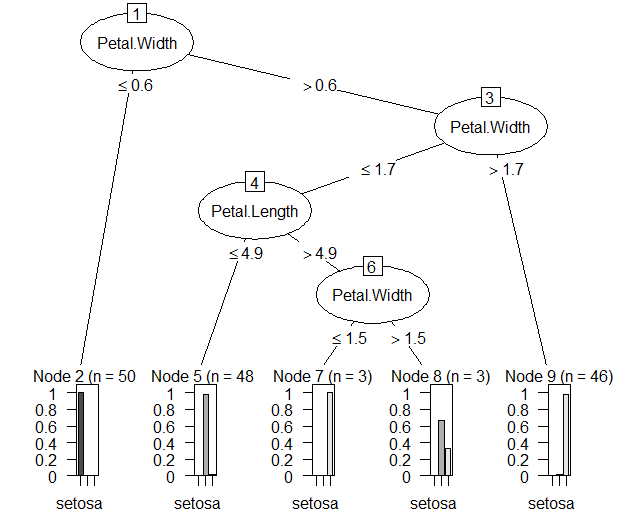
iris.rp<-train(Species ~., data = iris, method = 'rpart')
print(iris.rp$finalModel, digit = 1)
テキストの木と少し異なる。ただ、rpartで実行してもこの結果と同じ。テキストではmvpartを用いているので異なるか?ただ、mvpartはもうRでサポートされていない。
plot(iris.rp$finalModel, uniform = T, branch = 0.6, margin = 0.05)
text(iris.rp$finalModel, use.n = T, all = T)
木のプロットもmvpartを用いているテキストの図13.4とは異なるが、rpartのみの図と一致しているので、問題なし。
- 木の剪定
printcp(iris.rp$finalModel)
iris.rp1<-prune(iris.rp$finalModel, cp = 0.02)
plot(iris.rp1, uniform = T, branch = 0.6, margin = 0.05)
text(iris.rp1, use.n = T)
mvpart使用より最初から枝が少ないので、変化なし。
# plotcp(iris.rp$finalModel) は動かず、、、
caretならtuneGridで行くのだろう。例えばこんなか
(iris.rp.test<-train(Species ~., data = iris, method = 'rpart', tuneGrid = expand.grid(cp = seq(0.01, 0.50, 0.01))))
- 判別
even.n<-2 * (1:75) - 1
iris.train<-iris[even.n, ]
iris.test<-iris[-even.n, ]
set.seed(20)
iris.rp2<-train(Species ~., data = iris.train, method = 'rpart')
# plotcpは動かないので略
iris.rp3<-predict(iris.rp2, iris.test[, -5])
table(iris.test[, 5], iris.rp3)
回帰木
cars.rp<-train(dist ~ speed, data = cars, method = 'rpart')
cars.rp$finalModel
# printcpは略
cars.rp1<-prune(cars.rp$finalModel, cp = 0.044)
plot(cars.rp1, uniform = T, margin = 0.05)
text(cars.rp1, use.n = T)
多変量回帰木
- mvpartのデータセットspiderが必要であることと、目的変数が複数である回帰はあまり頻度が高くないと思われ、略
13.4 捕遣と注釈
- treeはなし
- J48
iris.j48<-train(Species ~., data = iris, method = 'J48')
iris.j48$finalModel
library(party)
plot(iris.j48$finalModel)
plot(iris.j48$finalModel, type = c('extended'))
テキストでは図13.14の(a), (b)が出力されるとなっているが、extendedの指定に関わらず同じ図で、テキストとも表示がやや異なる。RWeka/J48でもcaretのj48でも同じ図。
- ctree
cars.ctr<-train(dist ~., data = cars, method = 'ctree')
plot(cars.ctr$finalModel)
t.style<-node_hist(cars.ctr$finalModel, ymax = 0.06, xscale = c(0, 150), col = 'red', fill = hsv(0.6, 0.5, 1))
plot(cars.ctr$finalModel, terminal_panel = t.style)
第14章 ニューラルネットワーク
even.n<-2 * (1:75)
iris.train<-iris[even.n, ]
iris.test<-iris[-even.n, ]
iris.nnet<-train(Species ~., data = iris.train, method = 'nnet', tuneGrid = data.frame(size = 3, decay = 0.1))
iris.nnetp<-predict(iris.nnet, iris.test[, -5])
table(iris.test[, 5], iris.nnetp)
lvq
以下の通りに一応lvqは実施できるがlvqinitに対応するものや、lvq1, lvq2, lvq3, olvq1の別に対応するものはないようだ。
cd1<-train(Species ~., data = iris.train, method = 'lvq')
test.re<-predict(cd1, iris.test[, -5])
table(iris.test[, 5], test.re)
第15章 カーネル法とサポートベクターマシン
- kpcaはなし。
判別
- ksvm
library(kernlab); data(spam)
set.seed(50)
tr.num<-sample(4601, 2500)
spam.train<-spam[tr.num, ]
spam.test<-spam[-tr.num, ]
# set.seed(50) #テキストと同様にここで初期化をすると値がずれる。SVMの本質ではないのでどうでもよいがここでは初期化をなくした。
(spam.svm<-train(type ~., data = spam.train, method = 'svmRadial', trControl =
trainControl(method = 'CV', number = 3)))
Support Vector Machines with Radial Basis Function Kernel
2500 samples
57 predictor
2 classes: 'nonspam', 'spam'
No pre-processing
Resampling: Cross-Validated (3 fold)
Summary of sample sizes: 1667, 1666, 1667
Resampling results across tuning parameters:
C Accuracy Kappa
0.25 0.9044001 0.7960315
0.50 0.9152011 0.8200693
1.00 0.9215993 0.8345351
Tuning parameter 'sigma' was held constant at a value
of 0.02769061
Accuracy was used to select the optimal model using
the largest value.
The final values used for the model were sigma =
0.02769061 and C = 1.
spam.pre<-predict(spam.svm, spam.test[, -58])
(spam.tab<-table(spam.test[, 58], spam.pre))
spam.pre
nonspam spam
nonspam 1224 58
spam 95 724
1 - sum(diag(spam.tab)) / sum(spam.tab)
0.07282247
テキストと値が異なるがksvmでも同じ値となる。
y<-as.matrix(iris[51:150, 5])
iris1<-data.frame(iris[51:150, 3:4], y)
set.seed(0)
ir.ksvm<-train(y ~., data = iris1, method = 'svmRadial')
# plot(ir.ksvm, data = iris1[, 1:2]) #動かず、 ir.ksvm$finalModelだと作図されるが違う図
table(iris1$y, predict(ir.ksvm, iris1[, 1:2]))
versicolor virginica
versicolor 47 3
virginica 2 48
テキストと値が異なるがksvmでも同じ値となる。
回帰
x1<-seq(-10, 10, 0.1); set.seed(10)
y1<-50 * sin(x1) + x1^2 + 10 * rnorm(length(x1), 0, 1)
xy.svm<-train(y1 ~ x1, data = data.frame(x1, y1), method = 'svmRadial')
sy.pre<-predict(xy.svm, x1)
plot(x1, y1, type = 'l')
lines(x1, sy.pre, col = 'red', lty = 2)
legend(locator(1), c('実測値', '予測値'), lty = c(1, 2), col = c(1, 2))
epsilonを指定するメソッドがcaretのsvmにはないので略。
第16章 集団学習
テキストでは adabag パッケージを使用している。caretでは adabag パッケージを使用するものとして、AdaBag, AdaBoost.M1 があり、前者はバギングとブースティングの両方として紹介され、後者はブースティングとして紹介されている。ここでは前者をバギング、後者をブースティングとして示します。
バギング
library(mlbench)
data(BreastCancer)
even.n<-2 * (1:349)
BC.train<-BreastCancer[even.n, -1]
BC.test<-BreastCancer[-even.n, -1]
for(i in 1:9) {
BC.train[, i]<-as.integer(BC.train[, i])
BC.test[, i]<-as.integer(BC.test[, i])
}
# BC.train には欠損値があり、bagging ではそのまま扱えるようだが、下記ではそのまま扱えないので欠損値を埋める処理をする。
set.seed(20)
tmp.pre<-preProcess(BC.train, method = 'bagImpute')
BC.train<-predict(tmp.pre, BC.train)
set.seed(20)
BC.ba<-train(Class ~., data = BC.train, method = 'AdaBag')
# 同様にBC.test にも欠損値があり埋める処理をする。
set.seed(20)
tmp.pre<-preProcess(BC.test, method = 'bagImpute')
BC.test<-predict(tmp.pre, BC.test)
BC.bap<-predict(BC.ba, BC.test)
BC.bap<-data.frame(BC.bap); names(BC.bap)<-'class'
(ta.ba<-table(BC.test[, 10], BC.bap$class))
benign malignant
benign 216 11
malignant 3 120
テキストと値が異なってしまっている。欠損値の補完の問題だろうか、、、
1 - sum(diag(tb.ba)) / sum(tb.ba)
[1] 0.04
ブースティング
set.seed(20)
BC.ad<-train(Class ~., data = BC.train, method = 'AdaBoost.M1')
BC.adp<-predict(BC.ad, newdata = BC.test[, -10])
BC.adp<-data.frame(BC.adp); names(BC.adp)<-'class'
(tb.ad<-table(BC.adp$class, BC.test[, 10]))
benign malignant
benign 219 3
malignant 8 120
テキストと値が異なってしまっている。欠損値の補完の問題かもしれない。
1 - sum(diag(tb.ad)) / sum(tb.ad)
[1] 0.03142857
ランダムフォレスト
data(BreastCancer)
even.n<-2 * (1:349)
BC.train<-BreastCancer[even.n, -1]
BC.test<-BreastCancer[-even.n, -1]
for(i in 1:9) {
BC.train[, i]<-as.integer(BC.train[, i])
BC.test[, i]<-as.integer(BC.test[, i])
}
BC.train<-na.omit(BC.train)
set.seed(20)
BC.rf<-train(Class ~., data = BC.train, method = 'rf')
print(BC.rf$finalModel)
Call:
randomForest(x = x, y = y, mtry = param$mtry)
Type of random forest: classification
Number of trees: 500
No. of variables tried at each split: 2
OOB estimate of error rate: 3.85%
Confusion matrix:
benign malignant class.error
benign 214 7 0.03167421
malignant 6 111 0.05128205
テキストとは値が異なってしまっている。テキストとは異なり、 mtry = param$mtry が指定されてしまっているためか。パラメータの指定でどうにかなるかも知れないが、ここではスルーします。
summary(BC.rf$finalModel)
Length Class Mode
call 4 -none- call
type 1 -none- character
predicted 338 factor numeric
err.rate 1500 -none- numeric
confusion 6 -none- numeric
votes 676 matrix numeric
oob.times 338 -none- numeric
classes 2 -none- character
importance 9 -none- numeric
importanceSD 0 -none- NULL
localImportance 0 -none- NULL
proximity 0 -none- NULL
ntree 1 -none- numeric
mtry 1 -none- numeric
forest 14 -none- list
y 338 factor numeric
test 0 -none- NULL
inbag 0 -none- NULL
xNames 9 -none- character
problemType 1 -none- character
tuneValue 1 data.frame list
obsLevels 2 -none- character
caret使用時は、BC.rf$finalModel が randomForest のモデルに該当する以下も同様。
BC.rf$finalModel$type
[1] "classification"
plot(BC.rf$finalModel)
BC.rf$finalModel$importance
MeanDecreaseGini
Cl.thickness 10.3031669
Cell.size 31.0735166
Cell.shape 25.1238751
Marg.adhesion 10.2548693
Epith.c.size 13.5882463
Bare.nuclei 20.6794913
Bl.cromatin 20.7562703
Normal.nucleoli 19.0439173
Mitoses 0.9920097
varImpPlot(BC.rf$finalModel)
BC.rfp<-predict(BC.rf, BC.test[, -10])
(BC.rft<-table(BC.test[, 10], BC.rfp))
BC.rfp
benign malignant
benign 219 8
malignant 3 120
(1 - sum(diag(BC.rft)) / sum(BC.rft))
[1] 0.03142857