Rでパッケージ開発をする場合、他のパッケージとの依存関係の問題から、Devel版のRやDevel版の依存パッケージをインストールする必要がある。
この環境構築は結構めんどくさい。
XML, RCurlといったパッケージは、結構なパッケージが依存している割には、インストールするのにコツがいる。
また、linuxでRをインストールする場合、コンパイルされたRは配布されていないため、いちいちソース版のRをコンパイルする必要がある。
パッケージ開発者になると、大体一度作ったら終わりという事は無くて、長く面倒を見ていく必要がある。
なぜなら、今まで動いていたパッケージが、他の依存パッケージの仕様変更から、ある日いきなり動かなくなる可能性があるから。
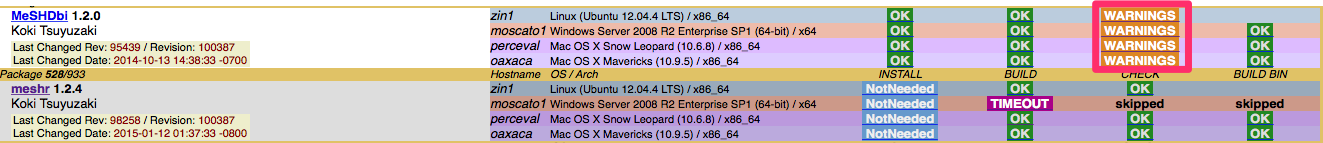
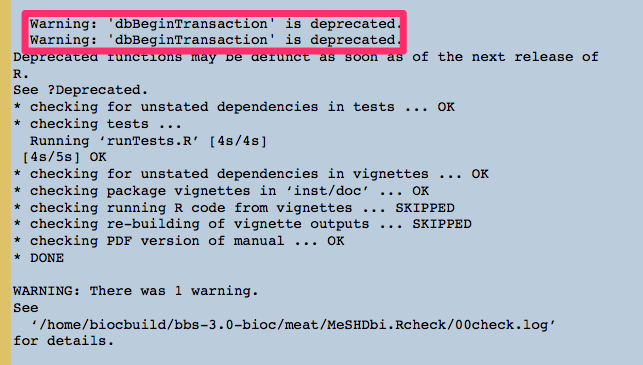
具体的に言うと、「Ha⚪︎ley-styleで開発するからw」みたいな無茶苦茶やる人がいたりして(参考)、「dbBeginTransactionやめてdbBeginって名前にしたからwww」(参考)みたいな過激な事をされると、今までdbBeginTransactionを使っていたパッケージは、ことごとく動かなるし、今私のパッケージは死んでます。
Bioconductorは全パッケージがきちんとインストールできるか毎日Daily Build Systemでチェックしているため、自分のパッケージにERROR/WARNINGSマークが付いていて、何も対応していないと、Core Teamから対応を迫られる(何もしないと多分そのうち消される)。
なので、環境構築は、パッケージが動かなくなる度に毎回する必要がある。
この手間を省くのに、Dockerをいじってたのだが(Bioconductorのパッケージ開発環境を作るためのDockerfile)、RやRのパッケージのリンク切れでビルドできなくなる可能性はあるので、たまにはメンテしないといけない。
最近はBioconductorが開発者のためにDocker containerで最新の環境を配布するようになったので( Docker containers for Bioconductor)、こっちを試しに使ってみた。
dockerの起動(Macの場合)
boot2docker start
devel_baseコンテナ内にbashで入る
docker run -ti bioconductor/devel_base bash
R
sessionInfo() # ちゃんとRがDevel版になっている
# > sessionInfo()
# R Under development (unstable) (2015-03-03 r67931)
# Platform: x86_64-unknown-linux-gnu (64-bit)
# Running under: Ubuntu 14.04.1 LTS
#
# locale:
# [1] LC_CTYPE=en_US.UTF-8 LC_NUMERIC=C
# [3] LC_TIME=en_US.UTF-8 LC_COLLATE=en_US.UTF-8
# [5] LC_MONETARY=en_US.UTF-8 LC_MESSAGES=en_US.UTF-8
# [7] LC_PAPER=en_US.UTF-8 LC_NAME=C
# [9] LC_ADDRESS=C LC_TELEPHONE=C
# [11] LC_MEASUREMENT=en_US.UTF-8 LC_IDENTIFICATION=C
#
# attached base packages:
# [1] stats graphics grDevices utils datasets methods base
#
# other attached packages:
# [1] MASS_7.3-39 BiocInstaller_1.17.5
library() # どんなパッケージがあるか確認
Packages in library ‘/usr/local/lib/R/library’:
# base The R Base Package
# BiocInstaller Install/Update Bioconductor and CRAN Packages
# boot Bootstrap Functions (Originally by Angelo Canty
# for S)
# class Functions for Classification
# cluster Cluster Analysis Extended Rousseeuw et al.
# codetools Code Analysis Tools for R
# compiler The R Compiler Package
# datasets The R Datasets Package
# foreign Read Data Stored by Minitab, S, SAS, SPSS,
# Stata, Systat, Weka, dBase, ...
# graphics The R Graphics Package
# grDevices The R Graphics Devices and Support for Colours
# and Fonts
# grid The Grid Graphics Package
# KernSmooth Functions for Kernel Smoothing Supporting Wand
# & Jones (1995)
# lattice Lattice Graphics
# MASS Support Functions and Datasets for Venables and
# Ripley's MASS
# Matrix Sparse and Dense Matrix Classes and Methods
# methods Formal Methods and Classes
# mgcv Mixed GAM Computation Vehicle with GCV/AIC/REML
# Smoothness Estimation
# nlme Linear and Nonlinear Mixed Effects Models
# nnet Feed-Forward Neural Networks and Multinomial
# Log-Linear Models
# parallel Support for Parallel computation in R
# rpart Recursive Partitioning and Regression Trees
# spatial Functions for Kriging and Point Pattern
# Analysis
# splines Regression Spline Functions and Classes
# stats The R Stats Package
# stats4 Statistical Functions using S4 Classes
# survival Survival Analysis
# tcltk Tcl/Tk Interface
# tools Tools for Package Development
# utils The R Utils Package
# (END)
BiocInstallerが既にあるので、いきなりbiocLiteが使えるみたい。
他にも、microarray, proteomics, sequencingのような、データ解析に必要なパッケージがあらかじめ入っているパッケージも公開されているっぽい。
必ず使うパッケージを入れておきたい場合や、インストールの時間を省いたり、全く同じ環境を提供したい状況(大学の講義とか)に使うのによさそう。