single-cell RNA-Seqで次元圧縮したのちに、外れ値細胞があった場合、それら細胞のIDを取得する方法。
ここでは、100細胞のうち、91〜100番目の細胞が外れ値細胞だと想定。
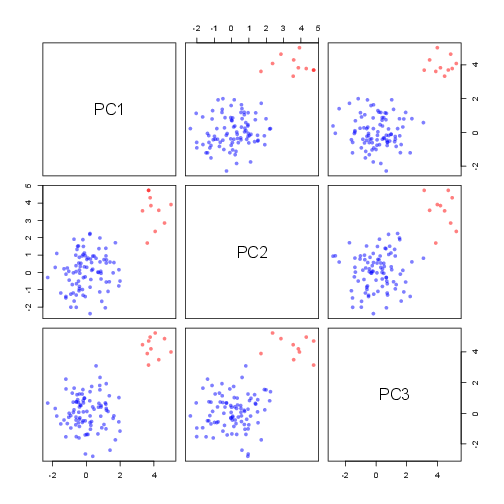
# PCAの結果だとする
# 91〜100番目に外れ値細胞が入っているけど、本番ではそれがわからないと仮定する
test.data <- data.frame(
PC1 = c(rnorm(90), rnorm(10, 4)),
PC2 = c(rnorm(90), rnorm(10, 4)),
PC3 = c(rnorm(90), rnorm(10, 4))
)
rownames(test.data) <- paste0("Cell", 1:100)
index.test.data <- c(rep(rgb(0,0,1,0.5), 90), rep(rgb(1,0,0,0.5), 10))
names(index.test.data) <- c(rep("Normal", 90), rep("Outlier", 10))
pairs(test.data, col=index.test.data, pch=16)
Rの標準関数(plot, pairsなど)では、点とラベルを同時に見やすく見せることが面倒くさそう。
そこで、幾つか拡張パッケージを試した。
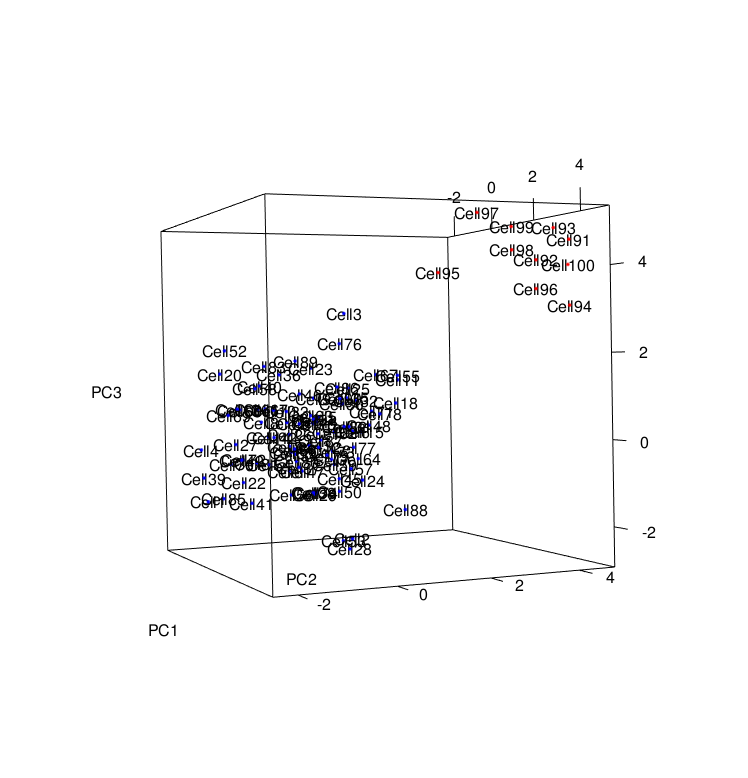
方法1 : rglパッケージ
library("rgl")
plot3d(test.data, col=index.test.data)
text3d(test.data, text=rownames(test.data))
この方法だと、あらかじめ細胞のIDをデータ点の上に重ねておく。
簡単だがデータ点が多くなるほど、IDが長くなるほど見づらくなるという弱点もある。
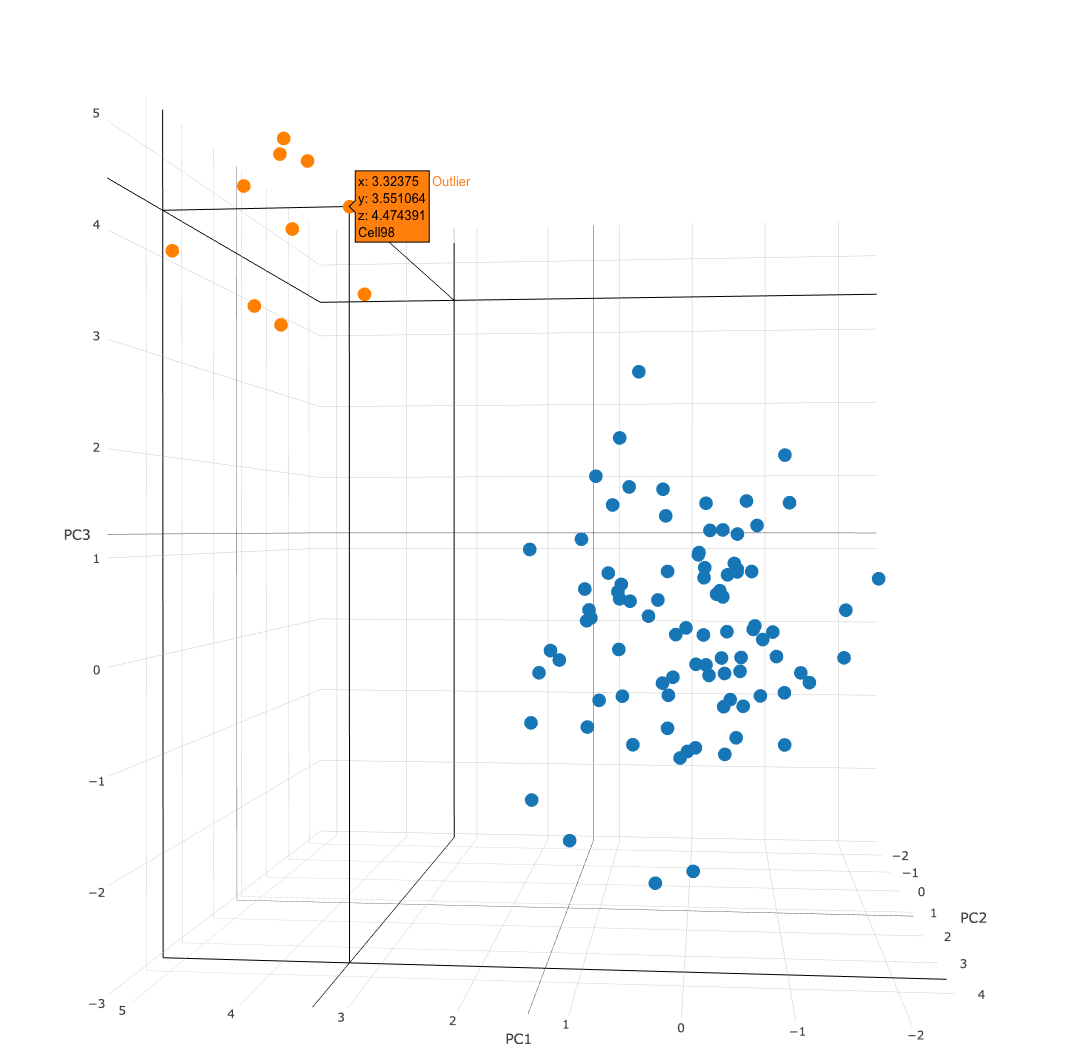
方法2 : plotlyパッケージ
library("plotly")
plot_ly(test.data, x = PC1, y = PC2, z = PC3,
type = "scatter3d", mode = "markers",
text = rownames(test.data),
group = names(index.test.data)
)
plotlyは一度plotlyサーバでアカウント作成、APIキー取得などの手順をふみ、サーバーサイドで描画するので、使いづらかったのだが、最近ローカルでも実行できるようになったみたいで、最近すごく使いやすい。
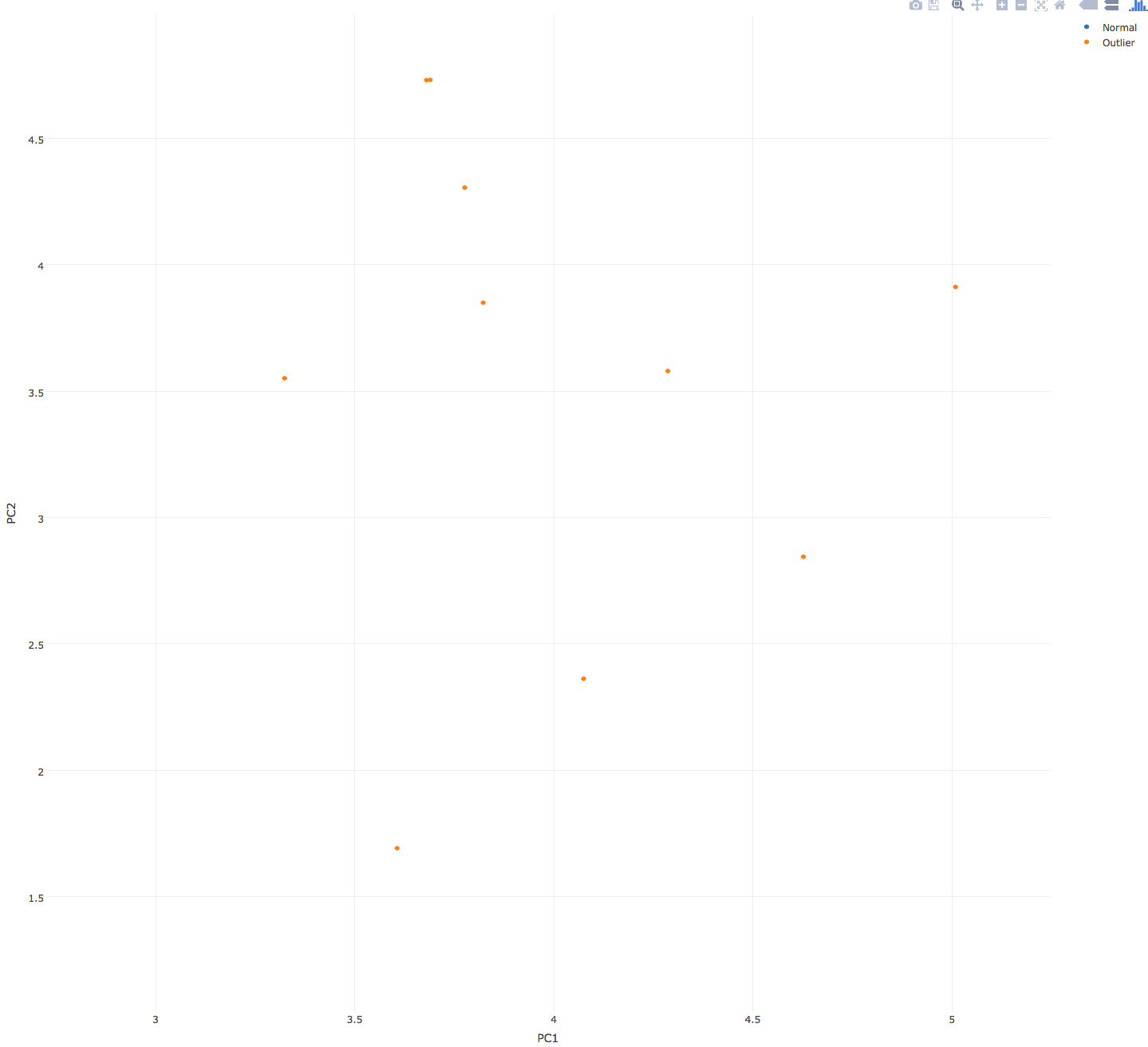
図のように、カーソルを合わせると、そのデータ点の細胞IDや座標、その他メタ情報が出てくる。
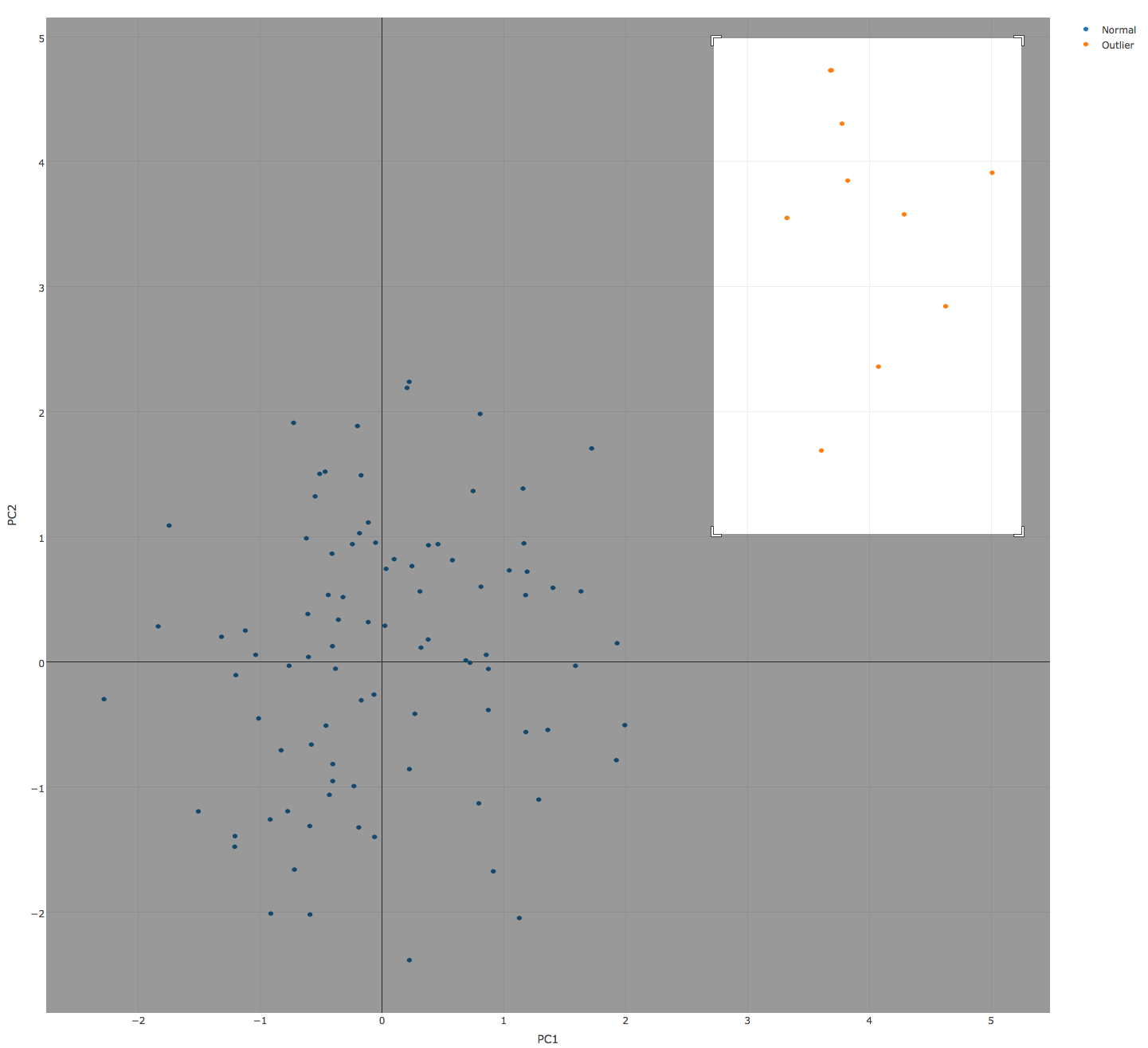
また、二次元の散布図だと、カーソルで選択したエリアだけズームするということもできる(三次元ではできない?)。
plot_ly(test.data[,1:2], x = PC1, y = PC2,
type = "scatter", mode = "markers",
text = rownames(test.data),
group = names(index.test.data)
)
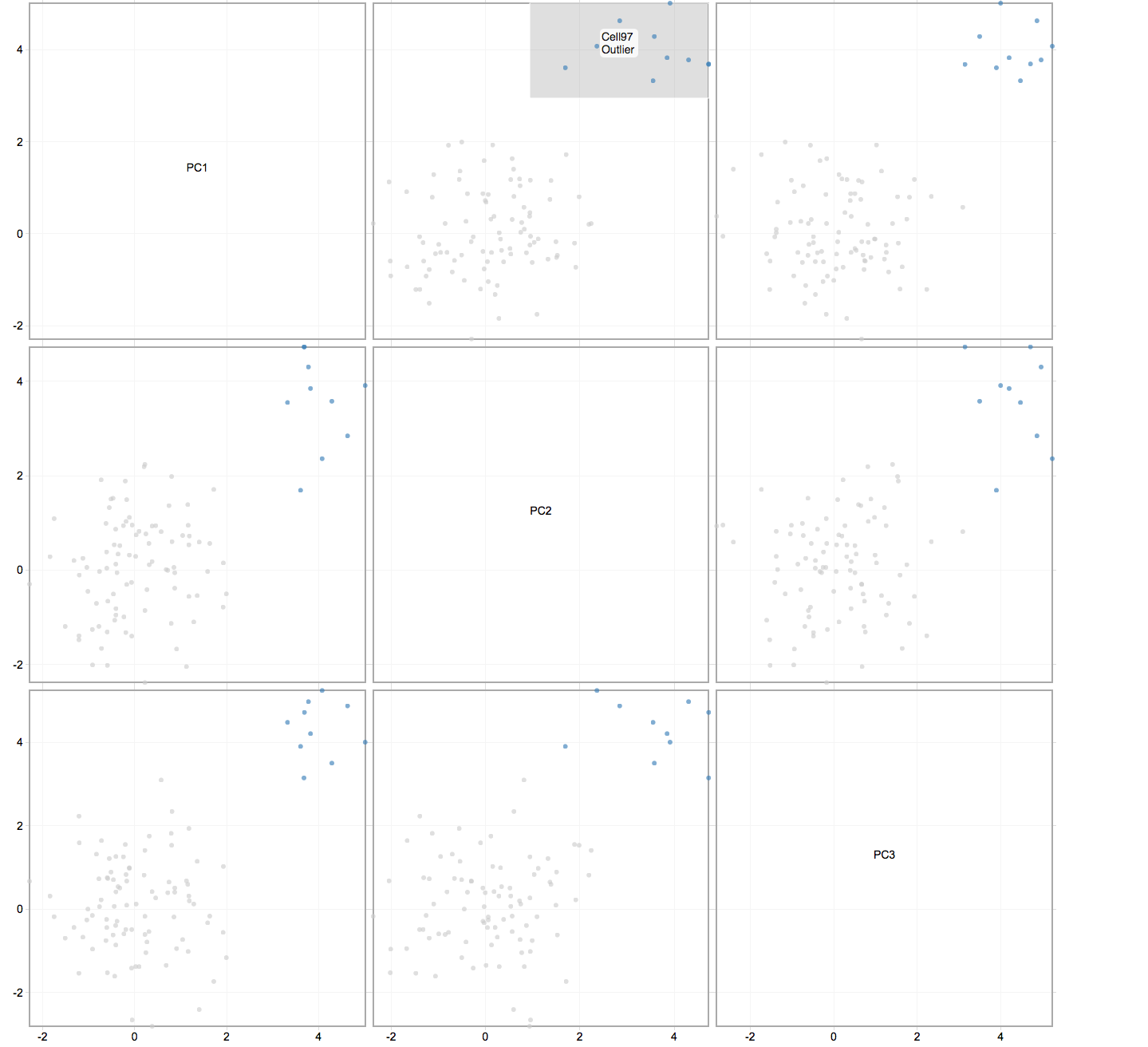
方法3 : pairsD3パッケージ
library("pairsD3")
pairsD3(test.data, group=names(index.test.data))
# Shinyでサーバーが立ち上がるが、止め方がわからない => Rを一回閉じることになる?
# shinypairs(test.data, group=names(index.test.data))
この方法は、pairs関数をそのままインタラクティブにした感じで、使いやすい。
shinypairs関数でShinyサーバーを立ち上げると、選択したデータのリストが取得できて便利だし、その場でパラメーターをいじることも可能になるのだが、この関数は一度起動すると止められないっぽい。