めったに起きることでも無いと思うが(結構起きる、コメント欄参照)、自分の開発しているパッケージscTensorがこけるパターンの一つとして、Gene NameとNCBI Gene ID(Entrez Gene ID)が一対一対応していない場合があった。
元を辿っていくと、内部で使っているbiomaRtが一つのNCBI Gene IDに対して、複数のGene Nameを返していた。
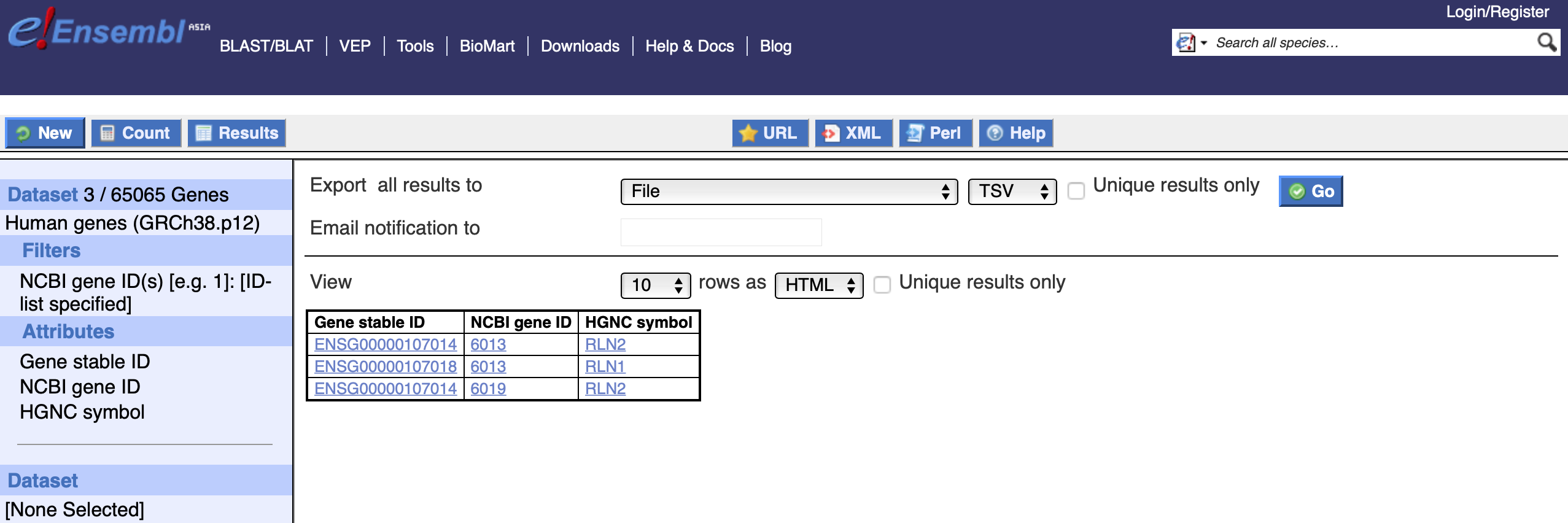
例えば、RLN1とRLN2は、NCBI側では、以下のように、6013、6019と別々のNCBI Gene IDが振られている。
- RLN1 : https://www.ncbi.nlm.nih.gov/gene/?term=6013
- RLN2 : https://www.ncbi.nlm.nih.gov/gene/?term=6019
しかし、biomaRtを使うと、
library("biomaRt")
ens <- useMart("ensembl", dataset="hsapiens_gene_ensembl",
host="asia.ensembl.org")
getBM(attributes=c('external_gene_name', 'entrezgene'),
filters = 'entrezgene',
values = "6013",
mart = ens)
# external_gene_name entrezgene
# 1 RLN2 6013
# 2 RLN1 6013
getBM(attributes=c('external_gene_name', 'entrezgene'),
filters = 'entrezgene',
values = "6019",
mart = ens)
# external_gene_name entrezgene
# 1 RLN2 6019
となるように、biomaRt的には、6013はRLN1でもRLN2でもあるみたいである。
そして、6019もまたRLN2である。
念の為、本家のWebサイトで手作業でやってみたが、同じ結果になった。
もう少し調べていくと、Ensembl側ではENSG00000107018のページのExternal referencesにNCBIのRLN1のリンクが貼られているが、
https://asia.ensembl.org/Homo_sapiens/Gene/Matches?db=core;g=ENSG00000107018;r=9:5334930-5339876
ENSG00000107014のページには、NCBIのRLN1とRLN2の両方のリンクが貼られていた。
https://asia.ensembl.org/Homo_sapiens/Gene/Matches?db=core;g=ENSG00000107014;r=9:5299864-5304716
RLN1とRLN2はゲノム上では隣同士に位置しており、Human cDNAsのところで繋がっているものがあるため、fusionして発現する場合があるのかもしれない。
それがこのID変換のトラブルに何か関係がありそう。
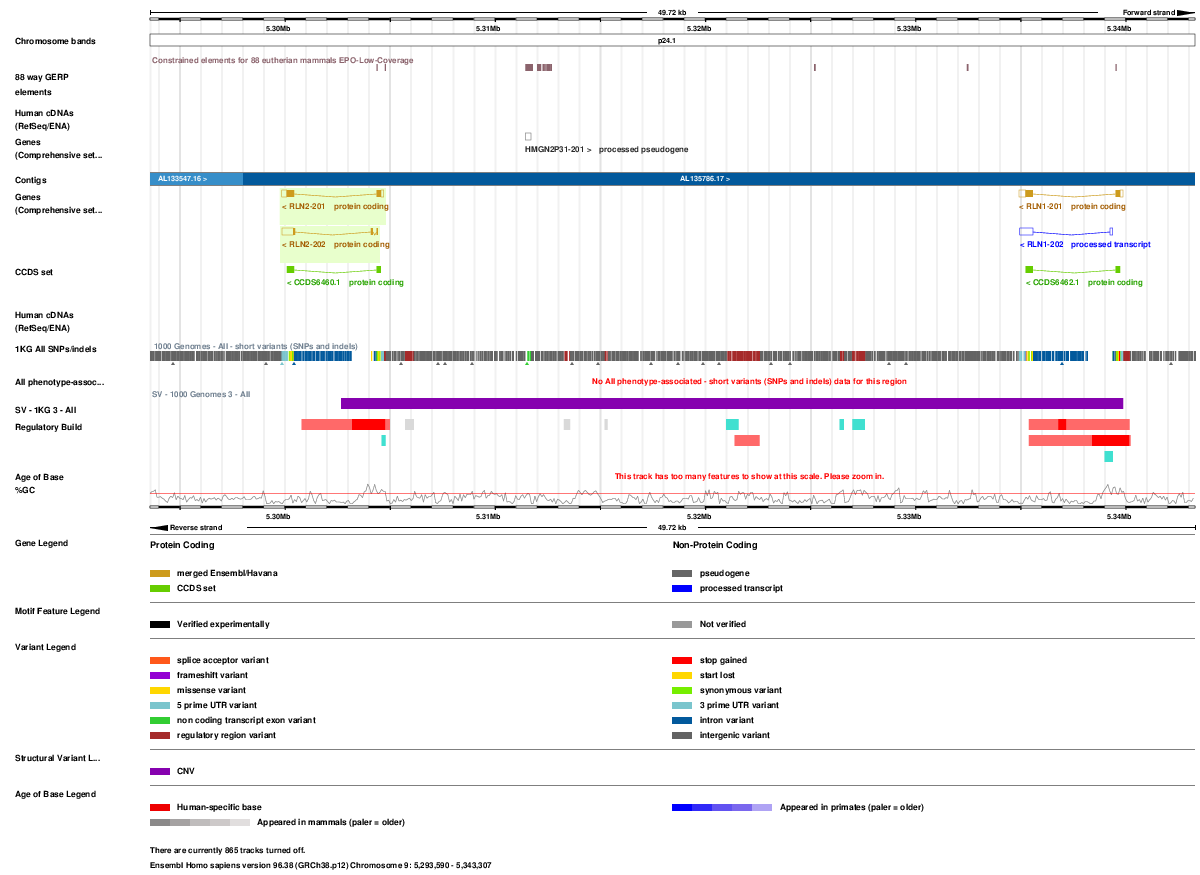
https://asia.ensembl.org/Homo_sapiens/Location/View?db=core;g=ENSG00000107014;r=9:5293590-5343307
ぐぐると、RLN1-RLN2 fusion exonについての言及が幾つかあった。
https://www.researchgate.net/publication/283261263_Identification_of_a_novel_fusion_transcript_between_human_relaxin-1_RLN1_and_human_relaxin-2_RLN2_in_prostate_cancer/figures?lo=1&utm_source=google&utm_medium=organic
https://www.ncbi.nlm.nih.gov/pubmed/26499396