目的
PURE system simulator1$^{,}$2$^{,}$3は、タンパク質合成反応に関わる241分子種が示す濃度変化を、実験結果と整合性がある形で観測できるシミュレーターである。そんな素晴らしいPURE system simulatorであるが、現在のところ、Matlab(有料)でしか動かない仕様である。Matlabのライセンスは高い。ちょっとシミュレーションに興味があるが、Matlabを買う程ではないなという人はたくさんいる(はず)。そんな人たちのために、MatlabではなくフリーソフトウェアのOctaveでPURE system simulatorを動かす方法をここに載せておきます。4
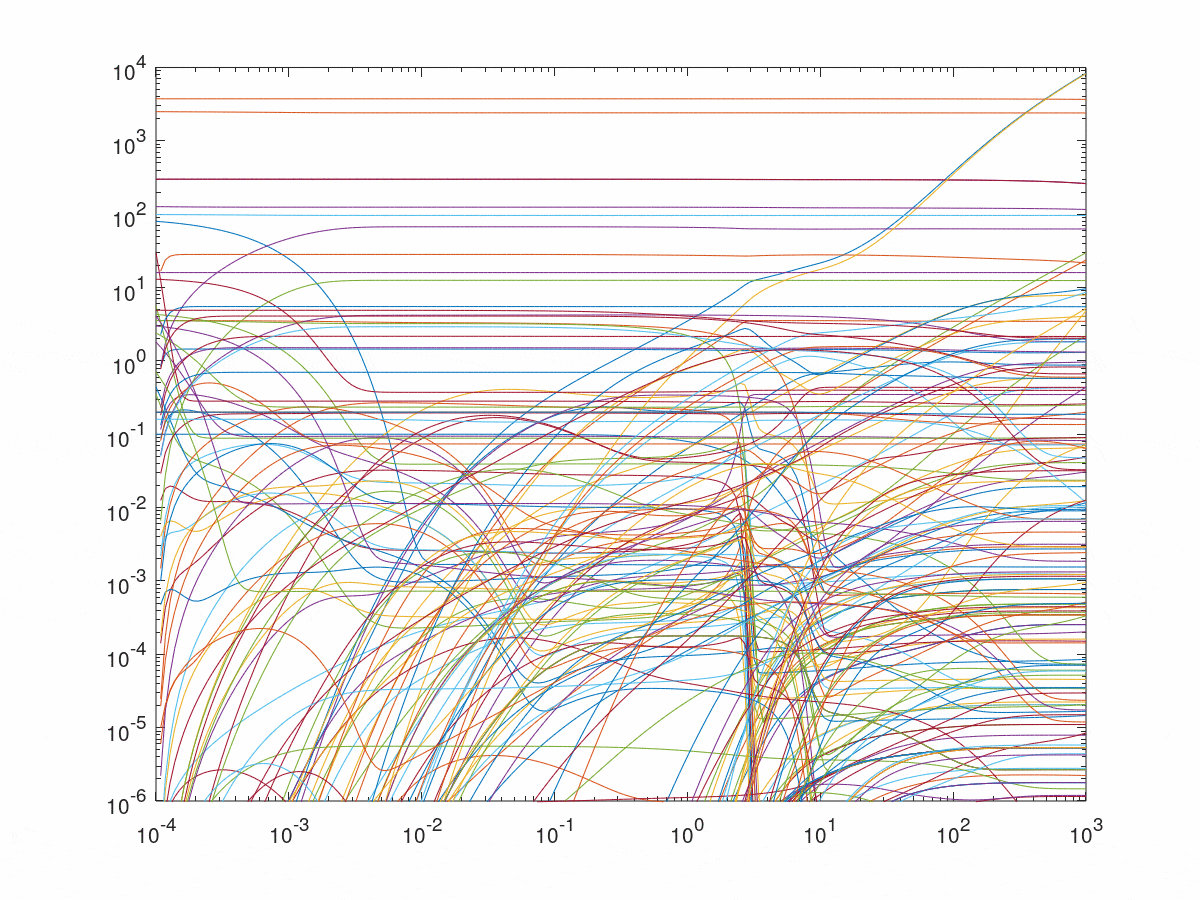
Octaveでのシミュレーション結果
Matsuura et al., (2017), PNASのFig. 2b2と同じ結果が得られました。
やり方
- Octaveをインストール
-
PURE system simulatorに置かれているサンプルプログラム
Simulate_fMGG_synthesis.zipを落としてきて適当なディレクトリに入れる。 -
fMGG_synthesis_Sample.m中の下記の行を変更 - (Option)
fMGG_synthesis_Sample.m中のグラフィックパラメータを変更。サンプルプログラムはプロットのデフォルト表示範囲が論文のFig. 2bと同じになっていない。論文と同じグラフを描きたい場合は、axis([10^(-4) 10^3 10^(-10) 10^5]);をaxis([10^(-4) 10^3 10^(-6) 10^4]);に変更する必要がある。 - 関数ファイル
fMGG_synthesis.m中の下記の行を変更
fMGG_synthesis_Sample.m内の変更部位
27,28行目 変更前
27: non_negative = 1:length(initial_values);
28: ode_opt = odeset('NonNegative', non_negative,'RelTol', 1e-3, 'AbsTol',1e-9);
27,28行目 変更後
lsode_options('relative tolerance', 1e-3);
lsode_options('absolute tolerance',1e-9);
lsode_options('integration method', 'stiff');
33行目 変更前
33: [t x] = ode15s(model_h, t,initial_values(:,1), ode_opt);
33行目 変更後
x = lsode(model_h, initial_values(:,3), t);
fMGG_synthesis.m内の変更部位
2674,2675行目 変更前
2674: time = varargin{1};
2675: state = varargin{2};
2674,2675行目 変更後
time = varargin{2};
state = varargin{1};
解説
Matlabではstiffな常微分方程式のソルバーとしてode15sが使われるが、Octaveではlsodeが使われる。やっていることは変わらないのだが、引数の渡し方や処理の仕方が少し違うのでMatlabからOctaveへ移植する際にはその変更が必要となる。まず、ode15sでは、optionを引数として渡すことができるのだが(28行目)、lsodeではそれができないので、lsode_optionsを使って事前に宣言することとなる。さらに、ode15s では(function, t, x0)とする引数の順番が、lsode では(function, x0, t)となる。また、返される値は、xの時間変化を示す配列のみである。2674,2675行目のvarargin は、fMGG_synthesisに渡した引数を個別に呼び出す関数である。varargin{1}となっていれば、関数fMGG_synthesisに渡した1番目の引数という意味である。その引数の順番が、ode15sとlsodeでは、異なるためにここの順番を入れ替える必要があると考えられえる
-
Reaction dynamics analysis of a reconstituted Escherichia coli protein translation system by computational modeling. Matsuura, T. et al., (2017), PNAS ↩
-
残念ながら、Matlabだと数秒で終わるシミュレーションがOctaveだとどうしても時間がかかります。大体20分ぐらい。仕様の問題なのか自分の変更方法が悪いのか誰か分かったら教えて欲しいです。 ↩