概要
データの可視化手法について、plt.plot(x, y)やplt.hist(data)ぐらいしか武器がないと、伝えられるものも伝えられないので、世の中的にはどんな可視化手法が使われているのか発掘し、それぞれどのように描かれているのか自分なりに考察していくシリーズ。Natureなどの記事から有用そうな論文を集めてきて、どうやったら描けるようになるのか具体的な手順を追っていく。なお、論文の内容にはあまり踏み込まず、図表だけを気にすることとする。図表から伝わるメッセージ性には着目する。ゆくゆくは自分でこういったプロットが描けるようになりたいという視点からなるべく具体的な手法を(推測で)埋めていく。なお、PythonやPowerpointを使用して描けることを目指す。
Natureから拾ってきた図表
「Organ aging signatures in the plasma proteome track health and disease」というタイトル。個々の臓器の加齢が人間の寿命や疾患にどのように影響するのかを調査した論文。具体的には、5,676人の成人を含む5つの独立したコホートで11の主要臓器の加齢を分析し、臓器の年齢を再現可能に推定。人口の約20%が一つの臓器で加速した加齢を示し、1.7%が多臓器加齢者であることを突き止めた。らしい。自分の臓器の年齢大丈夫かな、と大変気になる内容。
論文の要旨
この研究では、個々の臓器の加齢が人間の寿命や関連疾患にどのように影響するかを調査しました。研究チームは、特定の臓器から由来する人間の血漿タンパク質のレベルを使用して、生きている個人における臓器特有の加齢の違いを測定しました。5,676人の成人を含む5つの独立したコホートで11の主要臓器の加齢を分析し、臓器の年齢を再現可能に推定しました。人口の約20%が一つの臓器で加速した加齢を示し、1.7%が多臓器加齢者であることがわかりました。加速した臓器の加齢は20〜50%高い死亡リスクをもたらし、特定の臓器の疾患はその臓器の加速した加齢と関連しています。加速した心臓の加齢は心不全リスクを250%増加させ、加速した脳と血管の加齢は、プラズマpTau-181(現在最も優れた血液ベースのバイオマーカー)と同じくらい強く、独立してアルツハイマー病(AD)の進行を予測します。このモデルは、血管の石灰化、細胞外マトリックスの変化、およびシナプスタンパク質の剥離が早期認知低下と関連していることを示しています。この研究は、血漿プロテオミクスデータを使用した臓器の加齢を研究するための単純で解釈可能な方法を導入し、疾患と加齢の影響を予測します。
さて、こちらの論文に掲載されていた図表がこちら。左上のaから下のeまで順番に読み込んでいくらしい。
プロットの説明文(気になる方向け)
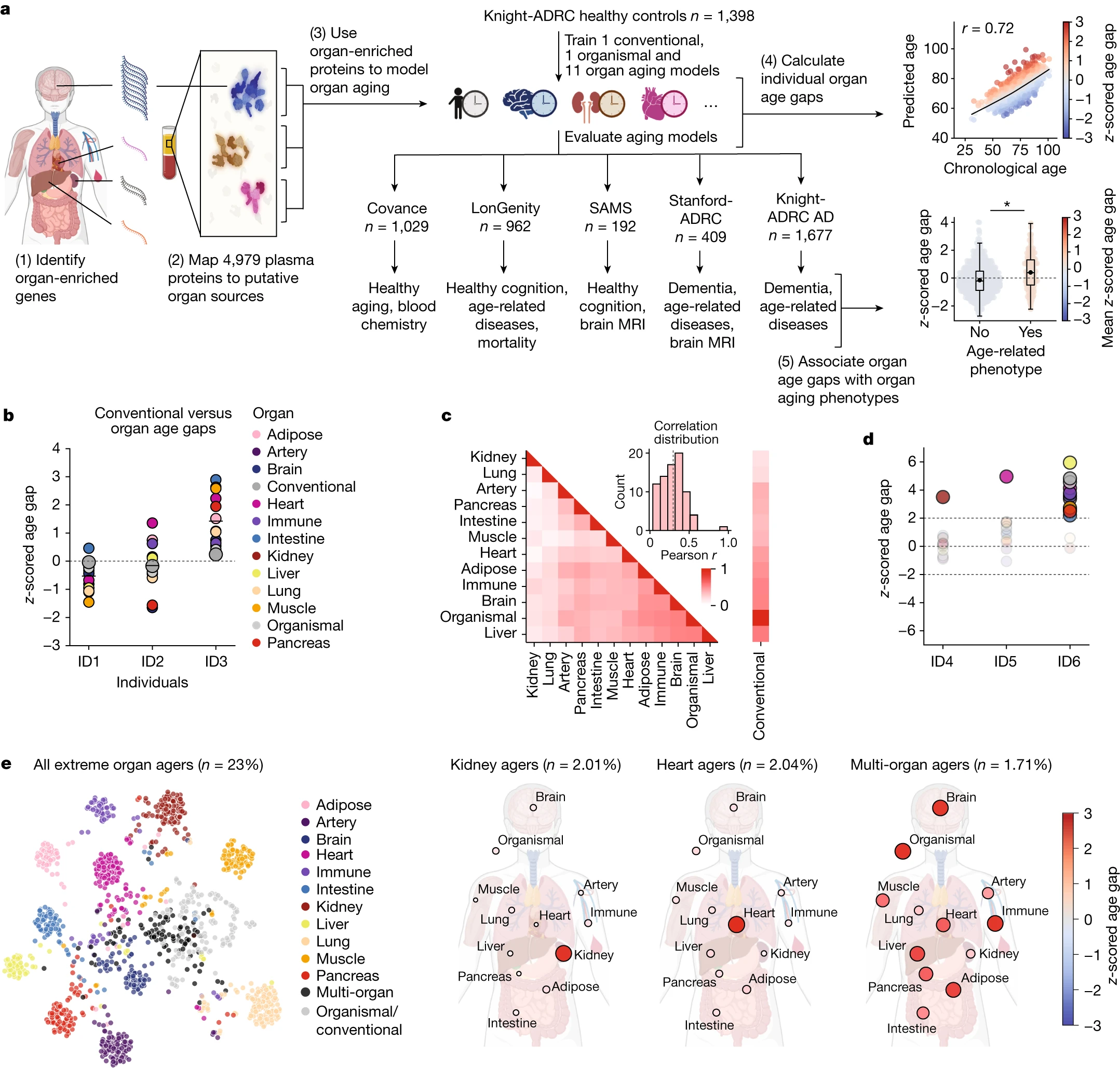
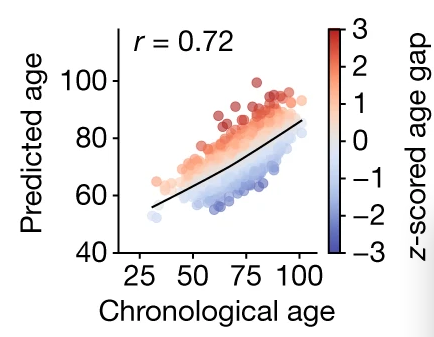
a, Study design to estimate organ-specific biological age. A gene was called organ-specific if its expression was four-fold higher in one organ compared to any other organ in GTEX bulk organ RNA-seq. This annotation was then mapped to the plasma proteome. Mutually exclusive organ-specific protein sets were used to train bagged LASSO chronological age predictors with data from 1,398 healthy individuals in the Knight-ADRC cohort. An ‘organismal’ model, which used the nonorgan-specific (organ shared) proteins, and a ‘conventional’ model, which used all proteins regardless of specificity, were also trained. Models were tested in four independent cohorts: Covance (n = 1,029), LonGenity (n = 962), SAMS (n = 192) and Stanford-ADRC (n = 420); models were also tested in the AD patients in the Knight-ADRC cohort (n = 1,677). To test the validity of organ aging models, the age gap was associated with multiple measures of health and disease. An example age prediction (predicted versus chronological age) and an example age gap versus phenotype association (age gap versus phenotype, standard boxplot) are shown.
臓器特異的な生物学的年齢を推定するための研究デザイン。GTEXのバルク臓器RNAシークエンスにおいて、ある臓器で他のどの臓器に比べて4倍以上の発現量を示す遺伝子を臓器特異的と定義しました。この注釈は血漿プロテオームにマッピングされました。Knight-ADRCコホートの1,398人の健康な個体のデータを用いて、相互に排他的な臓器特異的タンパク質セットでLASSO年齢予測器を訓練しました。非臓器特異的(共有臓器)タンパク質を使用した「全体生物学的」モデルと、特異性にかかわらずすべてのタンパク質を使用した「従来の」モデルも訓練されました。これらのモデルは、4つの独立したコホート(Covance(n = 1,029)、LonGenity(n = 962)、SAMS(n = 192)、Stanford-ADRC(n = 420))でテストされ、Knight-ADRCコホートのAD患者(n = 1,677)でもテストされました。臓器の加齢モデルの妥当性をテストするために、年齢差と健康・疾患の複数の指標との関連が調べられました。年齢予測(予測年齢と実年齢)の例と、年齢差と表現型との関連(年齢差と表現型、標準的なボックスプロット)の例が示されています。
a:研究デザインの説明
aの左〜真ん中の図は、Powerpointで解決。
右側のグラフは、技ありっぽい感じがする。読み解いてみる。
まず、実年齢と推定年齢の散布図になっている。それでもって、diff({実年齢}, {推定年齢})のz-scoreを求めてヒートマップ表示している。実年齢よりも加齢と判定された人は赤く表示されているらしい。注意しなければならないのは、このプロットはz-score基準であり、z-score=1は年齢推定誤差が1歳だったわけではなく、予測が1シグマ分外れているということを意味しているということだ。この図はmatplotlibで描けそう。
特筆すべきは、おそらく以下のような方法で相関係数を計算した上でグラフの上側にtextとして表示している点と、
# 相関係数を計算
correlation = np.corrcoef(chronological_age, predicted_age)[0, 1]
# 相関係数を表示
plt.text(0, 5, f'r = {correlation:.2f}', fontsize=12)
散布図のcの引数にz-scoreの数値が入力されているので、散布図の各プロットには濃淡が表現されている点。
# 散布図の描画
plt.figure(figsize=(6, 4))
scatter = plt.scatter(chronological_age, predicted_age, c=z_score_age_gap, cmap='coolwarm', alpha=0.6, edgecolors='w', marker='o')
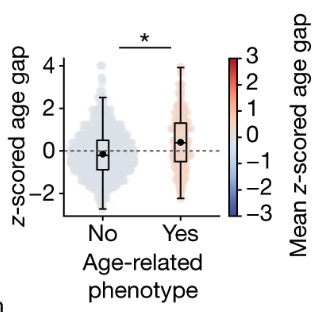
こちらのグラフは、上のage gapを「臓器の外見に加齢の特徴があるかどうか」というYes/Noで再度プロットしたものらしい。臓器外見的に特徴がある方が、痩せ型のプロットなので人数ボリュームは少ないらしい。また、box-plotの様子を見ると、加齢の外見的な特徴のある群(Yes)の方が、推定年齢を老いた方向に予測しがち(実年齢より老いて予測)される傾向があるということもわかるらしい。
臨床学的な意義は置いておいて、プロット的にはbiolin-plotとbox-plotを重ね合わせる表示は大変見やすい。その反面、Y軸に第二軸(右側)があり、情報詰め込み感があり、これはプロット分けた方が伝わりやすいのでは?と思った。自分の理解能力が足りていないだけなのかもしれないが。。。
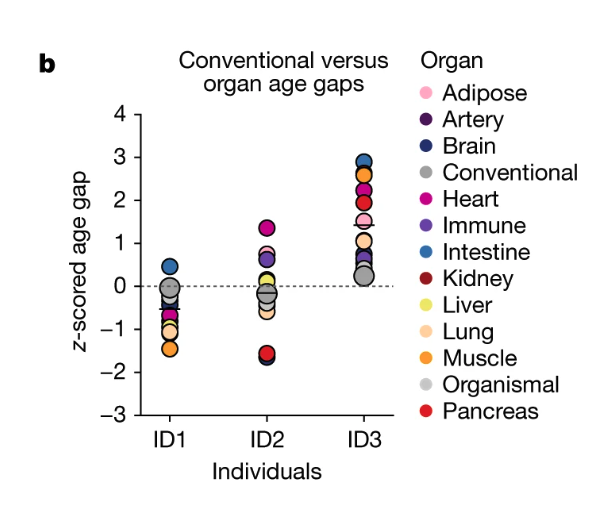
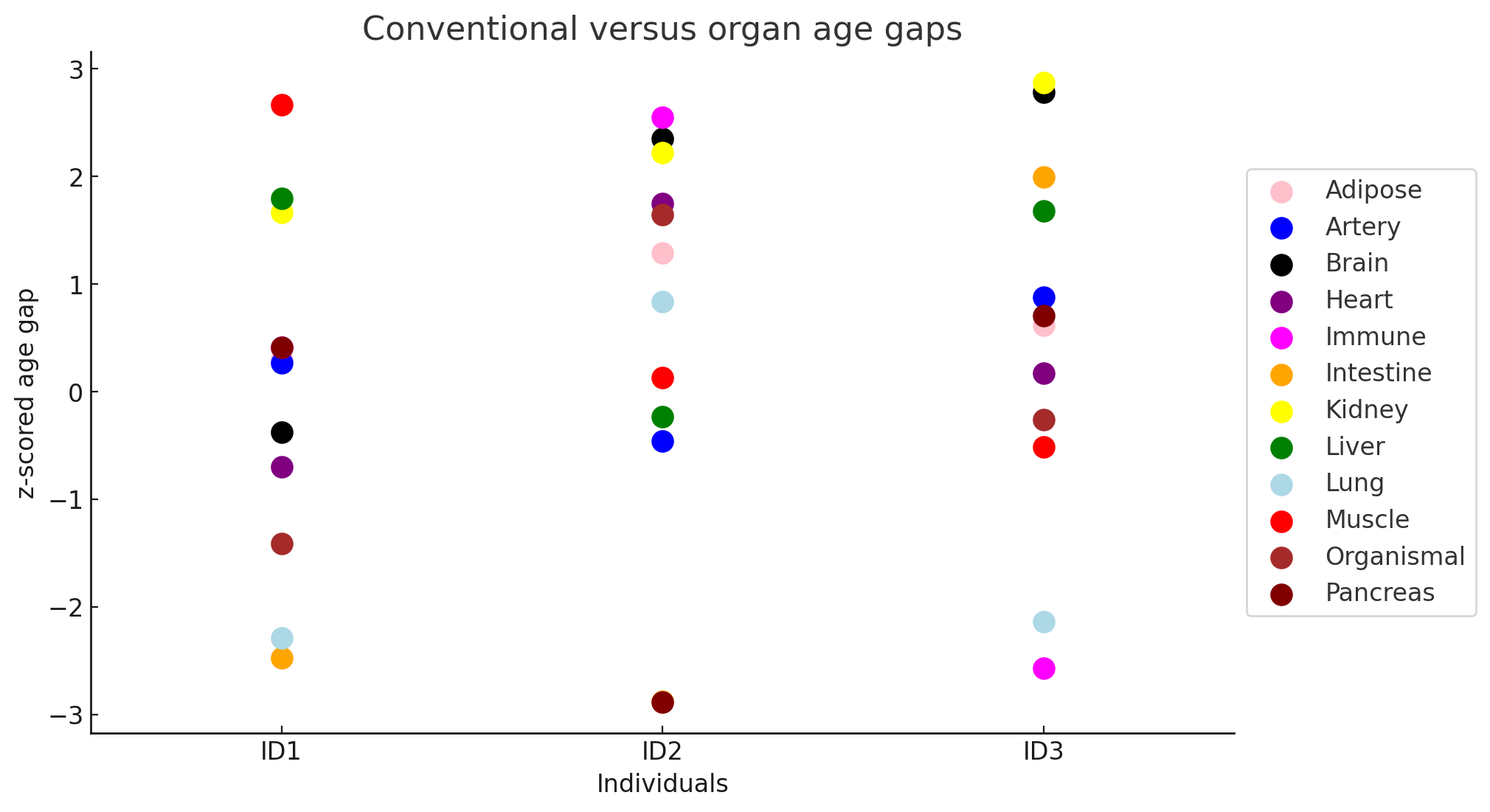
b: 臓器ごとの集計
代表的な3人の個々の臓器のage-gapの可視化。臨床的な意味は論文を読み進める必要がある。傾向としてID3の人は、実年齢よりも個々の臓器が老いている傾向が見て取れる。なぜだろうか。
プロットを勉強する意味で、chatgptに協力してもらいながら描いたプロットとスクリプト。それっぽくはなった。
scatterプロットを使って難しいことはせずにかけているので、プロット学的に新規性はあんまりなかった。
organs = {
'Adipose', 'Artery', 'Brain',
'Heart', 'Immune', 'Intestine',
'Kidney', 'Liver', 'Lung',
'Muscle', 'Organismal', 'Pancreas'
}
# ランダムなデータの生成
np.random.seed(0) # 再現性のためのシード設定
random_data = {organ: np.random.uniform(-3, 3, size=3) for organ in organs}
# pandas DataFrameの作成
df_random = pd.DataFrame(random_data, index=['ID1', 'ID2', 'ID3'])
# プロットの作成
fig, ax = plt.subplots()
# IDと臓器の年齢差をプロット
for organ in organs:
# 丸型のマーカーでプロット
ax.scatter(df_random.index, df_random[organ], label=organ, marker='o', s=100)
# 凡例をプロットの右側に表示
ax.legend(loc='center left', bbox_to_anchor=(1, 0.5))
# 補助線を削除
ax.grid(False)
# 軸のラベルを設定
ax.set_xlabel('Individuals')
ax.set_ylabel('z-scored age gap')
# タイトルを設定
ax.set_title('Conventional versus organ age gaps')
# プロットの表示
plt.show()
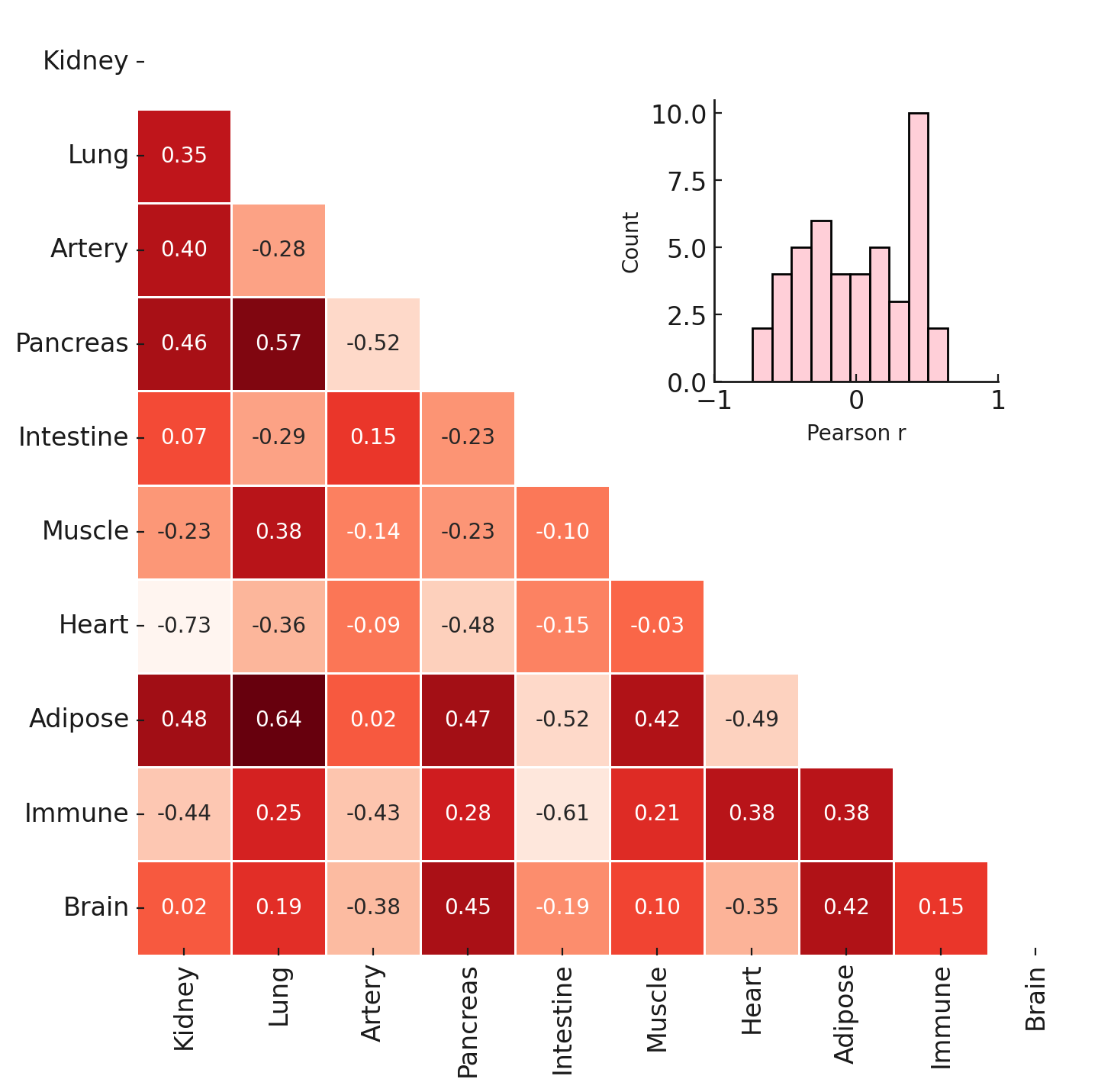
c: Headmap
Heatmapt自体はsns.heatmapを使ったものがよく見られうけども、折り紙のようにして非表示にするためには、np.triu(triangle-upperの略)をmaskとしてsns.heatmapに与えてあげると、右上の三角形が省かれたheatmapが描けるらしい。勉強になりました。
# 対角線より上のマスクを作成
mask = np.triu(np.ones_like(corr, dtype=bool))
# ヒートマップを描画
sns.heatmap(corr, annot=True, fmt=".2f", linewidths=.5, mask=mask, ax=ax, cbar=False, cmap="Reds")
また、これはあまり使う機会ないかもしれないけれど、from mpl_toolkits.axes_grid1.inset_locator import inset_axesを使うと、今回みたいにheatmapの余白に別のplotを描くことができるらしい。これは流石にマニアックすぎるしグラフに力入れすぎ感あるけれども、このライブラリ自体は知っておくと今後活きるかもしれない。
import matplotlib.pyplot as plt
import seaborn as sns
import pandas as pd
import numpy as np
from mpl_toolkits.axes_grid1.inset_locator import inset_axes
# 仮想データを生成(実際のデータがないため)
# 実際のデータがある場合は、それを読み込んで相関係数を計算します。
np.random.seed(0)
data = np.random.rand(10, 10)
organs = ['Kidney', 'Lung', 'Artery', 'Pancreas', 'Intestine', 'Muscle', 'Heart', 'Adipose', 'Immune', 'Brain']
df = pd.DataFrame(data, columns=organs, index=organs)
# 相関係数行列を計算
corr = df.corr()
# ヒートマップのレイアウトを設定
fig, ax = plt.subplots(figsize=(8, 8))
# ヒートマップを描画
# 対角線より上のマスクを作成
mask = np.triu(np.ones_like(corr, dtype=bool))
sns.heatmap(corr, annot=True, fmt=".2f", linewidths=.5, mask=mask, ax=ax, cbar=False, cmap="Reds")
# ヒートマップの上部にヒストグラムを追加
# widthとheightを30%に設定
ax_hist = inset_axes(ax, width="30%", height="30%", loc='upper right', borderpad=4)
# 相関係数の分布に基づいてヒストグラムを描画
# 補助線を解除し、binsを10に設定
sns.histplot(corr.values[np.triu_indices_from(corr, k=1)], bins=10, kde=False, color='pink', ax=ax_hist)
# ヒストグラムのX軸(相関係数の範囲)を設定
ax_hist.set_xlim(-1, 1)
# ヒストグラムの軸ラベルを設定
ax_hist.set_xlabel('Pearson r', fontsize=10) # X軸のラベル
ax_hist.set_ylabel('Count', fontsize=10) # Y軸のラベル
# ヒストグラムの補助線を解除
ax_hist.grid(False)
# ヒストグラムの軸ラベルを表示
ax_hist.xaxis.set_visible(True)
ax_hist.yaxis.set_visible(True)
# プロットの表示
plt.show()
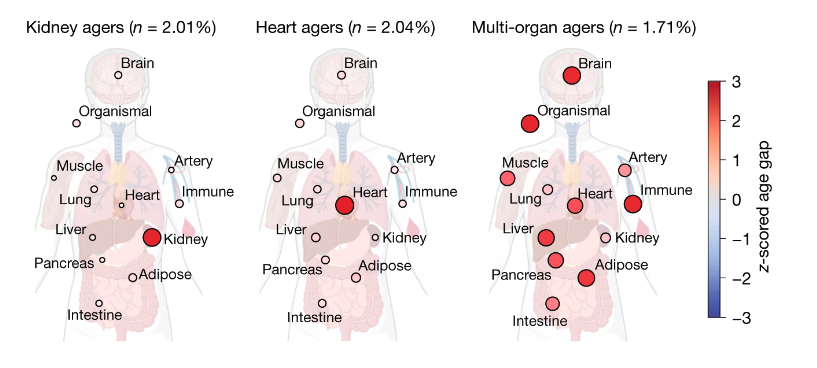
e: t-SNE
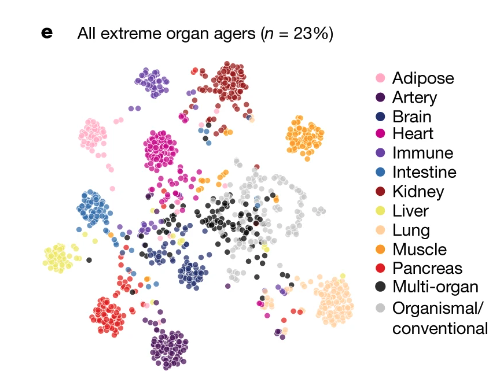
このプロットは、プロットの手法というよりは分析手法としてこのようなクラスタを生成する手法(t-SNE)を勉強する必要がありそう。今回は割愛。
こちらの図は、大変わかりやすい。イラストを背景として用意して、それぞれの座標を定義して、座標に沿ってz-scored age gapの数値をプロットしているようなのだけれども、この可視化はワンランク上どころか2ランク暗い上になりそうな気がするが、実務でも使えたら便利。
総括
やはり、データを可視化する技術が磨かれれば、伝えられるメッセージの量も増えるし、もっと上流の分析する力も磨かれるので、日頃から良いプロットを見て学ぶ機会を作った方が良いと感じました。また、便利なchatgptのおかげで、描きたいプロットを実現するための最初の一歩はかなり短縮されたので、大変ありがたい。