はじめに
一般公開されている、コロナウイルス(SARS-CoV-2)の生物情報の配列データ(biological sequence)の解読を試してみたので体験を共有します。
三回に渡って、生き物の遺伝情報である核酸 (DNA/RNA)の塩基配列やタンパク質を構成するアミノ酸配列の中身を探ってみます。
- Nextstrainでコロナウイルスのゲノム解析を行い解析結果をみてみる
- NCBIの生物情報配列データでコロナウイルスの突起蛋白のアミノ酸配列を解読してみる
- 同じく、コロナウイルスのRNAの塩基配列を解読してみる
一回目はオープンソースのゲノム解析ツールおよび解析結果の表示サイト、Nextstrainを取り上げます。
お断り: 筆者はプログラミングが専門で、医療生物は専門ではありません。できるかぎりウラをとったつもりですが、本稿の記載に知識や理解不足から誤りや誤解を生む箇所があるかもしれません。予めご了承ください。ご指摘をいただければさいわいです。
Nextstrain
Nextstrainはオープンソースで開発されている生物情報解析ツール(bioinformatics)です。
解析ツールを提供するほか、専門家が持ち寄るゲノムデータのオンラインデータベースであるGISAIDのデータをNextstrainを用いて解析した結果を見せるサイトも提供しています。
ツールを試す前に表示サイトを見てみました(2021/01/19時点)。
Genomic epidemiology of novel coronavirus - Global subsampling
https://nextstrain.org/ncov/global
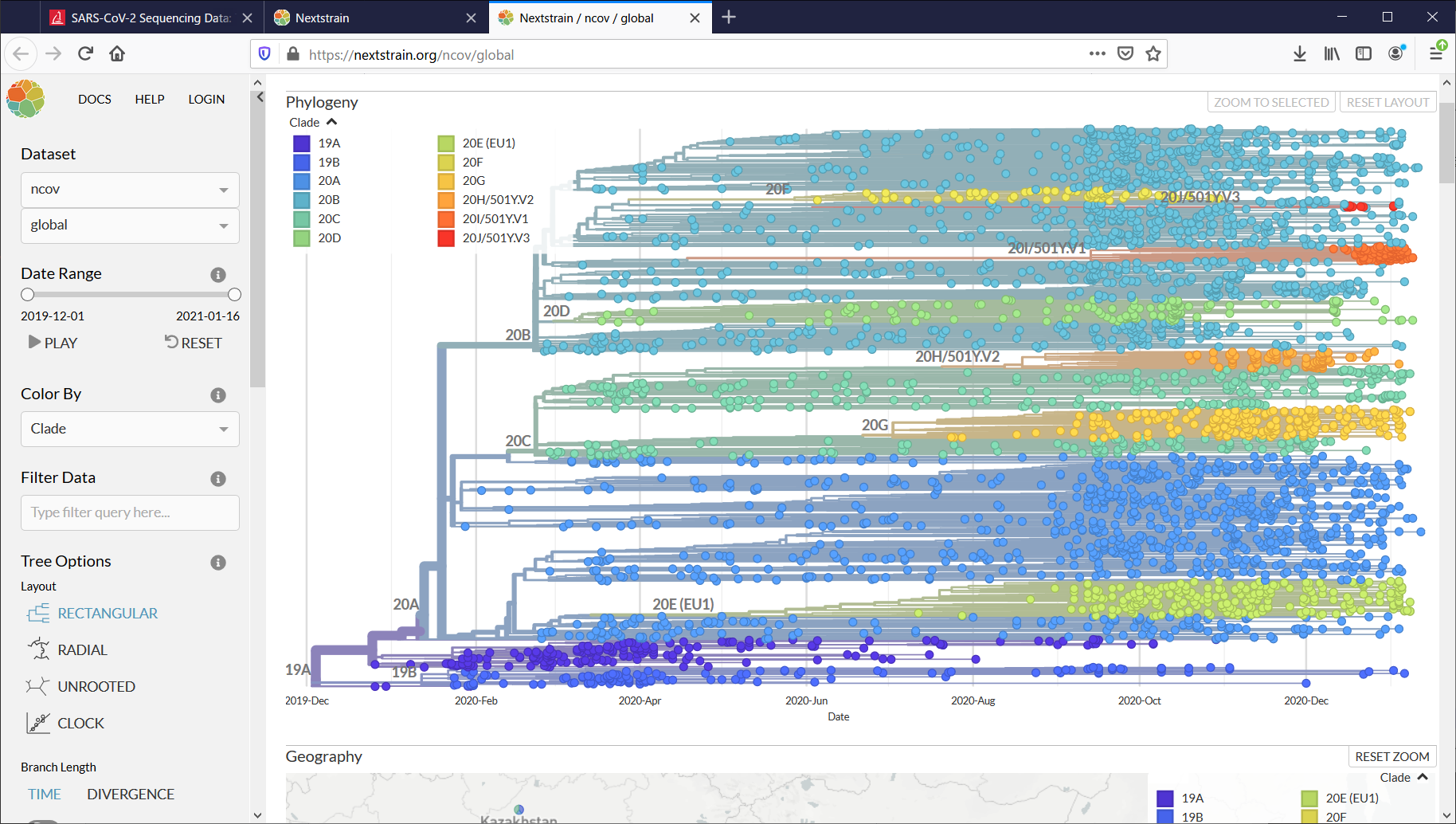
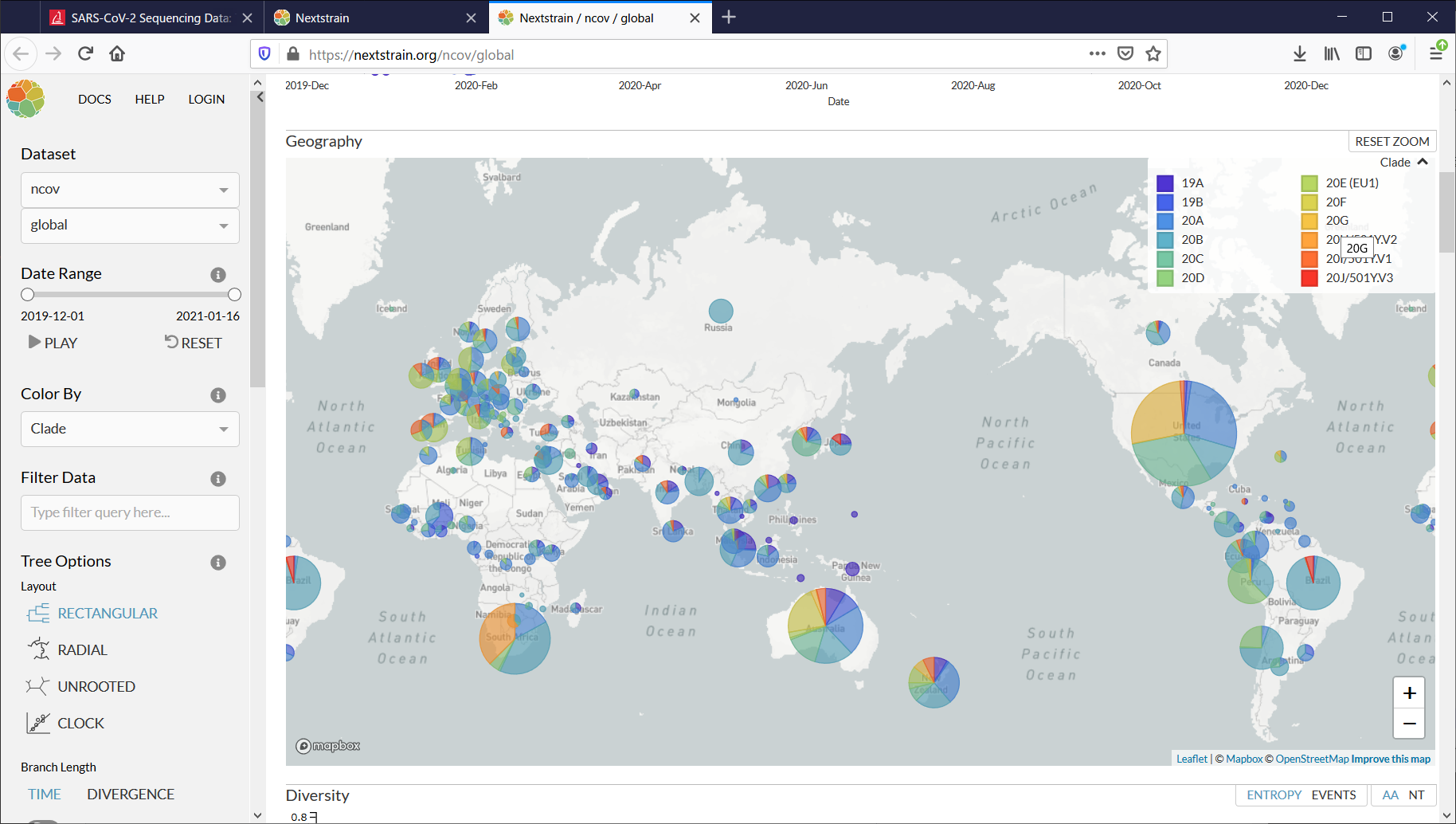
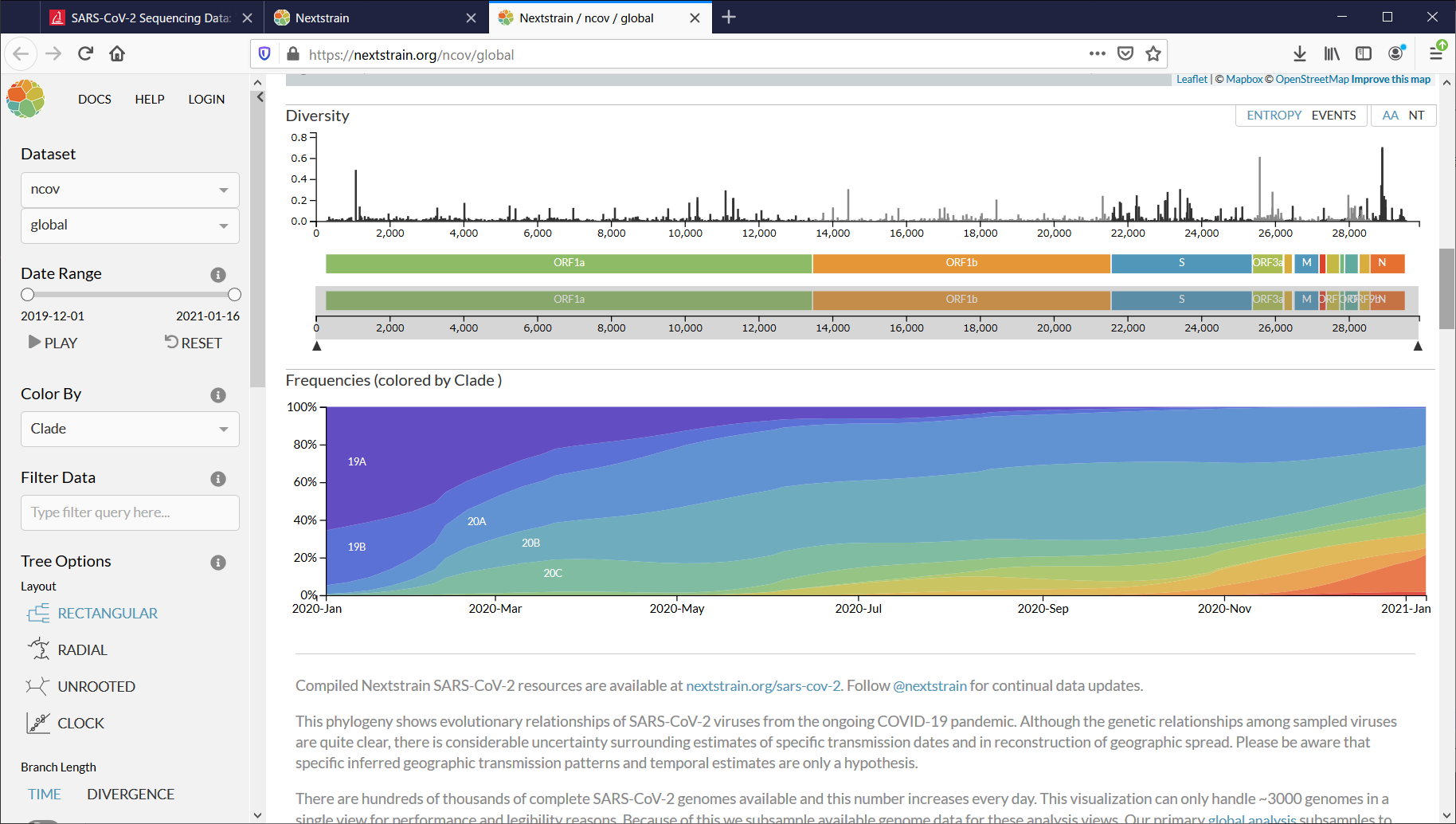
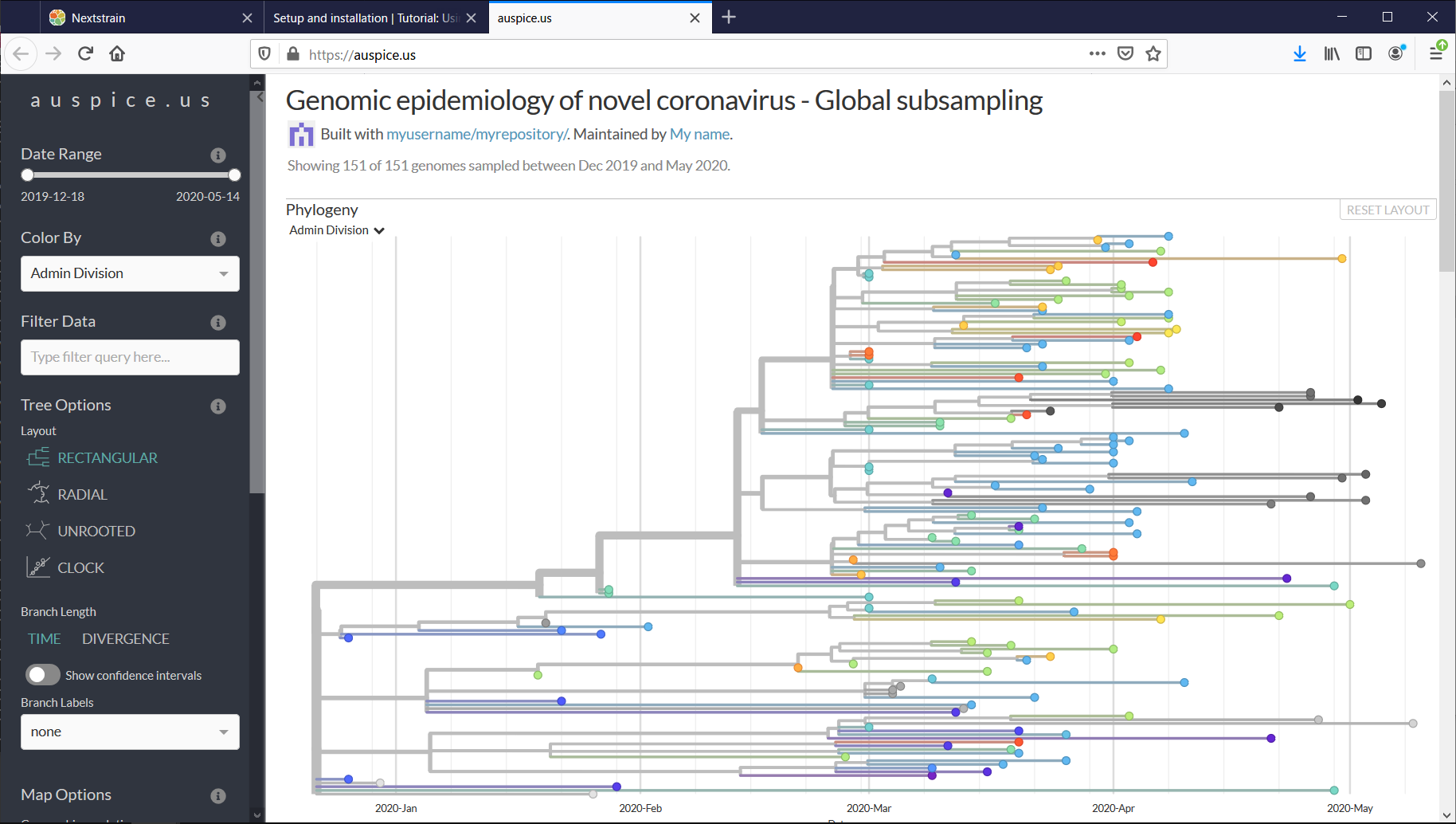
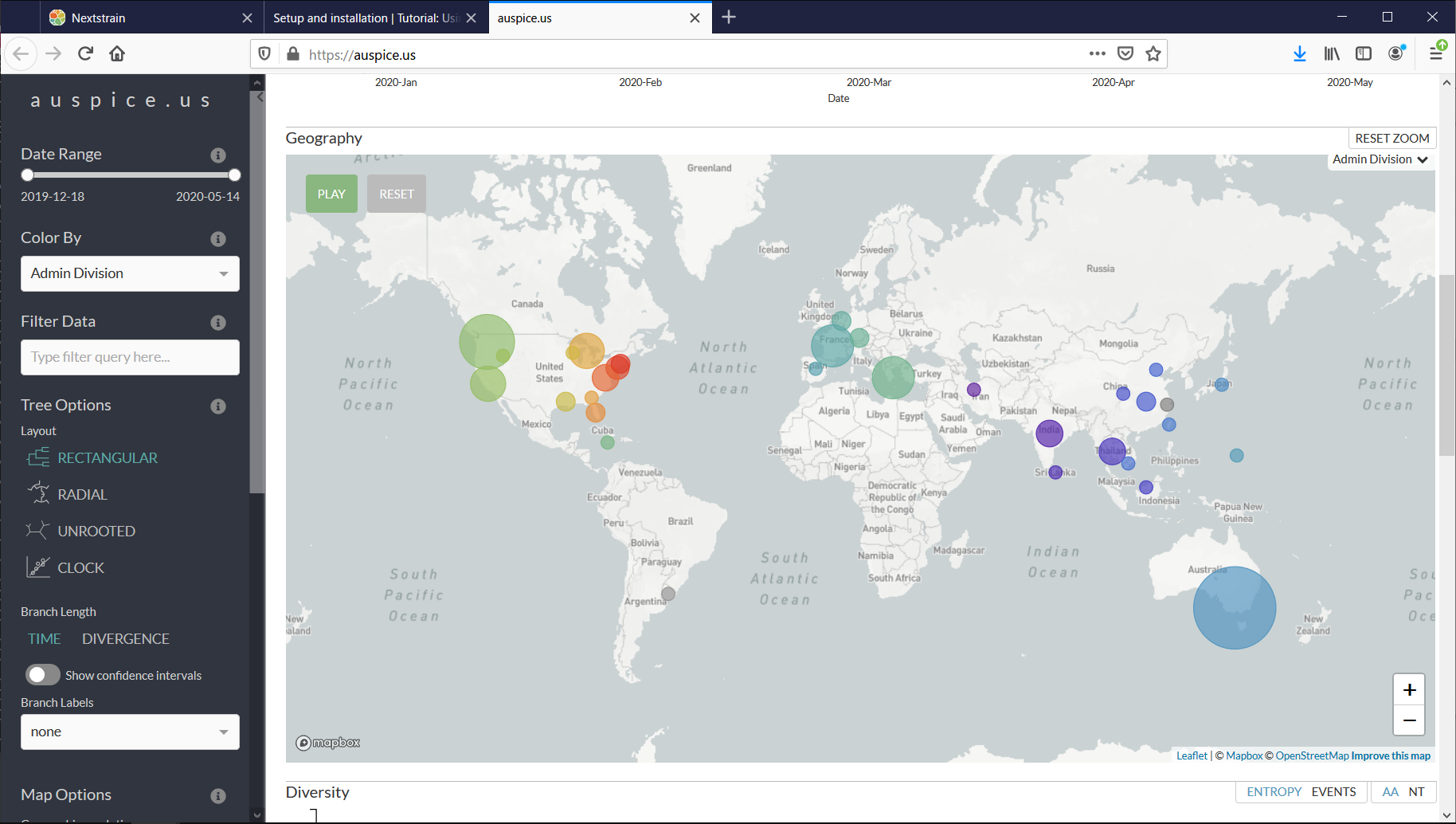
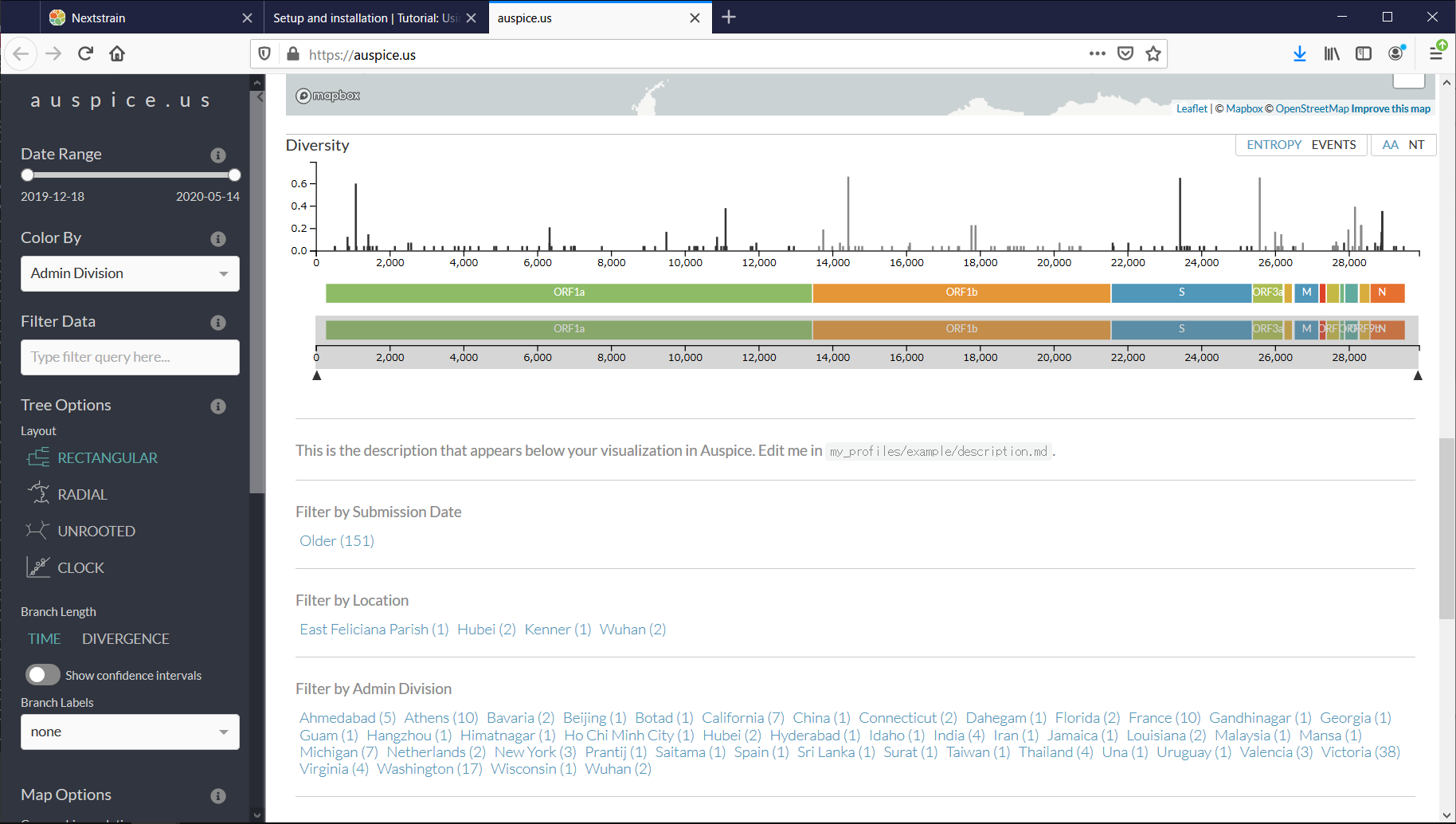
順にコロナウイルスの系統図、採取地を示す地図、変異の様子、がわかります。
[Phylogeny]
[Geography]
[Diversity]
Nextstrain解析ツール
Nextstrainの解析ツールはNextstrain CLIと呼ばれ、Python3で書かれています。
原理的にはLinuxのコマンドラインで使えるのですが、必要とするPythonのライブラリの依存関係があまりに複雑なため、Docker、Conda、AWS Batchなどの動作環境用にあらかじめ用意されたものをセットアップして使うことを勧める、と書かれています。
筆者はConda環境で動かせるチュートリアルを試してみました。
A Getting Started Guide to the Genomic Epidemiology of SARS-CoV-2
https://nextstrain.github.io/ncov/
Nextstrainのツールの使用は解析と表示の二段階に分かれます。
まず公開データや検査データを揃えてNextstrainの解析ツールを実行します。
こうして得られた解析結果は表示ツールでグラフィカルに表示できます。
表示ツールは自前でセットアップすることもできるようですが、Nextstrainの表示サイトを使うのが簡単です。
セットアップ
筆者がチュートリアルを試した環境です。
- Windows 10 PC (CPU: i5-7200, memory: 16GB)
- Windows Subsystem for Linux 2(WindowsでLinuxを動かすツール)
- Debian GNU/Linux 10 (buster) (Linuxのひとつ)
これにCondaをインストールしました。
CondaとはPythonの動作環境のひとつです。
Condaを使うには、Anacondaのミニ版のMinicondaをインストールするのが簡単、とのことで、そのとおりにしました。
Python3もなかったのでこれもインストールしました。
Conda環境にNextstrainをインストールします。
$ curl http://data.nextstrain.org/nextstrain.yml --compressed -o nextstrain.yml
$ conda env create -f nextstrain.yml
$ conda activate nextstrain
$ npm install --global auspice
続いてチュートリアルで使うデータをインストールします。
$ git clone https://github.com/nextstrain/ncov.git
Nextstrainの解析サイトでは専門家用のGISAIDのデータが使われていますが、チュートリアルでは一般公開されているNCBI(米国の国立衛生研究所などが運用している生物情報データベース)のデータを使うようになっています。
GISAID: https://www.gisaid.org/
NCBI: https://www.ncbi.nlm.nih.gov/sars-cov-2/
ゲノムデータ
チュートリアルデータには、解析対象のコロナウイルスのゲノム配列のサンプルデータが含まれています。
ファイルはexample_sequences.fastaです。
圧縮ファイルのexample_sequences.fasta.gzを解凍したものです。
$ pwd
/home/author/nextstrain/ncov/data
$ ls -l
total 13908
-rw-rw-r-- 1 author author 171914 Jan 18 15:06 example_metadata.tsv
-rw-rw-r-- 1 author author 12475184 Jan 19 13:27 example_sequences.fasta
-rw-rw-r-- 1 author author 1591123 Jan 18 15:06 example_sequences.fasta.gz
このファイルにはFASTAという形式で対象の全サンプルデータが収められています。
FASTA形式のファイルは、'>'で始まるヘッダ行(配列データの由来)の後ろに塩基配列またはアミノ酸配列が続きます。
データ中に改行のある場合とない場合とがあります。
example_sequences.fastaの場合は改行なしでウイルス一個の塩基配列が長い一行になっています。
配列の各要素は、塩基配列の場合は核酸(DNA/RNA)の四つの塩基コード(ACGT/U)、アミノ酸配列の場合はタンパク質を構成するアミノ酸コード(AsparagineならN、TyrosineならYなど)です。
$ cat example_sequences.fasta
>Wuhan/Hu-1/2019
ATTAAAGGTTTATACCTTCCCAGGTAACAAACCAACCAACTTTCGATCTCTTGTAGATCTGTTCTCTAAACGAACTTTAAAATCTGTGTGGCTGTCACTCGGCTGCATGCTTAGTGCACTCACGCAGTATAATTAATAACTAATTACTGT...
ごらんのとおりテキストであることがわかります。
最初のデータは武漢で取れたウイルスの塩基配列のようです。
残りのデータのヘッダ部分もみてみます。
$ grep '>' example_sequences.fasta
>Wuhan/Hu-1/2019
>Wuhan/WH01/2019
>Australia/VIC05/2020
>Australia/VIC1000/2020
...
>Wuhan/IPBCAMS-WH-03/2019
>Wuhan/IPBCAMS-WH-05/2020
>mink/Netherlands/NB01_01KS/2020
>mink/Netherlands/NB02_06KS/2020
$ grep '>' example_sequences.fasta | wc -l
418
全部で418件あります。
最初のデータが変異株の比較元(reference sequence)のように思えます。
武漢のもののほか、豪州、欧州、インド、米国のウイルスデータが多数あります。
日本、韓国、台湾、ベトナム、マレーシア、スリランカのデータが一件ずつありました。
最後のふたつはミンクを経由して人間に再感染した変異株と思われます。
サンプルデータの詳細データ(メタデータ)はexample_metadata.tsvにタブ区切りテキスト形式で収められています。
これを見ると各ウイルスデータの採取場所、採取日、採取者、DBへの登録番号、登録日などがわかります。
| strain | virus | gisaid_epi_isl | genbank_accession | date | region | country | division | ... |
|---|---|---|---|---|---|---|---|---|
| Australia/ VIC05/ 2020 | ncov | ? | MT450922 | 2020-03-05 | Oceania | Australia | Victoria | ... |
| ... |
このサンプルデータを使って解析ツールを動かしてみます。
解析実行
Nextstrainコマンドライン解析ツールは専用の実行制御ツール(snakemake)で実行するようになっています。
$ gzip -d -c data/example_sequences.fasta.gz > data/example_sequences.fasta
$ snakemake --cores 4 --profile ./my_profiles/getting_started
解析結果はncov_global.jsonというファイルに出力されます。
これを表示ツールにかければ解析結果が見られます。
Drag and drop
auspice/ncov_global.jsonontohttp://auspice.usin your browser.
Nextstrainの結果表示サイトを開いてブラウザの画面にファイルをドラッグドロップします。
表示サイトには、解析データはJavascriptで表示するのでサイトへは送られないと書かれています。
しばらく待つと解析結果が表示されます(20/01/19実施)。
先に見たNextstrainの表示サイトと似た画面ですが、表示対象のデータは、チュートリアルに含まれるサンプルデータから得られた解析結果です。
データ数が少ないことでそれとわかります。
[Phylogeny]
[Geography]
[Diversity]
まとめ
Nextstrainというコロナウイルス(SARS-CoV-2)の変異ウイルスの解析結果の表示サイト、および解析ツールを実際に使ってみた様子を紹介しました。
ウイルスの生データや解析ツールが一般人にも利用できることがわかり少し驚きました。
次回は、NCBIという生物情報配列データベースのデータを用いて、タンパク質のアミノ酸配列、具体的にはコロナウイルスが人間の細胞に取りつく部分である突起蛋白のアミノ酸配列を解読してみます。
その次は、同じくNCBIのデータを使って、核酸 (DNA/RNA)の塩基配列、具体的にはコロナウイルスのゲノム配列(RNA)を読んでみる予定です。
参考リンク
横浜工文社の関連ページ
Written 2021/02/07