はじめに
一般公開されている、コロナウイルス(SARS-CoV-2)の生物情報の配列データ(biological sequence)の解読を試してみたので体験を共有します。
三回に渡って、生き物の遺伝情報である核酸 (DNA/RNA)の塩基配列やタンパク質を構成するアミノ酸配列の中身を探ってみます。
- Nextstrainでコロナウイルスのゲノム解析を行い解析結果をみてみる
- NCBIの生物情報配列データでコロナウイルスの突起蛋白のアミノ酸配列を解読してみる
- 同じく、コロナウイルスのRNAの塩基配列を解読してみる
二回目は、コロナウイルスが人間の細胞に取り憑く部分の突起蛋白(S Protein)の配列データを解読してみます。
昨年の暮れごろ、イギリス南部、南アフリカ、ブラジルなどで次々と発見され、感染力が増したと疑われる変異株には、突起蛋白に同一のアミノ酸の置き換え(N501Y)が共通に見られることを知りました。そこで、誰でも利用できる生物情報データベース、NCBIからアミノ酸配列データをダウンロードして解読、変異の起こったデータを見つける実験を行いました。その経緯を紹介します。
お断り: 筆者はプログラミングが専門で、医療生物は専門ではありません。できるかぎりウラをとったつもりですが、本稿の記載に知識や理解不足から誤りや誤解を生む箇所があるかもしれません。予めご了承ください。ご指摘をいただければさいわいです。
変異データの発見
米国の国立衛生研究所が中心となって運用している生物情報データベース(NCBI)には、コロナウイルス(SARS-CoV-2)のRNAの塩基配列の全ゲノムデータが多数登録されています。
加えて、RNAから作られる、ウイルスを構成する様々なタンパク質のアミノ酸の配列データも提供されています。
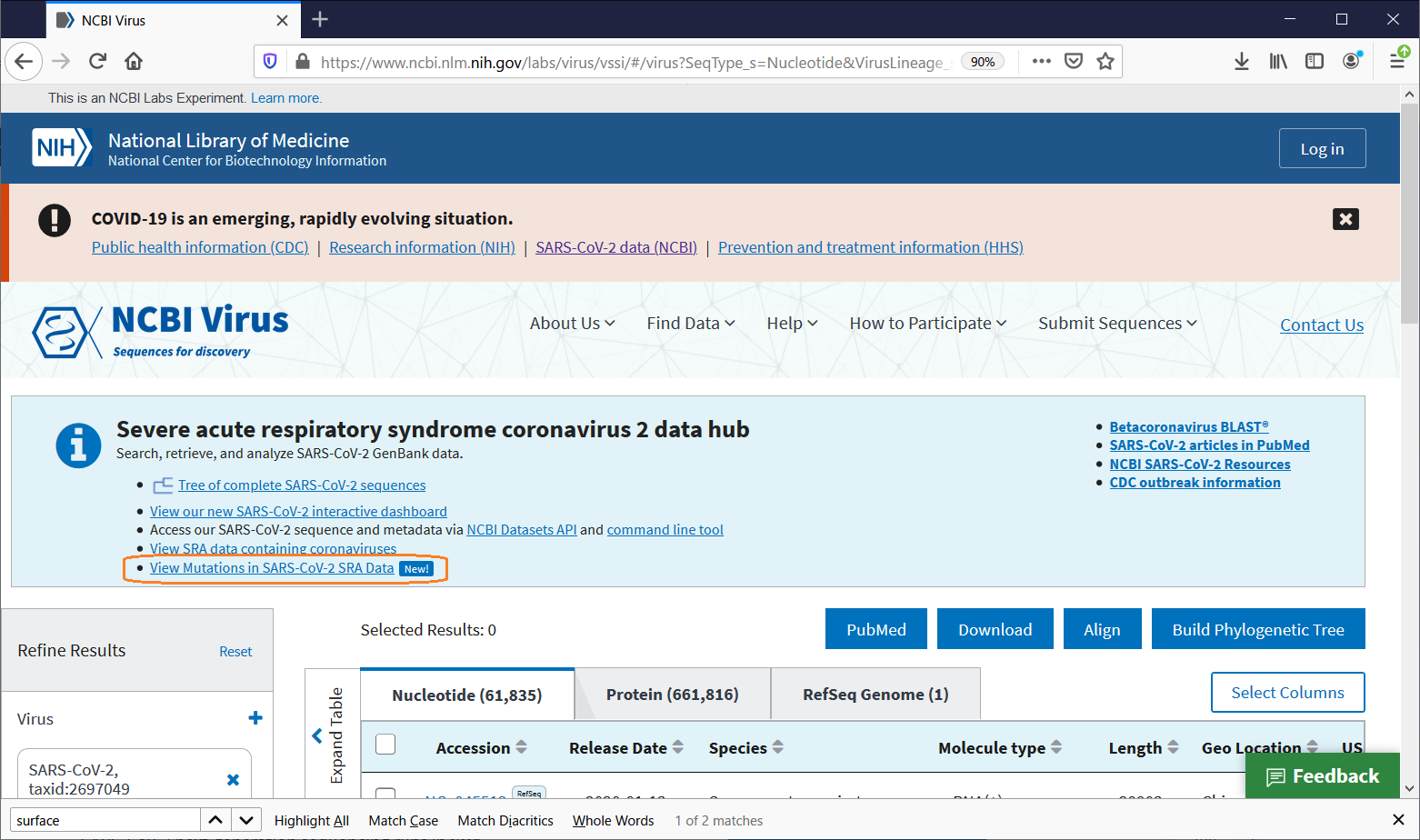
NCBIのコロナウイルスの配列データベースでは様々な方法で検索が行えます。
データ登録時に付与された番号(accession number)がわかればそれでダウンロードできます。
採取地域や採取日など条件を指定して検索することもできます。
配列データに特定のパターンを持つもの、採取データの一部と合致するもの、を探してもらうこともできます。
また、特定のアミノ酸の変異を持つデータの所在をおおまかに知る方法もありました。
そこで「N501Y」の変異を探してみます。
これを見ると、N501Y変異を持つウイルスのデータがデータベースに存在することがわかります。
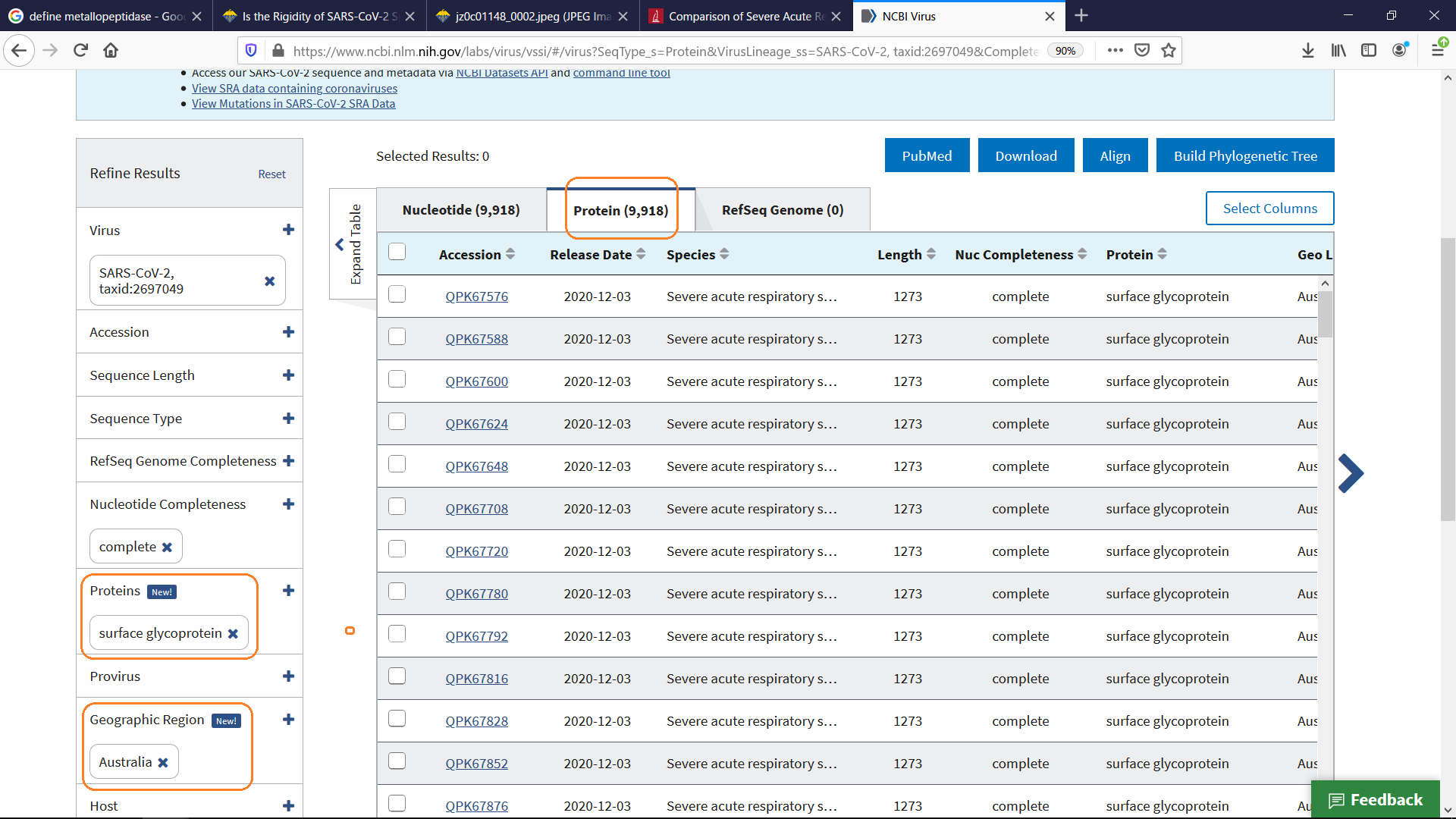
ただ、具体的なデータを辿る方法はわからなかったので、とりあえずオーストラリアのデータを全件取得しました(2012/01/23取得)。
その中からN501Y変異を持つ一番古い配列データを選びました(選んだ方法については省略)。
昨年の6月17に採取されたものでした。
基準となる配列の取得
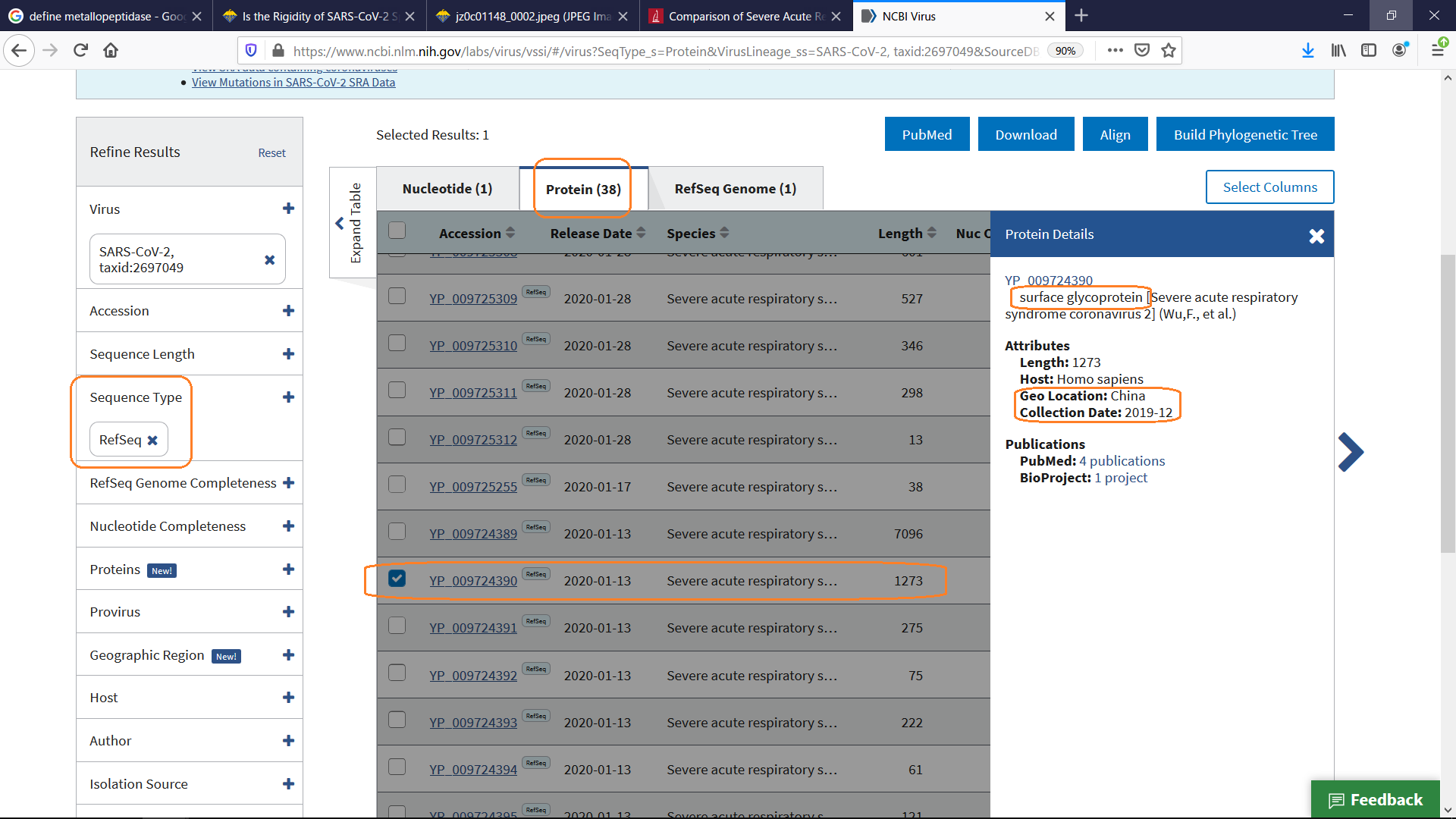
データベースに登録された、特定のウイルス種の変異を調べるためには基準となるゲノム配列データが必要です。
NCBIデータベースには、武漢でおととしの2019年12月に採取されたウイルスデータが参照用の配列データ(refseq = reference sequence)として登録されています。
これもダウンロードします。
配列データの比較
さて武漢の基準配列と豪州の変異株の配列が得られたところで両者を比較してみます。
NCBIからダウンロードした配列データも、初回記事で紹介したNextstrainのチュートリアルデータと同じFASTA形式です。
一行目が'>'ではじまるヘッダ行、それに配列データの英字コードが続きます。
ただしNCBIのデータには改行が入っています。
$ cat pro-refseq.fasta
>YP_009724390.1 |surface glycoprotein [Severe acute respiratory syndrome coronavirus 2]
MFVFLVLLPLVSSQCVNLTTRTQLPPAYTNSFTRGVYYPDKVFRSSVLHSTQDLFLPFFS
NVTWFHAIHVSGTNGTKRFDNPVLPFNDGVYFASTEKSNIIRGWIFGTTLDSKTQSLLIV
...
NGVEGFNCYFPLQSYGFQPT(N)GVGYQPYRVVVLSFELLHAPATVCGPKKSTNLVKNKCVN
...
QELGKYEQYIKWPWYIWLGFIAGLIAIVMVTIMLCCMTSCCSCLKGCCSCGSCCKFDEDD
SEPVLKGVKLHYT
$ cat pro-au-200617.fasta
>QLG76817.1 |surface glycoprotein [Severe acute respiratory syndrome coronavirus 2]
MFVFLVLLPLVSSQCVNLTTRTQLPPAYTNSFTRGVYYPDKVFRSSVLHSTQDLFLPFFS
NVTWFHAIHVSGTNGTKRFDNPVLPFNDGVYFASTEKSNIIRGWIFGTTLDSKTQSLLIV
...
NGVEGFNCYFPLQSYGFQPT(Y)GVGYQPYRVVVLSFELLHAPATVCGPKKSTNLVKNKCVN
...
QELGKYEQYIKWPWYIWLGFIAGLIAIVMVTIMLCCMTSCCSCLKGCCSCGSCCKFDEDD
SEPVLKGVKLHYT
サイズを比べてみます。
$ ls -l pro-*.fasta
-rw-rw-r-- 1 author author 1379 Jan 23 16:38 pro-au-200617.fasta
-rw-rw-r-- 1 author author 1383 Jan 24 09:18 pro-refseq.fasta
$ tail -n +2 pro-refseq.fasta | tr -d '\n' | wc -c
1273
$ tail -n +2 pro-au-200617.fasta | tr -d '\n' | wc -c
1273
ファイルサイズはコメントがあるので当然サイズは異なりますが、中身のデータ部分のサイズは同じです。
上記のコマンドラインの意味は、tailコマンドで先頭行のコメントを除外、その結果からtrコマンドで改行を除外、wcで文字数を把握。
「N501Y」という変異は、タンパク質を構成するアミノ酸のうち501番目のものがNからYに変わった、つまり、AsparagineからTyrosineに変わった、という意味です。
そこで、アミノ酸配列の501番目を比較してみます。
$ tail -n +2 pro-refseq.fasta | tr -d '\n' | cut -c 501
N
$ tail -n +2 pro-au-200617.fasta | tr -d '\n' | cut -c 501
Y
確かに「N」が「Y」に変わっていることが確認できました。
cutコマンドで指定の位置の文字を切り出して表示。
まとめ
誰もが利用可能なNCBIという生物情報配列データベースを用いて、タンパク質のアミノ酸配列、具体的にはコロナウイルスの突起蛋白のアミノ酸配列を解読してみました。
最終回は、同じくNCBIのデータを使って、核酸 (DNA/RNA)の塩基配列、具体的にはコロナウイルスのゲノム配列(RNA)を解読してみる予定です。
参考リンク
横浜工文社の関連ページ
Written 2021/02/07