これまで、ORACLE PGXを使おうと思い、参考資料通りにデモを行ってみたのですが、そろそろ実際に生物の領域(Network Biology)で用いてみようと思います!
今回行うこと
①データの入手
私の前の記事を見てくださっている方は分かると思いますが、おそらく皆さん、生物でどうやって使うんだよ!とか思いますよね?
簡単に言えば、今回行いたいことは生物のビックデータをよりよく解析するといったものです。そのほかにも生物学領域では機械学習やディープラーニングも頻繁に用いられることが増えてきているそうです(テキストマイニングなど)。
個人的に分かりやすくて面白い簡単な記事を紹介します!良かったら見てみてください!(おそらくここを使っている人々は既知のものだと思いますが・・・)
それではやっていきます!
まず、STRINGというタンパク質相互作用のデータベースのあるwebサイトから必要なデータを入手します。
1.今回はヒトのデータを用いました。→Downloads - Homo Sapiens
2.タンパク質相互作用とは、タンパク質どうしが互いにくっついて違う形になったりすることで様々な機能の変化が現れることです。詳しく知りたい方は調べてみてください!
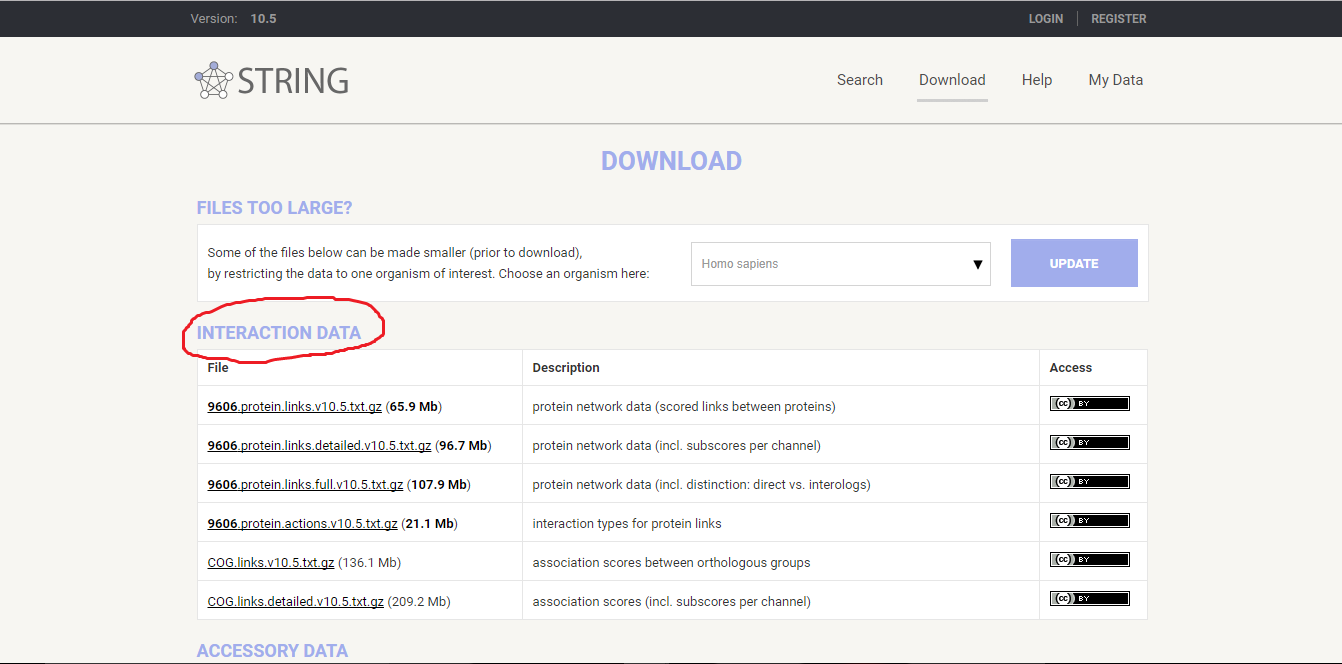
まず最初の画面から、画面右上の「Download」の欄を選択して、検索欄の場所に欲しい生物の学名を入力し、右の「UPDATE」を押します(今回はヒト)。次に、今回はタンパク質どうしのつながりが欲しい情報なので、「INTERACTION DATA」の項から該当するデータをダウンロードします(今回はとりあえず全部ダウンロードしました)。
その後、それらをすべてPGXを入れてあるサーバーへと移し、解凍します。
$ gzip -d [ファイル名]
②必要なデータを処理してPGXにロードできるようにする
今回すべてダウンロードしたわけですが、もちろん全部は使いません。ここから必要なものを選び、処理していきます。今回はタンパク質どうしのつながりが見たいので、タンパク質の情報(ID等)があるファイル(今回の9606.protein.links.v10.5.txt)と簡潔な繋がり具合を示したファイル(今回のuniprot.tsv)との大きく分けて2種類のものを使用しました。
ORACLE PGXはグラフ・プロパティのデータベースを扱う分析エンジンなので、ノードとエッジ(点と線)に情報を持たせればよいということだそうです!
私は今回シェルスクリプトを用いてファイルの整形、結合を行いました。
# !/bin/bash
awk -v FS='\t' -v OFS=',' '{if (NR != 1) print $1, $4}' uniprot.tsv > temp_unipro
t.csv
sed "s/9606\.//g" csv/9606.protein.links.v10.5.txt | sed "s/ /,/g" | \
awk -v FS=',' -v OFS=',' '{if (NR != 1) print $1,$2,$3}' | head -n $1 | sed "s/,
$/,0/g" > temp_links.csv
awk -v FS=',' -v OFS=',' '{print $1, "*", $1}' temp_links.csv | sort | uniq > temp
_nodes.csv
awk -v FS=',' -v OFS=',' '{print $2, "*", $2}' temp_links.csv | sort | uniq >> tem
p_nodes.csv
sort -t ',' -k 1 temp_nodes.csv | uniq > temp_nodes_sorted.csv
sort -t ',' -k 1 temp_uniprot.csv | sed "s/_HUMAN//g" > temp_uniprot_sorted.csv
q -d, "SELECT * FROM temp_uniprot_sorted.csv GROUP BY c1" > temp_uniprot_sorted_un
iq.csv
join -t ',' temp_nodes_sorted.csv temp_uniprot_sorted_uniq.csv -1 1 -2 1 -o 1.1,1.
2,2.2 > temp_nodes_2.csv
cat temp_nodes_2.csv temp_links.csv > protein_edgelist.csv
rm temp_*
簡潔に言うと余計な文字をsedにより削除、awkでデータの整形、sort, uniqで順序の整理、join, catでファイルの結合を行いました。(catでファイル全体の結合できることを初めて知りました、、、)
更にこれをPGXにロードするためのJSONファイルの作成も行いました。
{
"uri": "protein_edgelist.csv"
, "format": "edge_list"
, "vertex_id_type": "string"
, "vertex_props":[
{"name":"gene_name", "type":"string"}
]
, "edge_props":[
{"name":"score", "type":"integer"}
]
, "separator": ","
}
vertex_id_typeでノードのIDを定義して、vertex_propsでノードの情報(今回は遺伝子名)を定義しています。またedge_propsでエッジのスコア情報(繋がり具合)を定義しています。
ちなみに、、
uniprot.tsvはどこから入手したかというと、
(ⅰ)UniprotのID Mappingへ行きます
(ⅱ)そこで、ファイル選択の部分を押し、STRINGでダウンロードしたEnsemble IDのリスト(ノードに当たるリスト)を選択します。
(ⅲ)その後、下の方のSelect Optionsで、From Ensemble Proteinを選択し、GOのボタンを押します。
(ⅳ)次に表示されるページで、Download➡Tab-separated Formatを選択し、uniprot.tsvがダウンロードされます!
これで皆さんもタンパク質相互作用のネットワークを分析することが出来ますね!
最後駆け足になったかもしれませんが、有難うございました!
追記:ロードが上手くいかない状態なので上手くいったらまた訂正させていただきます、、
[8/23] シェルスクリプトをの部分を書き直しました!もう少し整形してあります。そして、読み込めない原因がダウンロードしたデータの方にあった(スコアの部分に何も書いてないところが散見された)ので、そこに0という値を入れるようにしてあります。別にダウンロード先ふざけんなよ!なんて言ってないですからね!笑
そして更に、「q」コマンドというものも使用しました!これはSQLのようなクエリを書けるコマンドらしいです!(すごい)
[8/30] シェルの一部を修正しましたsed "s/9606.//g" ⇒ sed "s/9606\.//g"