この記事はTSG Advent Calendar 2022の11日目のエントリです。
推論モデルが選択可能なONNXファイルを作りたい
複数のONNXモデルをユーザーの入力によって使い分けたいとき、呼び出し側で複数のInferenceSessionを立てるという手がありますが、呼び出し側のプログラムを変更するのが面倒というケースもあると思います。そこでこの記事では、複数のONNXを一つにまとめ内部で分岐させる方法を紹介します。
扱うモデルはresnet18, 34, 50として、引数depthに従って使われるモデルが変化するONNXを作成してみます。Pytorchで表現すると次のようになります。
import torch
from torch import nn
from torchvision import models
class Model(nn.Module):
def __init__(self):
super().__init__()
self.resnet18 = models.resnet18()
self.resnet34 = models.resnet34()
self.resnet50 = models.resnet50()
def forward(self, x, depth):
if depth == 0:
return self.resnet18(x)
elif depth == 1:
return self.resnet34(x)
else:
return self.resnet50(x)
ちなみにこのPytorchモデルはtorch.jit.script()でtorchscript化すればそのままtorch.onnx.export()でif文を適切に処理してくれるONNXを出力可能です。この記事ではONNXモデルのみが手元にある状態を仮定しています。
モデルのリネーム
まず複数のONNXモデルをマージするにあたって、名前衝突が起こらないようにそれぞれのONNXモデルに対してprefixを追加します。
from copy import deepcopy
from pathlib import Path
from typing import List
import torch
import onnx
import onnx.helper
import onnx.compose
def rename(graph, prefix: str, freeze_names: List[str]):
"""freeze_namesで指定した名前以外にprefixを追加する"""
for node in graph.input:
if node.name not in freeze_names:
node.name = prefix + node.name
for node in graph.output:
if node.name not in freeze_names:
node.name = prefix + node.name
for node in graph.node:
for i, n in enumerate(node.input):
if n not in freeze_names and n != "":
node.input[i] = prefix + n
node.name = prefix + node.name
for attr in node.attribute:
if hasattr(attr, "g"):
for subnode in attr.g.input:
if subnode.name not in freeze_names:
subnode.name = prefix + subnode.name
for subnode in attr.g.output:
if subnode.name not in freeze_names:
subnode.name = prefix + subnode.name
rename(attr.g, prefix, freeze_names)
for i, n in enumerate(node.output):
if n not in freeze_names and n != "":
node.output[i] = prefix + n
for init in graph.initializer:
init.name = prefix + init.name
graphs = [onnx.load(path).graph for path in ["resnet18.onnx", "resnet34.onnx", "resnet50.onnx"]]
# 最終的な入力・出力ノードは元のグラフのものを使いまわす。graphsのすべてのonnxにおいて入出力は同じshapeである必要がある。
input_nodes = deepcopy(list(graphs[0].input))
output_nodes = deepcopy(list(graphs[0].output))
# 入力ノードは共有したいのでgraph.inputにあるノードにprefixを付けない
for i, graph in enumerate(graphs):
rename(graph, f"m{i}.", [n.name for n in graph.input])
ちなみに、今回のresnetのように内部にGraphProtoを持たないものをリネームしたいときはonnx.compose.add_prefix_graph()を活用することができます。ただしIfやLoopなど、GraphProtoが入れ子になっているノードを含むONNXに対しては、onnx 1.12.0時点で正常に働かない可能性があり、その場合は上のような再帰関数を自前で実装する必要があります。
(onnx.composeについて:https://github.com/onnx/onnx/blob/main/docs/PythonAPIOverview.md#onnx-compose)
Ifノードの追加
次にgraphsをIfノードで連結します。Ifの仕様は以下のようになっています。
Attributes
else_branch : graph (required)
then_branch : graph (required)
Inputs
cond : B
Outputs (1 - ∞)
outputs (variadic, heterogeneous) : V
condの真偽によってthen_branchとelse_branchのどちらか一方のグラフが実行されアウトプットが取り出されるのですが、このとき注意すべきは、*_branchに指定するグラフにinputを指定してはいけないという点です(Tensorflowにおけるtf.condに指定するラムダ式に引数が存在しないことに似ていますね)。じゃあ入力を持つgraphは使えないのかというとそんなことは無く、Ifノードより前に宣言されたテンソルはグローバル変数のように利用することができます。
それではdepthによって呼び出すグラフが変わるようなモデルを作っていきます。
残念ながらswitch-caseにあたるものがonnxには無いので、今回のケースでは以下のようにIfを連結していきます。
depth = onnx.helper.make_tensor_value_info("depth", onnx.TensorProto.INT64, [1])
# Ifのbranchに使えるようにinputを消したグラフを作る
branches = []
for i, g in enumerate(graphs):
branches.append(onnx.helper.make_graph(
nodes=list(g.node),
name=f"branch_m{i}",
inputs=[],
outputs=list(g.output),
initializer=list(g.initializer)
))
# depthとの比較用の定数
offset_consts = []
for i in range(len(graphs) - 1):
offset_consts.append(onnx.helper.make_tensor(
name=f"offset_{i}",
data_type=onnx.TensorProto.INT64,
dims=(),
vals=[i]
))
# depthと定数の比較をするノード
select_conds = []
for i in range(len(graphs) - 1):
select_conds.append(onnx.helper.make_node(
"Equal",
inputs=["depth", f"offset_{i}"],
outputs=[f"select_cond_{i}"]
))
# Ifの入れ子になるようにグラフを繋ぐ
whole_graph = branches[-1]
for i in range(len(branches)-2, -1, -1):
if_node = onnx.helper.make_node(
"If",
inputs=[f"select_cond_{i}"],
outputs=[n.name for n in output_nodes],
then_branch=branches[i],
else_branch=whole_graph
)
whole_graph = onnx.helper.make_graph(
nodes=[select_conds[i], if_node],
name=f"branch{i}",
inputs=[],
outputs=output_nodes,
initializer=[offset_consts[i]]
)
opset = 14
whole_graph = onnx.helper.make_graph(
nodes=list(whole_graph.node),
name="whole_model",
inputs=[depth] + input_nodes,
outputs=output_nodes,
initializer=list(whole_graph.initializer)
)
whole_model = onnx.helper.make_model(whole_graph, opset_imports=[onnx.helper.make_operatorsetid("", opset)])
onnx.checker.check_model(whole_model)
onnx.save(whole_model, "whole_model.onnx")
これでモデルを合成できました。推論時は以下のように使うことができます。
import onnxruntime as ort
import numpy as np
ort_sess = ort.InferenceSession('whole_model.onnx')
outputs = ort_sess.run(None, {'input': np.zeros((1, 3, 224, 224), np.float32), 'depth': np.array([0], np.int64)})
最後にNetronでグラフを見てみます。
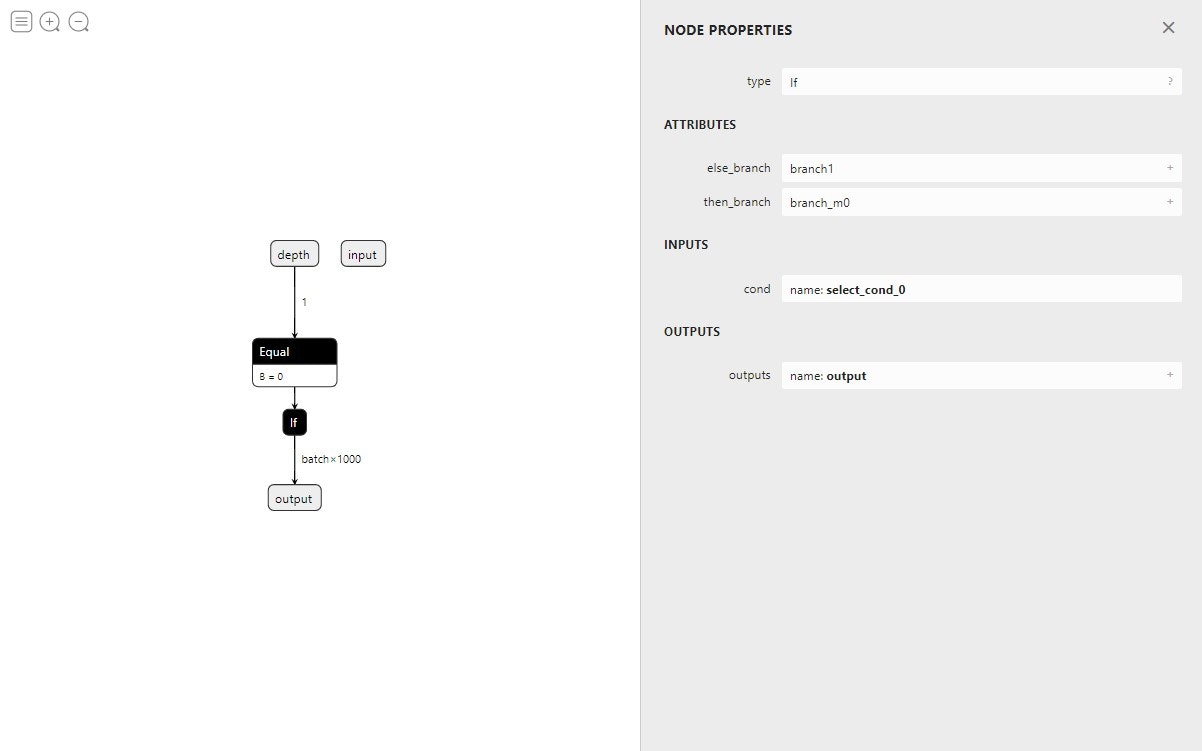
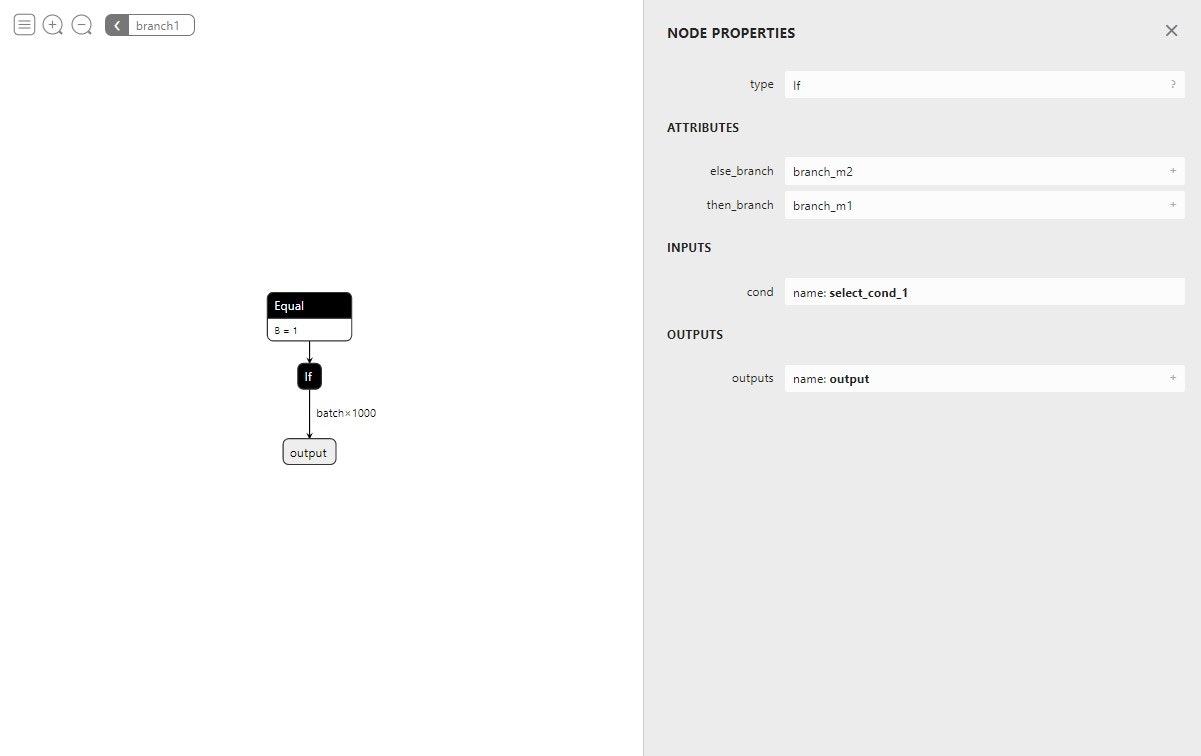
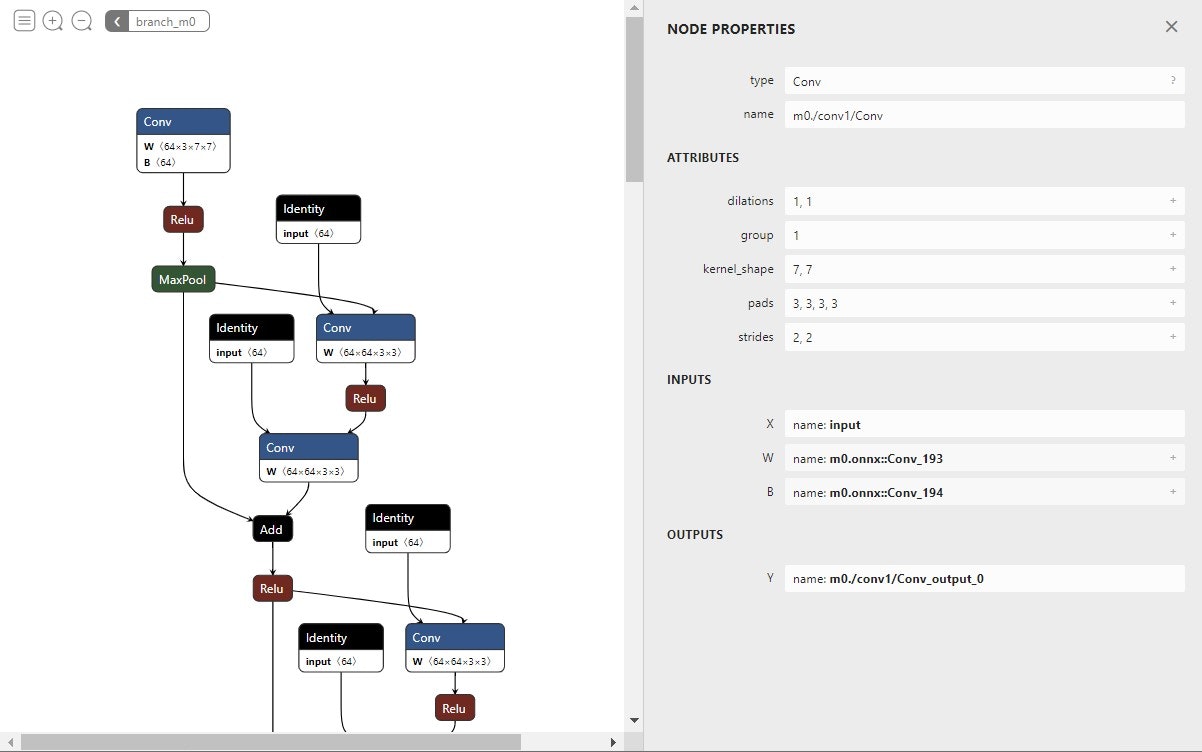
一部見た目が寂しい感じになっていますが、branch1などをクリックすると入れ子になっているグラフを観察することができます。
おわりに
Ifを使ってモデルを並列化しようとしたときにいろいろ調べまわる羽目になったので備忘録的にこの記事を書きました。基本的にはtorch.jit.scriptや呼び出し側の処理で何とかなる問題ですが、どうしてもONNXモデルをいじることになった方は参考にしてみてください!