NCBIの論文情報をAPI(E-Utils)を用いて取得し、ローカル環境でPythonで解析しましたので、その方法について記録します。
記載時:2021/9/10
環境:macOS Catalina(10.15.7)、Python 3.8.5
1.E-Utilsについて
E-Utilsを用いたGEOへのアクセスについては下記に概要が説明されています。
簡単に説明すると、例えばPMIDを引数として、論文タイトルやDOIを返り値として取得という作業をCUIで行うことができます。
2.PUbMedから論文情報を取得してみる。
E-UtilsにはeSearch、eSummary、eFetch、ePost、eLink、eInfoなどのプログラムがあります。
今回は試しにeSummaryを使用して、引数として渡したPubMed IDからDOIを返すプログラムを作成してみます。
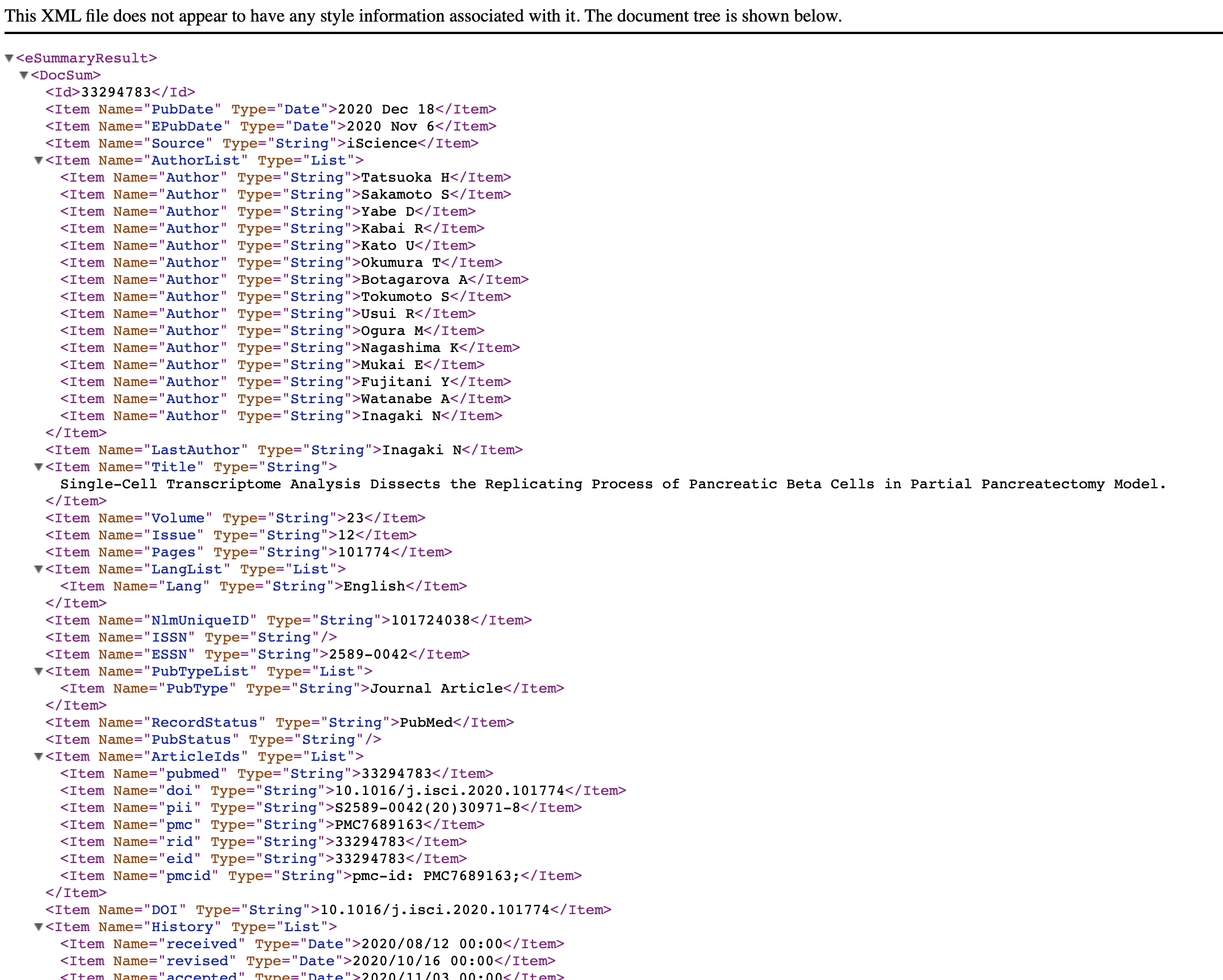
まず、Webブラウザでアクセスして概要を把握します。今回はXML形式でデータを取得します。実際に(PMID) 33294783の論文(私の学位論文 笑)の情報を取得してみます。
URLは{使用するプログラムのURL}?{クエリパラメータ}となります。
例:https://eutils.ncbi.nlm.nih.gov/entrez/eutils/esummary.fcgi?db=pubmed&id=33294783&retmode=xml
このように表示されます。
PythonでこのXMLデータをローカル取得し、必要な情報を抽出したいと思います。
3.PythonでAPIからアクセス
# pubmed idを入力してDOIを返すプログラム
import sys
import urllib.parse as parse
import urllib.request as req
from bs4 import BeautifulSoup
# 基礎の指定
API = "https://eutils.ncbi.nlm.nih.gov/entrez/eutils/esummary.fcgi"
# 引数を取得
if len(sys.argv) <= 1:
print("USAGE: pmid_doi.py (PMID)")
sys.exit()
pmid = sys.argv[1]
# パラメータをURLエンコード
query = {
"db": "pubmed",
"id": pmid,
"retmode" : "xml",
}
params = parse.urlencode(query)
# HTMLを取得
url = API + "?" + params
print(url)
res = req.urlopen(url)
# Beautifulsoupで解析
soup = BeautifulSoup(res, "html.parser")
# セレクタの結果はリストになるので抽出
DOI = soup.select("Item[Name='DOI']")[0]
# 文字列のみ抽出
DOI = DOI.string
# 結果表示
print(DOI)
解説;APIへのアクセスのためにurllibモジュールのparseパッケージとrequestパッケージをインポートします。取得したXMLデータの解析のため、BeautifulSoupをインポートしておきます。
まず、基礎となるプログラムのURLを指定します(eSummaryを使用するのであれば
https://eutils.ncbi.nlm.nih.gov/entrez/eutils/esummary.fcgi
を指定します)。
続いて、URLクエリパラメータをdictionary形式で指定します。
今回はデータベースとしてpubmed、データ形式はxmlとします。
辞書データをparseしてURLとして渡せる形式にします。
urlopenで取得した結果をBeautifulSoupで解析。"html.parser"を使用します。
属性と名前を指定し、情報を取得します(リスト形式になっているため0で抽出します)。
文字列のみsrtingで取得すると完成です。
$ python3 pmid_doi.py 33294783
https://eutils.ncbi.nlm.nih.gov/entrez/eutils/esummary.fcgi?db=pubmed&id=33294783&retmode=xml
10.1016/j.isci.2020.101774
うまくいっているようです。
同様に様々な情報を取得することができます。
参考:rettype、retmodeなどのパラメータの組み合わせについてはlinkのtableを参考に。