記事の目的
分散固定の場合の混合ガウスモデルのギブスサンプリングをRで実装することです。
参考書籍: 佐藤一誠. ノンパラメトリックベイズ. 講談社. 2016
この書籍のアルゴリズム4.2に対応します
目次
1. モデルの説明
2. 使用データ
3. 分散固定の混合ガウスモデルのギブスサンプリング
4. クラスタリング確認
1. モデルの説明
2. 使用データ
> X <- iris[,1:4]
> D <- ncol(X)
> N <- nrow(X)
> head(X)
Sepal.Length Sepal.Width Petal.Length Petal.Width
1 5.1 3.5 1.4 0.2
2 4.9 3.0 1.4 0.2
3 4.7 3.2 1.3 0.2
4 4.6 3.1 1.5 0.2
5 5.0 3.6 1.4 0.2
6 5.4 3.9 1.7 0.4
>
3. 分散固定の混合ガウスモデルのギブスサンプリング
library(dplyr)
library(mvtnorm)
# (1)
K <- 3
# (2)
u <- matrix(rep(apply(X, 2, mean),each=K), nrow=K, ncol=D)
# (3)
var <- 1
pi <- rep(1/K, K)
# (4)
max.iter <- 30
Norm <- {}
z <- {}
n <- {}
set.seed(1)
for(s in 1:max.iter){
# (ⅰ)
for(i in 1:N){
for(k in 1:K){
Norm[k] <- dmvnorm(X[i,], u[k,], diag(D))
}
z_prob <- Norm/sum(Norm)
z[i] <- rmultinom(1, 3, z_prob) %>% which.max()
}
# (ⅱ)
for(k in 1:K){
n[k] <- z[z==k] %>% length()
x.k <- X[z==k,] %>% apply(2, mean)
u[k,] <- rmvnorm(1, n[k]*x.k/(n[k]+var), diag(D)/(n[k]+var))
}
s <- s+1
}
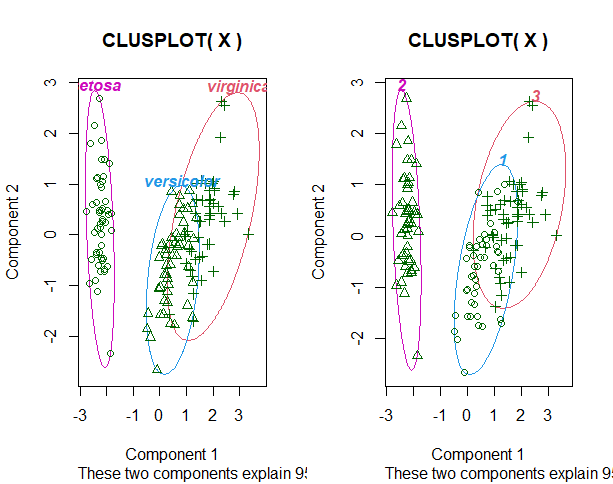
4. クラスタリング確認
library(cluster)
par(mfrow=c(1,2))
clusplot(X, iris[,5], color=TRUE, shade=FALSE, labels=4, lines=0)
clusplot(X, z, color=TRUE, shade=FALSE, labels=4, lines=0)