はじめに
本記事は分子描画フリーソフトのJmolを用いた分子軌道の自動描画法について紹介します。
対象者は計算化学者、特に初学者の方です。普段、手作業で分子軌道を貼りまくっている方、Jmolで自動化しましょう。
Jmolのダウンロードについては以下を参考にしてください。
Jmol でできること
Jmolはフリーの描画ソフトです。以下のような情報を描画できます。
- 分子構造
- 分子振動
- 分子軌道 など
Jmolは汎用的な量子化学計算プログラムのフォーマットに対応しています。
XYZ, GAMESS, Gaussian, MOLPRO, MOPAC, NWCHEM, QOUT, GRO, Amber, VASP, Molden など
詳細は https://jmol.sourceforge.net/ を確認してください。
Jmolはほぼすべての機能をスクリプトで操作できます(というかマウスでは操作しにくい)。
そのため、実行したい機能をスクリプトに書けば操作を自動化することができます。
基本的な操作
Chem-Station さんで紹介されています。こちらをご覧ください。
スクリプトで自動化する
今、このようなjmol_testディレクトリにいるとします。xxx.logは量子化学計算プログラムが出力した分子軌道を含むlogファイルだとします。
jmol_test/ --- ch4.log
h2o.log
nh3.log
Jmol > File > Script Editor をクリックしてスクリプトエディターを開き、以下のようなスクリプトを記入してください。
load ./h2o.log
background [x101010]
frank off
set showMultipleBonds off
mo color red blue
mo resolution 10
mo translucent
mo fill mesh
mo cutoff 0.05
mo titleformat ""
rotate best
mo ON
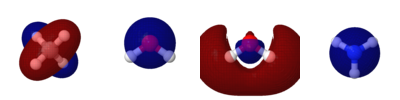
mo HOMO
write IMAGE 600 600 PNGT 10 ./h2o_HOMO.png
mo LUMO
write IMAGE 600 600 PNGT 10 ./h2o_LUMO.png
ここで上記のコマンドを簡単に紹介します。
load ./h2o.log ファイル読み込み
background [x101010] 背景色を変更(透明化?)
frank off Jmolロゴを削除
set showMultipleBonds off 多重結合の表示をoff
mo color red blue 分子軌道の色を設定(yellow purpleも指定可能)
mo resolution 10 解像度を指定
mo translucent 分子軌道を透過させる
mo fill mesh 分子軌道をメッシュで表す
mo cutoff 0.05 カットオフ
mo titleformat "" タイトル(文字)を削除
rotate best 分子をある基準で回転させる
mo ON 分子軌道描画をスタート
mo HOMO HOMOを描画。mo n とすると、n番目の分子軌道を描画
write IMAGE 600 600 PNGT 10 ./h2o_HOMO.png
Jmolの画面を画像として保存
他にもコマンドはたくさんあります。
例えばこちらのように。
##----------------------------------
## データの読み書き
##----------------------------------
load INPUT-PATH
INPUT-PATHで指定されたファイルをロード。
write IMAGE 2400 2400 PNGT 10 "OUTPUT-PATH"
OUTPUT-PATHで指定されたファイルに書き出す。
##----------------------------------
## 分子の回転、移動
##----------------------------------
rotate best
分子の長軸が画面上(平面上)かつ短軸が画面に垂直になるように分子を自動的に配置。
rotate x 90
x軸を+90°回転。
rotate BRANCH {C2} {C1}
2番目の炭素の後ろに1番目の炭素を重ねるように配置(たぶん)。
rotate @{quaternion({1 0 0}, 180)}
x軸を180°回転。
translate x 4.5
x軸方向に平行移動。
translate y 10
y軸方向に平行移動。
moveto 0 1 0 0 -90;
view from top
moveto 0 0 1 0 90;
view from right
moveto 0 0 1 0 -90;
view from left
moveto 2 1 0 0 90;
view from bottom with smooth 2-second transition
moveto 0 0 0 0 0 200;
default orientation with instantaneous transition and a zoom of 200%
##----------------------------------
## 便利な設定、Defaultの設定
##----------------------------------
frank off
右下に表示されるJmolを削除。
background [x101010] / white
背景を透明化。色を指定することもできる。
set showMultipleBonds off
多重結合の表示の有無。
set defaultTranslucent off / 0 to 1
透過度のdefaltを設定。
set axesColor black
座標軸の色を設定
set diffusePercent 60 ## 0 to 100 : 0 means no diffuse of light
光源の広がりを指定。0にすると非常にまばらな光、100にするとスポットのようになる。
set specularPercent 60 ## 0 to 100 : The reflection of light
光の反射の度合い。
set specular off
光の反射の有無。
set fontSize 12
ラベルの大きさ(フォントサイズ)を指定。
set labelAtom ON
原子にラベルを表示。
set perspectiveDepth off
遠近感の有無。
##----------------------------------
## 座標軸の設定
##----------------------------------
axes ON
座標軸の表示の有無
axes 2
座標軸の太さ。
axes SCALE 2
座標軸の長さ。
axes CENTER {0.0 0.0 0.0}
座標軸の原点を指定。
center {C1}
座標軸の原点を1番目の炭素に指定。
axes LABELS "" "" "z" "y" "x" ""
座標軸にラベルを付ける。x, y, z, -x, -y, -zの順になる。
unitcell boundbox
分子が収まるようなunitcellを箱型で表示。
unitcell 0.02
unitcellの辺の太さを変更。
##----------------------------------
## 分子の設定
##----------------------------------
spacefill 0.08 ## The radius of atom
分子の大きさ。
wireframe 0.04 ## THe radius of bond
結合の太さ。
##----------------------------------
## 分子軌道の設定
##----------------------------------
mo Delete
分子軌道を削除。
mo CUTOFF 0.002058 ## 0.005-0.05(default)
分子軌道の広がりを調整。小さいほど空間的に広がった軌道になる。
mo LUMO+8
任意の分子軌道を可視化。
mo resolution 10
分子軌道の解像度を指定。
mo mesh nofill
分子軌道をメッシュで表現。surfaceはなし。
mo fill nomesh
分子軌道をsurfaceのみで表現。
mo translucent (0-1の数値を指定可) ## or mo opaque
分子軌道を透過させる。
mo titleformat ""
左上に任意のデータを表示。JmolのHPに書式がある。
mo color red blue ## or yellow purple
分子軌道の色を指定。
mo plane "xz"
任意の座標空間に射影した分子軌道を可視化。
その後、スクリプトエディターのRunコマンドを押してスクリプトを実行(Run)しましょう。
すると、Jmol 上で分子軌道が描画されます。
writeコマンドを指定したので、それぞれの分子軌道がpngファイルとして保存されます。
jmol_test/ --- ch4.log
h2o.log
h2o_HOMO.png
h2o_LUMO.png
nh3.log
他のファイルの分子軌道も描画したい場合はloadとwriteを繰り返せばOKです。
load ./h2o.log
mo HOMO
write IMAGE 600 600 PNGT 10 ./h2o_HOMO.png
load ./nh3.log
mo HOMO
write IMAGE 600 600 PNGT 10 ./nh3_HOMO.png
load ./ch4.log
mo HOMO
write IMAGE 600 600 PNGT 10 ./ch4_HOMO.png
今回の例では、loadやwriteを繰り返しあらわに書きましたが、Jmolスクリプトのloop構文を使えばよりスッキリしたスクリプトを書くこともできます。
ただJmolスクリプトのloopは(少なくとも私にとっては)わかりにくいと思います。
そのため、Jmolスクリプトでloopを使うよりも、みなさんが使い慣れた言語(例えば Python)でJmolスクリプトを生成するスクリプトを書くほうが無難です。
sptファイルで自動化する
Jmolスクリプトはテキストファイル(spt形式)から直接実行することもできます。
例えば、先ほど紹介したスクリプトをjmol_test.sptとして保存し、Jmol > File > Open で開いてみてください。
(Linuxの場合はjmol jmol_test.sptを実行すればよい。)
load ./h2o.log
...
mo HOMO
write IMAGE 600 600 PNGT 10 ./h2o_HOMO.png
mo LUMO
write IMAGE 600 600 PNGT 10 ./h2o_LUMO.png
すると、スクリプトエディターで記入したときと同じような画像が出力されます。
分子配向の制御が難しい
分子軌道を描画するときは分子の向きを揃えたり、見やすい向きに調整する必要があります。
Jmolのrotate bestコマンドは分子の長軸と短軸を使って向きを揃えてくれますが、分子軌道の確認には不都合な場合が多いです。
私自身、分子軌道を自動描画する前にJmolで素性の良い分子配向になるようなrotate、translateコマンドを見つけ出し、スクリプトに追加するようにしています。ただ、これはかなり面倒です。
配向制御に関して何かいいアイディアがあればコメントいただければ幸いです。
分子軌道の画像に情報を出力
mo TITLEFORMATコマンドを使うことで、画像(デフォルトでは左上)に電子状態計算により得られた情報を出力することもできます。
コマンドの使い方は以下のとおりです(日本語マニュアル)。
mo TITLEFORMAT “format"
format で利用可能な変数
- %E (エネルギー)
- %F (ファイル名)
- %I (MOの番号)
- %M (モデルの番号)
- %N (MOの数)
- %O (占有数)
- %S (対称性)
- %U (エネルギー単位)
- | (改行)
- ? (データがなければ無視する)
デフォルトは
"%F|Model %M MO %I/%N|Energy = %E %U|?Symmetry = %S|?Occupancy = %O"
ImageMagick と組み合わせる
ImageMagickは画像を操作・表示するためのソフトウェアです。日本語マニュアルもあるようです。
Windows、Macでも使えるようですが、ここではLinuxコマンドを紹介します。
分子軌道をタイル状に並べる
分子軌道の画像をたくさん作った後、PowerPointなどに貼り付ける人が多いのではないでしょうか。
何十枚もの画像をpptに貼り付けるのは手間がかかります。自動化しましょう。
ImageMagickのmontageは画像を"タイル状"に並べてくれる機能です。
今、このような状況だったとします。
jmol_test/ --- ch4.log
ch4_HOMO.png
h2o.log
h2o_HOMO.png
h2o_LUMO.png
nh3.log
nh3_HOMO.png
ここで以下のようなコマンドを入力すると、4個のpngファイルを1行4列(4x1)で配置したmo.pngが生成されます。
montage *.png -background '#00000000' -tile 4x1 -geometry 100x100 mo.png
分子軌道変化のアニメーションを作る
ImageMagickのconvertコマンドを使えば、複数の画像データからgifアニメを作ることもできます。
ここでは、切り替わる間隔が1秒でループし続けるアニメーションを作ることができます。
convert -delay 100 -loop 0 *.png output.gif
-delay :画像が切り替わる時間幅(1/100秒単位。-delay 100で1フレーム/秒。)
-loop :ループ回数(0の場合、無限回ループする。)
詳しくはImageMagick で GIF 処理をご覧ください。
Q&A
Q1. Gaussian16のfchkファイルが開けない
JmolはGaussian16のformchkに対応していない可能性があります。
私の環境ではGaussian16のformchkで生成したfchkファイルを開こうとすると
Error reading file at end of file
null
for file /Path2fchk
type GaussianFchk
のようなエラーメッセージが出力されます。このバグ(?)はGaussian09のformchkを使うことで回避することができます。
Q2. 構造最適化後の分子軌道が描画されない
Jmolで構造最適化計算や固有反応座標(IRC)計算の結果を開くと、最終構造だけではなく途中経過の構造も認識されます。
Jmolスクリプトでは、これらの構造をframeコマンドで操作することができます。
構造最適化後の分子軌道やIRCに沿った分子軌道変化を描画するためには、frameを次に送りながらmo HOMOなどを入力する必要があります。
frameコマンドは以下のように使うことができます。
load ./h2o_opt.log
animation ON
frame NEXT
mo HOMO
write IMAGE 600 600 PNGT 10 ./h2o_opt1_HOMO.png
frame 5
mo HOMO
write IMAGE 600 600 PNGT 10 ./h2o_opt5_HOMO.png
frame LAST
mo HOMO
write IMAGE 600 600 PNGT 10 ./h2o_optLAST_HOMO.png
また、animation ONコマンドを使うと、frameに沿ったアニメーションを見ることができます。
frameやanimationコマンドの詳細は以下をご覧ください。
おわりに
少しでも多くの人が分子軌道描画・貼り付け地獄から解放されますように。