RDKitのPandasToolで画像が表示されなくなる問題
最初から解決策を知りたい方はこちら
問題
RDKitのPandasToolsで画像が表示されなくなる時がある
RDKitのPandasTools、便利ですよね。でもデータフレームを操作していくうちにROMolカラムの画像が出力されなくなる時ありませんか?
例えばこんな感じ↓
# とりあえずいつものインポート
import pandas as pd
from rdkit import Chem
from rdkit.Chem import PandasTools, Descriptors
# PandasToolsのLoadSDFでsdfファイルをロードしてデータフレームを作成
data = PandasTools.LoadSDF('./data/example_data.sdf')
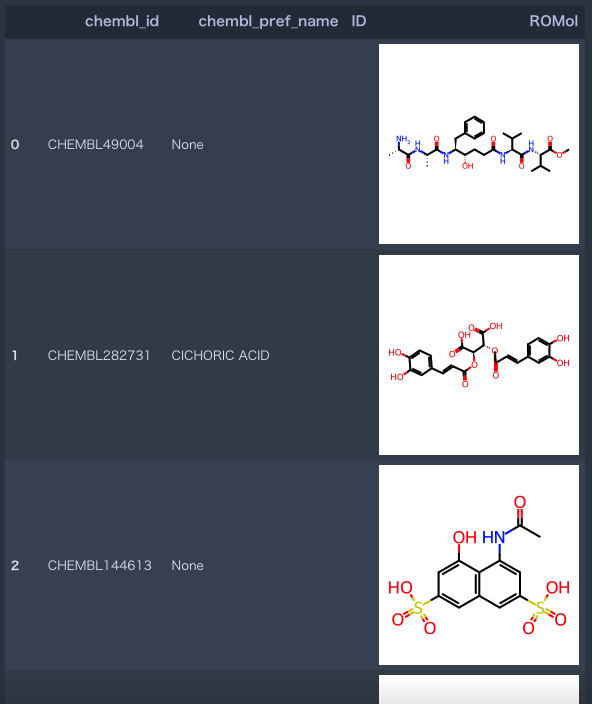
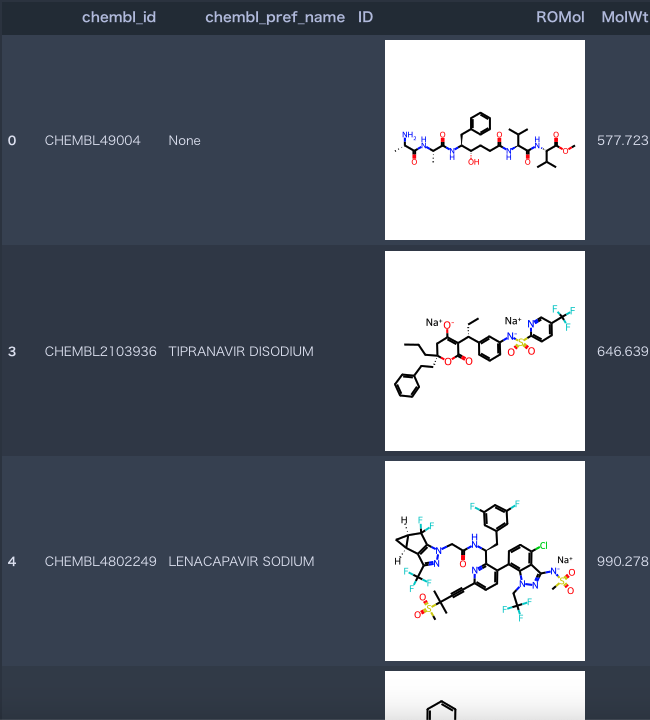
data # とりあえず、データフレームを表示してみる
この時点ではきちんと化合物の画像が表示される
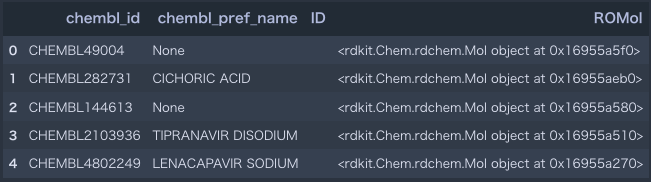
でも.head()で表示すると…
data.head()
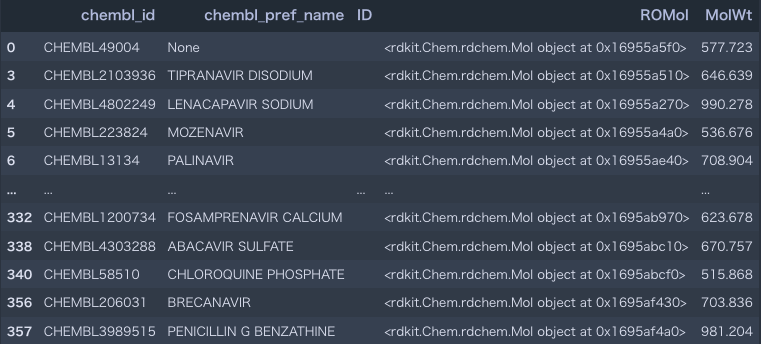
このように画像が文字表現になってしまっている。
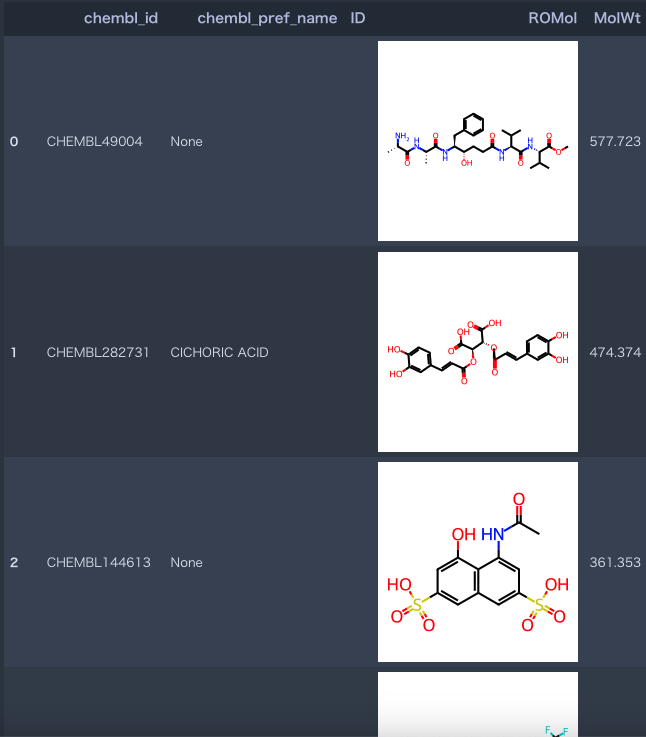
他のケースとして、例えば記述子での絞り込みをしたい時↓
data['MolWt'] = data.ROMol.map(Descriptors.MolWt) # 例として分子量をカラムに追加
data # 表示
このままだと、普通に画像込みで表示されるが、
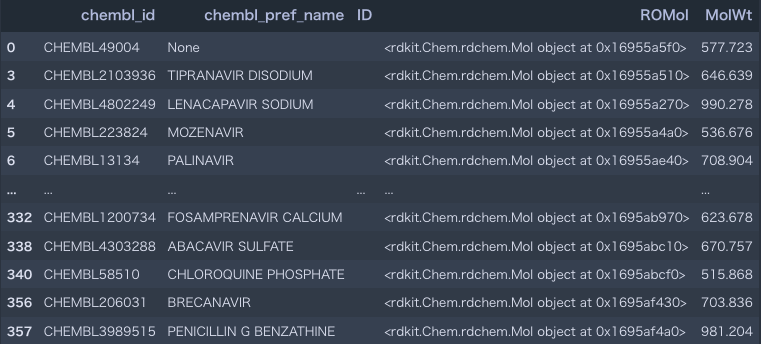
data[data.MolWt >= 500] # 分子量500以上を表示
.head() と同じように画像が文字表現で出力されてしまう
原因と解決方法
先に解決策が知りたい方はこちら
原因
正直、文字表現でもmolオブジェクトとしては普通に扱えるので、気にならなければこのままでも良い。
しかし、慣れないうちは色々と確認しながら作業したいし、操作してる化合物がどんなものなのか知りたいと思うことは多い。
そもそも、なぜこんなことが起こるのか簡単に説明すると
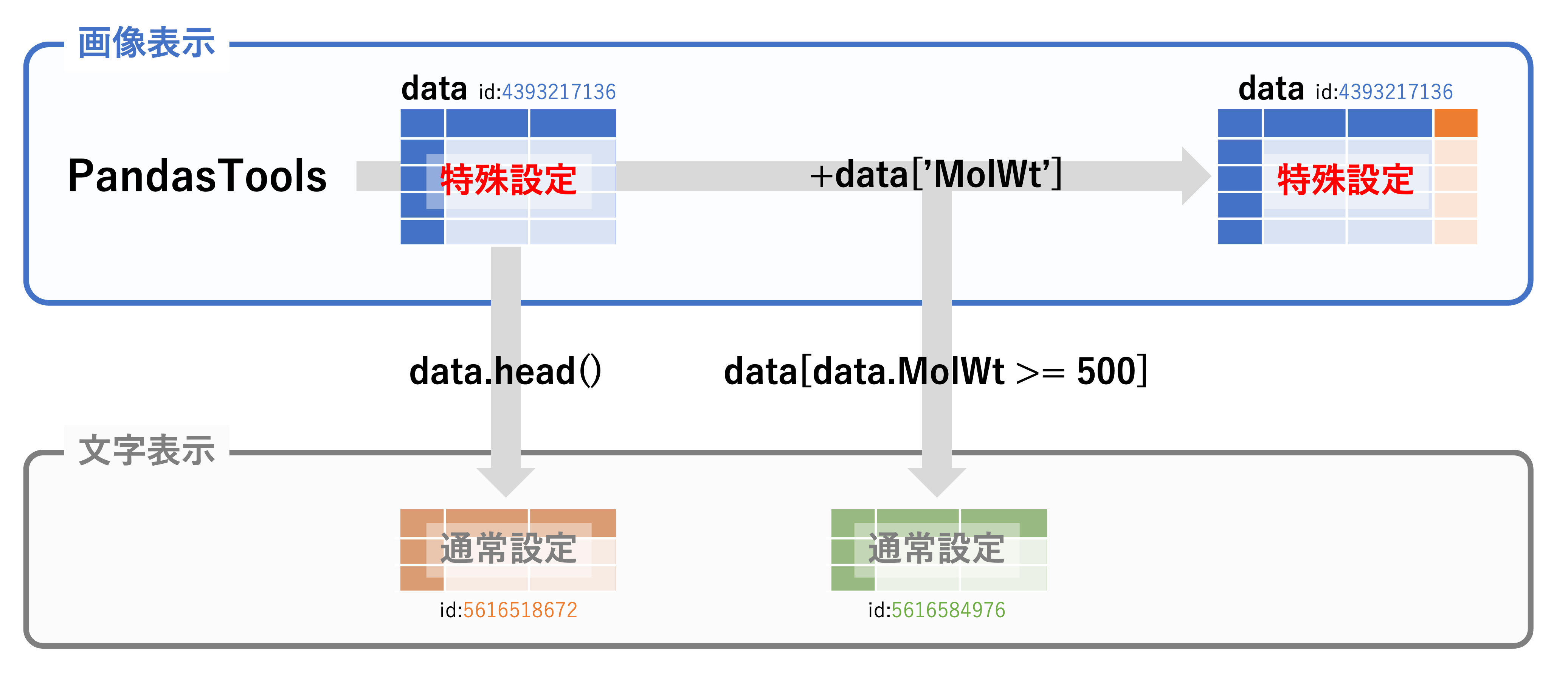
最初にPandasToolsで作成されたデータフレームインスタンス:data_0
と
.head()などで作成されたデータフレームインスタンス:data_1
は似て非なるもの
だからなのです。
どういことか、コードで説明しますと…
data = PandasTools.LoadSDF('./data/example_data.sdf') #再ロード
print(id(data), id(data.head()) # idを確認してみる
4393217136 5616518672
この通り、dataとdata.head()で違うidが振り当てられています。
記述子の絞り込みも確認
pre_id = id(data) # 一応MolWtカラムを追加する前にidを変数に代入
data['MolWt'] = data.ROMol.map(Descriptors.MolWt) # MolWt
print(f'MolWt追加前:{pre_id}, 追加後:{id(data)}, 絞り込み:{id(data[data.MolWt >= 500])}') # idを確認してみると...
MolWt追加前:4393217136, 追加後:4393217136, 絞り込み:5616584976
追加前と追加後(つまりdata)は同じid(4393217136)ですが、
絞り込みのデータフレーム(data[data.MolWt >= 500])は別のid(5616584976)が振り当てられているのがわかります。
これが画像の表示にどう関係してくるかとういうと、
PandasToolsで生成されたデータフレームインスタンスdataでは化合物が表示されるような特殊な設定(レンダリング)が使用されるので化合物が表示されますが、data.head()で生成されたインスタンスのように、一旦別のデータフレームになってしまうと通常のpandasの設定が使用されてしまい、画像が表示されなくなってしまうということです。
解決策
PandasTools.ChangeMoleculeRendering(frame)
画像表示させたいデータフレームをPandasToolsのChangeMoleculeRenderingに入れると、画像表示する設定がデータフレームに付与され、画像が表示されるようになります。
data = PandasTools.LoadSDF('./data/example_data.sdf') # 再ロード
data['MolWt'] = data.ROMol.map(Descriptors.MolWt) # MolWt
pre_id = id(data) #一応id
data_mw_500 = data[data.MolWt >= 500] # 絞り込み
mw500_id = id(data_mw_500) # 一応idも
data_mw_500 #一応表示
もちろん画像はなし
PandasTools.ChangeMoleculeRendering(data_mw_500) # ChangeMoleculeRenderingにdata_mw_500
data_mw_500 # 表示
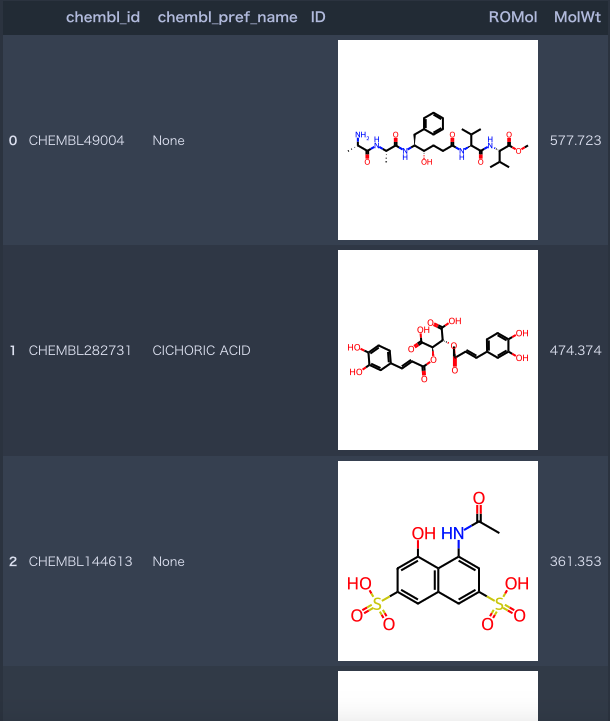
画像が表示された!!
print(pre_id, mw500_id, id(data_mw_500))
4400685360 5965143824 5965143824
data_mw_500のidを確認するとdataとは違うインスタンスになっている。つまり、data別のインスタンスdata_mw_500に対して画像表示の設定が適用されるようになったということ。
ちなみに、画像表示しているデータフレームを表示させないようにすることもできる
data = PandasTools.LoadSDF('./data/example_data.sdf') # ロード
#このままだと画像を表示してしまうので
PandasTools.ChangeMoleculeRendering(data) # ChangeMoleculeRenderingにdata
data # 表示
PandasTools.RenderImagesInAllDataFrames(bool)
PandasTools.ChangeMoleculeRendering()は任意のデータフレームのみに対して文字表現or画像表現の設定を変更するものでした。
でも、
「新しいインスタンスを生成する度にこれを実行するのは面倒臭い。」「最初から全部のデータフレームで画像が表示されるようになって欲しい!」と思いますよね。
PandasTools.RenderImagesInAllDataFrames()がおすすめ
PandasTools.RenderImagesInAllDataFrames(True)にすればノートブック内の全データフレームで画像が表示されるようになり
逆に画像が煩わしければPandasTools.RenderImagesInAllDataFrames(False)にすればノートブック内の全データフレームで画像が表示されなくなる。
data = PandasTools.LoadSDF('./data/example_data.sdf')
PandasTools.RenderImagesInAllDataFrames(True)
data.head()
data.head()でも表示された!!
注意
RenderImagesInAllDataFramesはグローバルな変更になるので、他のpythonのセッションに影響を及ぼすことがあります。その場合はChangeMoleculeRenderingを使いましょう。