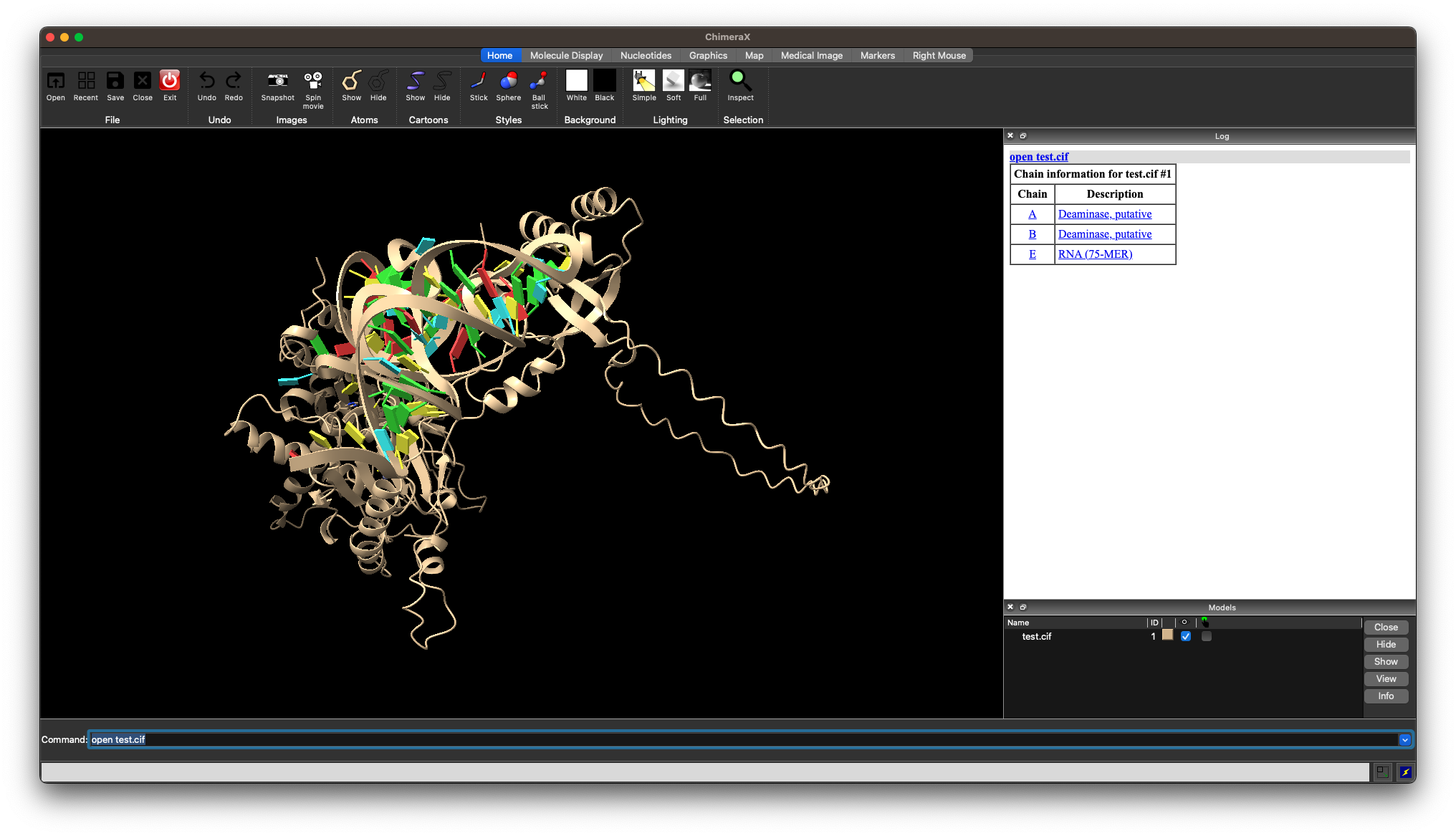
わかりずらいよね、mmCIFファイル
みなさん、こんにちは!
AlphaFoldのノーベル賞受賞で、構造バイオインフォマティクスがすごく注目されてきていますよね。
そんな注目されている構造バイオインフォを陰で支えているのが タンパク質の構造情報を収録しているファイル形式です!
近年のProtein Data Bank(PDB)、AlphaFoldDatabase(AFDB)、それからAlphaFold3の予測構造を収録しているファイル形式は mmCIF と呼ばれるファイル形式になります。
使ったことのある人はわかると思うのですが、このmmCIFファイル形式……
どこに何が書かれているのかわかりずらい!!!
特に今までPDB形式に慣れていた人やこれから構造バイオインフォを始めた人にとってはなんのこっちゃわからんでしょう。
例えば座標情報だけでも、
# PDB形式
ATOM 1 N THR A 1 67.510 52.432 1.100 1.00 53.47 N
ATOM 2 CA THR A 1 66.663 52.938 2.184 1.00 53.80 C
ATOM 3 C THR A 1 66.660 51.955 3.361 1.00 53.28 C
ATOM 4 O THR A 1 65.713 51.931 4.167 1.00 53.34 O
ATOM 5 CB THR A 1 65.183 53.192 1.684 1.00 54.32 C
ATOM 6 OG1 THR A 1 64.737 51.920 1.096 1.00 54.92 O
ATOM 7 CG2 THR A 1 65.042 54.354 0.698 1.00 54.82 C
ATOM 8 N PRO A 2 67.726 51.174 3.426 1.00 52.46 N
ATOM 9 CA PRO A 2 67.891 50.164 4.478 1.00 51.28 C
ATOM 10 C PRO A 2 67.711 50.807 5.853 1.00 49.46 C
ATOM 11 O PRO A 2 67.423 52.004 5.962 1.00 49.75 O
ATOM 12 CB PRO A 2 69.315 49.642 4.270 1.00 51.74 C
ATOM 13 CG PRO A 2 69.536 49.823 2.782 1.00 52.12 C
ATOM 14 CD PRO A 2 68.872 51.170 2.486 1.00 52.56 C
PDB形式の場合調べなくても、なんとなくどの列に何が書いてあるのか推測できます。
一方でmmCIFの場合↓
# mmCIF形式
ATOM 1 N N . THR A 1 1 ? 67.510 52.432 1.100 1.00 53.47 ? 1 THR A N 1
ATOM 2 C CA . THR A 1 1 ? 66.663 52.938 2.184 1.00 53.80 ? 1 THR A CA 1
ATOM 3 C C . THR A 1 1 ? 66.660 51.955 3.361 1.00 53.28 ? 1 THR A C 1
ATOM 4 O O . THR A 1 1 ? 65.713 51.931 4.167 1.00 53.34 ? 1 THR A O 1
ATOM 5 C CB . THR A 1 1 ? 65.183 53.192 1.684 1.00 54.32 ? 1 THR A CB 1
ATOM 6 O OG1 . THR A 1 1 ? 64.737 51.920 1.096 1.00 54.92 ? 1 THR A OG1 1
ATOM 7 C CG2 . THR A 1 1 ? 65.042 54.354 0.698 1.00 54.82 ? 1 THR A CG2 1
ATOM 8 N N . PRO A 1 2 ? 67.726 51.174 3.426 1.00 52.46 ? 2 PRO A N 1
ATOM 9 C CA . PRO A 1 2 ? 67.891 50.164 4.478 1.00 51.28 ? 2 PRO A CA 1
ATOM 10 C C . PRO A 1 2 ? 67.711 50.807 5.853 1.00 49.46 ? 2 PRO A C 1
ATOM 11 O O . PRO A 1 2 ? 67.423 52.004 5.962 1.00 49.75 ? 2 PRO A O 1
ATOM 12 C CB . PRO A 1 2 ? 69.315 49.642 4.270 1.00 51.74 ? 2 PRO A CB 1
ATOM 13 C CG . PRO A 1 2 ? 69.536 49.823 2.782 1.00 52.12 ? 2 PRO A CG 1
ATOM 14 C CD . PRO A 1 2 ? 68.872 51.170 2.486 1.00 52.56 ? 2 PRO A CD 1
なんか列多くない!?しかもおんなじような列が複数ある???となった人は少なくないはず。このようにPDBとmmCIFでは座標情報の行だけでも結構異なる点が多いです。
そしてもう一つ問題なのが、
どうやってファイルから情報を抜き出せばいいの??
ってことがあると思います。
熟練のプログラム巧者であれば、grepコマンドとかを使って抜き出したりできるかもしれませんが、私のようなプログラムを始めたての人にとっては中々厳しいですし、そのまま抜き取ったとしても、そこから解析したりグラフを書いたりするは結構面倒です。
というわけで今回は、Pythonのデータ分析でよく用いられる pandas・polars と cifのパーサーライブラリであるGEMMIを使って、cifの中身をテーブル形式にしてわかりやすく閲覧・編集していく方法について書いていこうと思います!!
使うもの・バージョン
- python 3.12
- pandas 2.2.3
- polars 1.16.0
- gemmi 0.7.0(もしくは0.6.7 下記参照)
他のバージョンでも動くと思います。
インストール方法は各ライブラリのドキュメントを確認してください(全部pipでインストールできると思います)。
どうやら、GEMMIのPython APIのドキュメントがversion 0.6.7のもののままになっているようです。本記事においては0.7.0でも動くことを確認済みですが、APIと照らし合わせて動かしたい方は0.6.7をインストールすることを推奨します。
GEMMIとは
公式docのintroductionをClaude先生を翻訳してもらいました
GEMMI - library for structural biology
GEMMIは、主にマクロ分子結晶学(MX)で使用するために開発されたライブラリとプログラム群です。以下のデータを扱うことができます:
- マクロ分子モデル(PDB、PDBx/mmCIF、mmJSONファイルの内容)
- 精密化の拘束条件(CIFファイル)
- 反射データ(MTZおよびmmCIFフォーマット)
- 3次元グリッド上のデータ(電子密度マップ、マスク、MRC/CCP4フォーマット)
- 結晶学的対称性
このライブラリの一部は、構造バイオインフォマティクス(対称性を考慮したタンパク質モデルの解析)、化学結晶学、およびCIFファイルを使用する他の分子構造科学で有用です(最速のオープンソースCIFパーサーを提供しています)。
GEMMIはオープンソース(MPL)で移植性が高く、Linux、Windows、MacOSで動作し、WebAssemblyにコンパイルすればWebブラウザ内でも動作します。C++14で書かれており、Python(3.8以上)バインディングと、部分的なCとFortran 2003インターフェースを備えています。
時折、このプロジェクトはPDBデータの可視化にも取り組んでいます。
GEMMIは、Global Phasing Ltd.とCCP4の共同プロジェクトです。Gemmi Passにちなんで名付けられました。この名前はGEneral MacroMolecular I/o(一般的なマクロ分子入出力)としても展開できます。
ソースコードリポジトリ:https://github.com/project-gemmi/gemmi
とのこと。
このgemmiは、mmCIFファイルを構文ベースで作成・編集できるだけでなく、BioPythonのBio.PDBのように座標情報からSMCRAデータ構造を構築して構造解析もできるCIFファイルに特化した構造バイオインフォマティクス向けのライブラリになります(CIFファイル以外も扱えます)。
今回は構文ベースの方を取り扱います。GEMMIで構造解析する方法もどこかで記事を書こうと思います(やる気があれば)。
ちなみに、GEMMIはC++で書かれていますが、PythonのAPIも提供されています。今回はPythonでの使用をメインに書いていきます。
GEMMIのPython APIを使ってmmCIFを読み込む
Documentクラス
早速CIFファイルを読み込んでみましょう。
構文ベースでCIFを読み込むにはgemmi.cif のread関数を使います。
from gemmi import cif
cif.read('1alk.cif')
<gemmi.cif.Document with 1 blocks (1ALK)>
読み込むと、gemmi.cif.Documentクラスのインスタンスが生成されます。ちなみに、cif.gz でも読み込むことができます。
cif.read('1alk.cif.gz')
>> <gemmi.cif.Document with 1 blocks (1ALK)> # readの場合.cif.gzもOK
# read_fileという関数もある
cif.read_file('1alk.cif')
>> <gemmi.cif.Document with 1 blocks (1ALK)>
cif.read_file('1alk.cif.gz') # read_fileは.cifのみ
>> ValueError: 1alk.cif.gz:1:0(0): expected block header (data_)
read_fileという関数もありますが、特にこだわりなければreadを使っておけば問題ないでしょう。他にもread_mmjson read_stringなどもあります。
化合物版のCIFファイルであるChemCompファイルもreadで読み込むことができます。
cif.read('ATP.cif')
>> <gemmi.cif.Document with 1 blocks (ATP)>
Block
読み込んでインスタンス化されたgemmi.cif.Documentには一つ以上のcif.gemmi.Blockオブジェクトが含まれています(通常含まれるのはーつ)。このブロックはcifファイルの先頭に書かれているdata_****の区画が一つのBlockになります。
つまり、
- Document → CIFファイル全体
- Block → 全体の内 data_****の部分
Blockが複数あるのは、ChemCompがまとまってるcomponents.cif.gzや自分でcifファイルを結合したファイルを読んだときに起こり得ます。
- Block → 全体の内 data_****の部分
start = time.time()
doc = cif.read('components.cif.gz') # 800万行あるcomponents.cif
# <gemmi.cif.Document with 44466 blocks (000, 001, 002...)>
print(f'{time.time() - start:.2f} s')
>> 3.26 s
DocumentからBlockを取り出す
Documentにも色々メソッドはありますが、情報が含まれているのはBlockの方になります。
DocumentからBlockを取り出す方法は複数ありますが、Blockが一つしかない場合はBlock.sole_blockメソッドがおすすめです。
from gemmi import cif
doc = cif.read('1alk.cif.gz')
# 1つしか無いことがわかっている場合
doc.sole_block() # solo_blockメソッド
doc[0] # __getitem__(index:int)
# ブロック名がわかっている場合(cifファイルの先頭の行に書かれている data_****の****の部分)
doc.find_block('1ALK') # find_blockメソッド
doc['1ALK'] # __getitem__(name:str)
<gemmi.cif.Block 1ALK>
また、Documentはイテレーターとしても機能します。
doc = cif.read('components.cif.gz')
for block in doc:
print(block)
<gemmi.cif.Block 000>
<gemmi.cif.Block 001>
<gemmi.cif.Block 002>
...
GEMMIでcifファイルの情報を取得する
mmCIFファイルのデータ記法
Blockから情報を抜き出す前に、mmCIFファイルにおけるデータの記法について簡単に知っておきましょう。
mmCIF内の情報は _category.item の形式(例:_entry.id )で書かれており、記述方法はkey-valueとloopの二通り存在します。
-
key-value
-
一つのカテゴリに対して一つの値しかない場合(1対1)
-
例 (
_entryなど)_entry.id 1ALK # _audit_conform.dict_name mmcif_pdbx.dic _audit_conform.dict_version 5.393 _audit_conform.dict_location http://mmcif.pdb.org/dictionaries/ascii/mmcif_pdbx.dic
-
-
loop
-
一つのカテゴリに複数の値がある場合(1対多)
-
例(
_atom_siteなど)loop_ _atom_site.group_PDB _atom_site.id _atom_site.type_symbol _atom_site.label_atom_id _atom_site.label_alt_id _atom_site.label_comp_id _atom_site.label_asym_id _atom_site.label_entity_id _atom_site.label_seq_id _atom_site.pdbx_PDB_ins_code _atom_site.Cartn_x _atom_site.Cartn_y _atom_site.Cartn_z _atom_site.occupancy _atom_site.B_iso_or_equiv _atom_site.pdbx_formal_charge _atom_site.auth_seq_id _atom_site.auth_comp_id _atom_site.auth_asym_id _atom_site.auth_atom_id _atom_site.pdbx_PDB_model_num ATOM 1 N N . THR A 1 1 ? 67.510 52.432 1.100 1.00 53.47 ? 1 THR A N 1 ATOM 2 C CA . THR A 1 1 ? 66.663 52.938 2.184 1.00 53.80 ? 1 THR A CA 1 ATOM 3 C C . THR A 1 1 ? 66.660 51.955 3.361 1.00 53.28 ? 1 THR A C 1 ATOM 4 O O . THR A 1 1 ? 65.713 51.931 4.167 1.00 53.34 ? 1 THR A O 1 ATOM 5 C CB . THR A 1 1 ? 65.183 53.192 1.684 1.00 54.32 ? 1 THR A CB 1 ATOM 6 O OG1 . THR A 1 1 ? 64.737 51.920 1.096 1.00 54.92 ? 1 THR A OG1 1 ATOM 7 C CG2 . THR A 1 1 ? 65.042 54.354 0.698 1.00 54.82 ? 1 THR A CG2 1
-
イメージとしてはkey-valueが辞書、loopがcsvなどのテーブルデータって感じですね。
Blockから情報を取り出して Dataframeに格納する
さてお待たせしました、本題の情報の抜き出しになります。
情報の抜き出しにはgemmi.Blockのget_***もしくはfind_***メソッドを使うことができます。これらには色々とメソッドがありますが、今回はget_mmcif_categoryをメインにを扱っていきます。get_mmcif_categoryはcifファイルの_categoryからitemを辞書形式で取得することができます
{
'item_a':['value_1', 'value_2', 'value_3' ...'value_n'], #_category.item_aの中身
'item_b':['value_1', 'value_2', 'value_3' ...'value_n'], #_category.item_bの中身
...
}
このままだとよくわからないと思うので、試しに1alkを例にして、座標情報が書かれている_atom_site カテゴリを取得してみましょう。
from gemmi import cif
doc = cif.read('1alk.cif.gz')
block = doc.sole_block()
names = block.get_mmcif_category_names() # block内にある _category を全部取得する
if '_atom_site.' in names:
atoms = block.get_mmcif_category('_atom_site.')
print(atoms) # 辞書
print(list(atoms.keys())) #辞書のkeys = itemの一覧
{'group_PDB': ['ATOM', 'ATOM', 'ATOM', 'ATOM', 'ATOM'...
['group_PDB', 'id', 'type_symbol', 'label_atom_id', 'label_alt_id', ...
このままではみずらい...
そこで、Pandas/PolarsのDataFrameにしてみましょう!!
atoms = block.get_mmcif_category('_atom_site')
df = pd.DataFrame(atoms)
# polarsの場合
# df = pl.DataFrame(atoms, strict=False)
df
うおおおおおおおお!!!みやすーーーーい!!!!
この通り、テーブル形式になるととてもみやすくなります。もちろん他のカテゴリも同様にテーブルにできます。
試しに、ChemCompファイルから_chem_comp (key-value記法)の情報を抜き出してみましょう!
doc = cif.read('ATP.cif')
block = doc.sole_block()
df = pd.DataFrame(block.get_mmcif_category('_chem_comp.')).T.reset_index() #わかりやすくするために転置
df.columns = ['_chem_comp', 'value']
df
みやすいですねー
全部のカテゴリをdataframeにしてprintしてみましょう。
from gemmi import cif
import polars as pl # polarsのがprintは見やすいので
doc = cif.read('1alk.cif.gz')
block = doc.sole_block()
for ctg_name in block.get_mmcif_category_names():
print(f'*****{ctg_name}*****')
print(pl.DataFrame(block.get_mmcif_category(ctg_name), strict=False))
結果(長い)
*****_entry.*****
shape: (1, 1)
┌──────┐
│ id │
│ --- │
│ str │
╞══════╡
│ 1ALK │
└──────┘
*****_audit_conform.*****
shape: (1, 3)
┌────────────────┬──────────────┬─────────────────────────────────┐
│ dict_name ┆ dict_version ┆ dict_location │
│ --- ┆ --- ┆ --- │
│ str ┆ str ┆ str │
╞════════════════╪══════════════╪═════════════════════════════════╡
│ mmcif_pdbx.dic ┆ 5.393 ┆ http://mmcif.pdb.org/dictionar… │
└────────────────┴──────────────┴─────────────────────────────────┘
*****_database_2.*****
shape: (2, 4)
┌─────────────┬───────────────┬─────────────────────────┬─────────────────────┐
│ database_id ┆ database_code ┆ pdbx_database_accession ┆ pdbx_DOI │
│ --- ┆ --- ┆ --- ┆ --- │
│ str ┆ str ┆ str ┆ str │
╞═════════════╪═══════════════╪═════════════════════════╪═════════════════════╡
│ PDB ┆ 1ALK ┆ pdb_00001alk ┆ 10.2210/pdb1alk/pdb │
│ WWPDB ┆ D_1000170967 ┆ null ┆ null │
└─────────────┴───────────────┴─────────────────────────┴─────────────────────┘
*****_pdbx_audit_revision_history.*****
shape: (4, 5)
┌─────────┬───────────────────┬────────────────┬────────────────┬───────────────┐
│ ordinal ┆ data_content_type ┆ major_revision ┆ minor_revision ┆ revision_date │
│ --- ┆ --- ┆ --- ┆ --- ┆ --- │
│ str ┆ str ┆ str ┆ str ┆ str │
╞═════════╪═══════════════════╪════════════════╪════════════════╪═══════════════╡
│ 1 ┆ Structure model ┆ 1 ┆ 0 ┆ 1994-01-31 │
│ 2 ┆ Structure model ┆ 1 ┆ 1 ┆ 2008-03-24 │
│ 3 ┆ Structure model ┆ 1 ┆ 2 ┆ 2011-07-13 │
│ 4 ┆ Structure model ┆ 1 ┆ 3 ┆ 2024-06-05 │
└─────────┴───────────────────┴────────────────┴────────────────┴───────────────┘
*****_pdbx_audit_revision_details.*****
shape: (1, 7)
┌─────────┬────────────────────┬────────────────────┬────────────┬─────────┬─────────────┬─────────┐
│ ordinal ┆ revision_ordinal ┆ data_content_type ┆ provider ┆ type ┆ description ┆ details │
│ --- ┆ --- ┆ --- ┆ --- ┆ --- ┆ --- ┆ --- │
│ str ┆ str ┆ str ┆ str ┆ str ┆ null ┆ null │
╞═════════╪════════════════════╪════════════════════╪════════════╪═════════╪═════════════╪═════════╡
│ 1 ┆ 1 ┆ Structure model ┆ repository ┆ Initial ┆ null ┆ null │
│ ┆ ┆ ┆ ┆ release ┆ ┆ │
└─────────┴────────────────────┴────────────────────┴────────────┴─────────┴─────────────┴─────────┘
*****_pdbx_audit_revision_group.*****
shape: (6, 4)
┌─────────┬──────────────────┬───────────────────┬───────────────────────────┐
│ ordinal ┆ revision_ordinal ┆ data_content_type ┆ group │
│ --- ┆ --- ┆ --- ┆ --- │
│ str ┆ str ┆ str ┆ str │
╞═════════╪══════════════════╪═══════════════════╪═══════════════════════════╡
│ 1 ┆ 2 ┆ Structure model ┆ Version format compliance │
│ 2 ┆ 3 ┆ Structure model ┆ Version format compliance │
│ 3 ┆ 4 ┆ Structure model ┆ Data collection │
│ 4 ┆ 4 ┆ Structure model ┆ Database references │
│ 5 ┆ 4 ┆ Structure model ┆ Derived calculations │
│ 6 ┆ 4 ┆ Structure model ┆ Other │
└─────────┴──────────────────┴───────────────────┴───────────────────────────┘
*****_pdbx_audit_revision_category.*****
shape: (8, 4)
┌─────────┬──────────────────┬───────────────────┬────────────────────────┐
│ ordinal ┆ revision_ordinal ┆ data_content_type ┆ category │
│ --- ┆ --- ┆ --- ┆ --- │
│ str ┆ str ┆ str ┆ str │
╞═════════╪══════════════════╪═══════════════════╪════════════════════════╡
│ 1 ┆ 4 ┆ Structure model ┆ chem_comp_atom │
│ 2 ┆ 4 ┆ Structure model ┆ chem_comp_bond │
│ 3 ┆ 4 ┆ Structure model ┆ database_2 │
│ 4 ┆ 4 ┆ Structure model ┆ pdbx_database_status │
│ 5 ┆ 4 ┆ Structure model ┆ pdbx_struct_conn_angle │
│ 6 ┆ 4 ┆ Structure model ┆ struct_conn │
│ 7 ┆ 4 ┆ Structure model ┆ struct_ref_seq_dif │
│ 8 ┆ 4 ┆ Structure model ┆ struct_site │
└─────────┴──────────────────┴───────────────────┴────────────────────────┘
*****_pdbx_audit_revision_item.*****
shape: (38, 4)
┌─────────┬──────────────────┬───────────────────┬─────────────────────────────────┐
│ ordinal ┆ revision_ordinal ┆ data_content_type ┆ item │
│ --- ┆ --- ┆ --- ┆ --- │
│ str ┆ str ┆ str ┆ str │
╞═════════╪══════════════════╪═══════════════════╪═════════════════════════════════╡
│ 1 ┆ 4 ┆ Structure model ┆ _database_2.pdbx_DOI │
│ 2 ┆ 4 ┆ Structure model ┆ _database_2.pdbx_database_acce… │
│ 3 ┆ 4 ┆ Structure model ┆ _pdbx_database_status.process_… │
│ 4 ┆ 4 ┆ Structure model ┆ _pdbx_struct_conn_angle.ptnr1_… │
│ 5 ┆ 4 ┆ Structure model ┆ _pdbx_struct_conn_angle.ptnr1_… │
│ … ┆ … ┆ … ┆ … │
│ 34 ┆ 4 ┆ Structure model ┆ _struct_conn.ptnr2_label_seq_i… │
│ 35 ┆ 4 ┆ Structure model ┆ _struct_ref_seq_dif.details │
│ 36 ┆ 4 ┆ Structure model ┆ _struct_site.pdbx_auth_asym_id │
│ 37 ┆ 4 ┆ Structure model ┆ _struct_site.pdbx_auth_comp_id │
│ 38 ┆ 4 ┆ Structure model ┆ _struct_site.pdbx_auth_seq_id │
└─────────┴──────────────────┴───────────────────┴─────────────────────────────────┘
*****_pdbx_database_status.*****
shape: (1, 12)
┌───────────┬──────────┬───────────┬───────────┬───┬───────────┬───────────┬───────────┬───────────┐
│ status_co ┆ entry_id ┆ recvd_ini ┆ deposit_s ┆ … ┆ status_co ┆ status_co ┆ status_co ┆ methods_d │
│ de ┆ --- ┆ tial_depo ┆ ite ┆ ┆ de_sf ┆ de_cs ┆ de_nmr_da ┆ evelopmen │
│ --- ┆ str ┆ sition_da ┆ --- ┆ ┆ --- ┆ --- ┆ ta ┆ t_categor │
│ str ┆ ┆ te ┆ null ┆ ┆ null ┆ null ┆ --- ┆ y │
│ ┆ ┆ --- ┆ ┆ ┆ ┆ ┆ null ┆ --- │
│ ┆ ┆ str ┆ ┆ ┆ ┆ ┆ ┆ null │
╞═══════════╪══════════╪═══════════╪═══════════╪═══╪═══════════╪═══════════╪═══════════╪═══════════╡
│ REL ┆ 1ALK ┆ 1993-03-0 ┆ null ┆ … ┆ null ┆ null ┆ null ┆ null │
│ ┆ ┆ 3 ┆ ┆ ┆ ┆ ┆ ┆ │
└───────────┴──────────┴───────────┴───────────┴───┴───────────┴───────────┴───────────┴───────────┘
*****_audit_author.*****
shape: (2, 2)
┌─────────────┬──────────────┐
│ name ┆ pdbx_ordinal │
│ --- ┆ --- │
│ str ┆ str │
╞═════════════╪══════════════╡
│ Kim, E.E. ┆ 1 │
│ Wyckoff, W. ┆ 2 │
└─────────────┴──────────────┘
*****_citation.*****
shape: (4, 14)
┌─────────┬────────────┬───────────┬───────────┬───┬───────────┬───────────┬───────────┬───────────┐
│ id ┆ title ┆ journal_a ┆ journal_v ┆ … ┆ journal_i ┆ book_publ ┆ pdbx_data ┆ pdbx_data │
│ --- ┆ --- ┆ bbrev ┆ olume ┆ ┆ d_CSD ┆ isher ┆ base_id_P ┆ base_id_D │
│ str ┆ str ┆ --- ┆ --- ┆ ┆ --- ┆ --- ┆ ubMed ┆ OI │
│ ┆ ┆ str ┆ str ┆ ┆ str ┆ null ┆ --- ┆ --- │
│ ┆ ┆ ┆ ┆ ┆ ┆ ┆ str ┆ str │
╞═════════╪════════════╪═══════════╪═══════════╪═══╪═══════════╪═══════════╪═══════════╪═══════════╡
│ primary ┆ Reaction ┆ J.Mol.Bio ┆ 218 ┆ … ┆ 0070 ┆ null ┆ 2010919 ┆ 10.1016/0 │
│ ┆ mechanism ┆ l. ┆ ┆ ┆ ┆ ┆ ┆ 022-2836( │
│ ┆ of ┆ ┆ ┆ ┆ ┆ ┆ ┆ 91)90724- │
│ ┆ alkaline… ┆ ┆ ┆ ┆ ┆ ┆ ┆ K │
│ 1 ┆ Bacillus ┆ J.Biol.Ch ┆ 266 ┆ … ┆ 0071 ┆ null ┆ null ┆ null │
│ ┆ Subtilis ┆ em. ┆ ┆ ┆ ┆ ┆ ┆ │
│ ┆ Alkaline ┆ ┆ ┆ ┆ ┆ ┆ ┆ │
│ ┆ Pho… ┆ ┆ ┆ ┆ ┆ ┆ ┆ │
│ 2 ┆ Structure ┆ Clin.Chim ┆ 186 ┆ … ┆ 0769 ┆ null ┆ null ┆ null │
│ ┆ of ┆ .Acta ┆ ┆ ┆ ┆ ┆ ┆ │
│ ┆ Alkaline ┆ ┆ ┆ ┆ ┆ ┆ ┆ │
│ ┆ Phosphat… ┆ ┆ ┆ ┆ ┆ ┆ ┆ │
│ 3 ┆ Refined ┆ J.Mol.Bio ┆ 186 ┆ … ┆ 0070 ┆ null ┆ null ┆ null │
│ ┆ Structure ┆ l. ┆ ┆ ┆ ┆ ┆ ┆ │
│ ┆ of ┆ ┆ ┆ ┆ ┆ ┆ ┆ │
│ ┆ Alkaline … ┆ ┆ ┆ ┆ ┆ ┆ ┆ │
└─────────┴────────────┴───────────┴───────────┴───┴───────────┴───────────┴───────────┴───────────┘
*****_citation_author.*****
shape: (15, 4)
┌─────────────┬──────────────────────┬─────────┬──────────────────┐
│ citation_id ┆ name ┆ ordinal ┆ identifier_ORCID │
│ --- ┆ --- ┆ --- ┆ --- │
│ str ┆ str ┆ str ┆ null │
╞═════════════╪══════════════════════╪═════════╪══════════════════╡
│ primary ┆ Kim, E.E. ┆ 1 ┆ null │
│ primary ┆ Wyckoff, H.W. ┆ 2 ┆ null │
│ 1 ┆ Hulett, F.M. ┆ 3 ┆ null │
│ 1 ┆ Kim, E.E. ┆ 4 ┆ null │
│ 1 ┆ Bookstein, C. ┆ 5 ┆ null │
│ … ┆ … ┆ … ┆ … │
│ 3 ┆ Sowadski, J.M. ┆ 11 ┆ null │
│ 3 ┆ Handschumacher, M.D. ┆ 12 ┆ null │
│ 3 ┆ Murthy, H.M.K. ┆ 13 ┆ null │
│ 3 ┆ Foster, B.A. ┆ 14 ┆ null │
│ 3 ┆ Wyckoff, H.W. ┆ 15 ┆ null │
└─────────────┴──────────────────────┴─────────┴──────────────────┘
*****_entity.*****
shape: (5, 10)
┌─────┬─────────────┬────────────┬─────────────┬───┬─────────┬─────────────┬─────────────┬─────────┐
│ id ┆ type ┆ src_method ┆ pdbx_descri ┆ … ┆ pdbx_ec ┆ pdbx_mutati ┆ pdbx_fragme ┆ details │
│ --- ┆ --- ┆ --- ┆ ption ┆ ┆ --- ┆ on ┆ nt ┆ --- │
│ str ┆ str ┆ str ┆ --- ┆ ┆ str ┆ --- ┆ --- ┆ null │
│ ┆ ┆ ┆ str ┆ ┆ ┆ null ┆ null ┆ │
╞═════╪═════════════╪════════════╪═════════════╪═══╪═════════╪═════════════╪═════════════╪═════════╡
│ 1 ┆ polymer ┆ man ┆ ALKALINE ┆ … ┆ 3.1.3.1 ┆ null ┆ null ┆ null │
│ ┆ ┆ ┆ PHOSPHATASE ┆ ┆ ┆ ┆ ┆ │
│ 2 ┆ non-polymer ┆ syn ┆ ZINC ION ┆ … ┆ null ┆ null ┆ null ┆ null │
│ 3 ┆ non-polymer ┆ syn ┆ MAGNESIUM ┆ … ┆ null ┆ null ┆ null ┆ null │
│ ┆ ┆ ┆ ION ┆ ┆ ┆ ┆ ┆ │
│ 4 ┆ non-polymer ┆ syn ┆ PHOSPHATE ┆ … ┆ null ┆ null ┆ null ┆ null │
│ ┆ ┆ ┆ ION ┆ ┆ ┆ ┆ ┆ │
│ 5 ┆ water ┆ nat ┆ water ┆ … ┆ null ┆ null ┆ null ┆ null │
└─────┴─────────────┴────────────┴─────────────┴───┴─────────┴─────────────┴─────────────┴─────────┘
*****_entity_poly.*****
shape: (1, 8)
┌───────────┬────────────┬────────────┬────────────┬───────────┬───────────┬───────────┬───────────┐
│ entity_id ┆ type ┆ nstd_linka ┆ nstd_monom ┆ pdbx_seq_ ┆ pdbx_seq_ ┆ pdbx_stra ┆ pdbx_targ │
│ --- ┆ --- ┆ ge ┆ er ┆ one_lette ┆ one_lette ┆ nd_id ┆ et_identi │
│ str ┆ str ┆ --- ┆ --- ┆ r_code ┆ r_code_ca ┆ --- ┆ fier │
│ ┆ ┆ str ┆ str ┆ --- ┆ n ┆ str ┆ --- │
│ ┆ ┆ ┆ ┆ str ┆ --- ┆ ┆ null │
│ ┆ ┆ ┆ ┆ ┆ str ┆ ┆ │
╞═══════════╪════════════╪════════════╪════════════╪═══════════╪═══════════╪═══════════╪═══════════╡
│ 1 ┆ polypeptid ┆ no ┆ no ┆ TPEMPVLEN ┆ TPEMPVLEN ┆ A,B ┆ null │
│ ┆ e(L) ┆ ┆ ┆ RAAQGNITA ┆ RAAQGNITA ┆ ┆ │
│ ┆ ┆ ┆ ┆ PGGARRLTG ┆ PGGARRLTG ┆ ┆ │
│ ┆ ┆ ┆ ┆ DQT… ┆ DQT… ┆ ┆ │
└───────────┴────────────┴────────────┴────────────┴───────────┴───────────┴───────────┴───────────┘
*****_pdbx_entity_nonpoly.*****
shape: (4, 3)
┌───────────┬───────────────┬─────────┐
│ entity_id ┆ name ┆ comp_id │
│ --- ┆ --- ┆ --- │
│ str ┆ str ┆ str │
╞═══════════╪═══════════════╪═════════╡
│ 2 ┆ ZINC ION ┆ ZN │
│ 3 ┆ MAGNESIUM ION ┆ MG │
│ 4 ┆ PHOSPHATE ION ┆ PO4 │
│ 5 ┆ water ┆ HOH │
└───────────┴───────────────┴─────────┘
*****_entity_poly_seq.*****
shape: (449, 4)
┌───────────┬─────┬────────┬────────┐
│ entity_id ┆ num ┆ mon_id ┆ hetero │
│ --- ┆ --- ┆ --- ┆ --- │
│ str ┆ str ┆ str ┆ str │
╞═══════════╪═════╪════════╪════════╡
│ 1 ┆ 1 ┆ THR ┆ n │
│ 1 ┆ 2 ┆ PRO ┆ n │
│ 1 ┆ 3 ┆ GLU ┆ n │
│ 1 ┆ 4 ┆ MET ┆ n │
│ 1 ┆ 5 ┆ PRO ┆ n │
│ … ┆ … ┆ … ┆ … │
│ 1 ┆ 445 ┆ ALA ┆ n │
│ 1 ┆ 446 ┆ LEU ┆ n │
│ 1 ┆ 447 ┆ GLY ┆ n │
│ 1 ┆ 448 ┆ LEU ┆ n │
│ 1 ┆ 449 ┆ LYS ┆ n │
└───────────┴─────┴────────┴────────┘
*****_entity_src_gen.*****
shape: (1, 48)
┌───────────┬───────────┬───────────┬───────────┬───┬───────────┬───────────┬───────────┬──────────┐
│ entity_id ┆ pdbx_src_ ┆ pdbx_alt_ ┆ pdbx_seq_ ┆ … ┆ expressio ┆ plasmid_n ┆ plasmid_d ┆ pdbx_des │
│ --- ┆ id ┆ source_fl ┆ type ┆ ┆ n_system_ ┆ ame ┆ etails ┆ cription │
│ str ┆ --- ┆ ag ┆ --- ┆ ┆ id ┆ --- ┆ --- ┆ --- │
│ ┆ str ┆ --- ┆ null ┆ ┆ --- ┆ null ┆ null ┆ null │
│ ┆ ┆ str ┆ ┆ ┆ null ┆ ┆ ┆ │
╞═══════════╪═══════════╪═══════════╪═══════════╪═══╪═══════════╪═══════════╪═══════════╪══════════╡
│ 1 ┆ 1 ┆ sample ┆ null ┆ … ┆ null ┆ null ┆ null ┆ null │
└───────────┴───────────┴───────────┴───────────┴───┴───────────┴───────────┴───────────┴──────────┘
*****_chem_comp.*****
shape: (24, 7)
┌─────┬───────────────┬───────────────┬───────────────┬──────────────┬──────────────┬──────────────┐
│ id ┆ type ┆ mon_nstd_flag ┆ name ┆ pdbx_synonym ┆ formula ┆ formula_weig │
│ --- ┆ --- ┆ --- ┆ --- ┆ s ┆ --- ┆ ht │
│ str ┆ str ┆ str ┆ str ┆ --- ┆ str ┆ --- │
│ ┆ ┆ ┆ ┆ null ┆ ┆ str │
╞═════╪═══════════════╪═══════════════╪═══════════════╪══════════════╪══════════════╪══════════════╡
│ ALA ┆ L-peptide ┆ y ┆ ALANINE ┆ null ┆ C3 H7 N O2 ┆ 89.093 │
│ ┆ linking ┆ ┆ ┆ ┆ ┆ │
│ ARG ┆ L-peptide ┆ y ┆ ARGININE ┆ null ┆ C6 H15 N4 O2 ┆ 175.209 │
│ ┆ linking ┆ ┆ ┆ ┆ 1 ┆ │
│ ASN ┆ L-peptide ┆ y ┆ ASPARAGINE ┆ null ┆ C4 H8 N2 O3 ┆ 132.118 │
│ ┆ linking ┆ ┆ ┆ ┆ ┆ │
│ ASP ┆ L-peptide ┆ y ┆ ASPARTIC ACID ┆ null ┆ C4 H7 N O4 ┆ 133.103 │
│ ┆ linking ┆ ┆ ┆ ┆ ┆ │
│ CYS ┆ L-peptide ┆ y ┆ CYSTEINE ┆ null ┆ C3 H7 N O2 S ┆ 121.158 │
│ ┆ linking ┆ ┆ ┆ ┆ ┆ │
│ … ┆ … ┆ … ┆ … ┆ … ┆ … ┆ … │
│ THR ┆ L-peptide ┆ y ┆ THREONINE ┆ null ┆ C4 H9 N O3 ┆ 119.119 │
│ ┆ linking ┆ ┆ ┆ ┆ ┆ │
│ TRP ┆ L-peptide ┆ y ┆ TRYPTOPHAN ┆ null ┆ C11 H12 N2 ┆ 204.225 │
│ ┆ linking ┆ ┆ ┆ ┆ O2 ┆ │
│ TYR ┆ L-peptide ┆ y ┆ TYROSINE ┆ null ┆ C9 H11 N O3 ┆ 181.189 │
│ ┆ linking ┆ ┆ ┆ ┆ ┆ │
│ VAL ┆ L-peptide ┆ y ┆ VALINE ┆ null ┆ C5 H11 N O2 ┆ 117.146 │
│ ┆ linking ┆ ┆ ┆ ┆ ┆ │
│ ZN ┆ non-polymer ┆ false ┆ ZINC ION ┆ null ┆ Zn 2 ┆ 65.409 │
└─────┴───────────────┴───────────────┴───────────────┴──────────────┴──────────────┴──────────────┘
*****_pdbx_poly_seq_scheme.*****
shape: (898, 12)
┌─────────┬───────────┬────────┬────────┬───┬─────────────┬───────────────┬──────────────┬────────┐
│ asym_id ┆ entity_id ┆ seq_id ┆ mon_id ┆ … ┆ auth_mon_id ┆ pdb_strand_id ┆ pdb_ins_code ┆ hetero │
│ --- ┆ --- ┆ --- ┆ --- ┆ ┆ --- ┆ --- ┆ --- ┆ --- │
│ str ┆ str ┆ str ┆ str ┆ ┆ str ┆ str ┆ bool ┆ str │
╞═════════╪═══════════╪════════╪════════╪═══╪═════════════╪═══════════════╪══════════════╪════════╡
│ A ┆ 1 ┆ 1 ┆ THR ┆ … ┆ THR ┆ A ┆ false ┆ n │
│ A ┆ 1 ┆ 2 ┆ PRO ┆ … ┆ PRO ┆ A ┆ false ┆ n │
│ A ┆ 1 ┆ 3 ┆ GLU ┆ … ┆ GLU ┆ A ┆ false ┆ n │
│ A ┆ 1 ┆ 4 ┆ MET ┆ … ┆ MET ┆ A ┆ false ┆ n │
│ A ┆ 1 ┆ 5 ┆ PRO ┆ … ┆ PRO ┆ A ┆ false ┆ n │
│ … ┆ … ┆ … ┆ … ┆ … ┆ … ┆ … ┆ … ┆ … │
│ B ┆ 1 ┆ 445 ┆ ALA ┆ … ┆ ALA ┆ B ┆ false ┆ n │
│ B ┆ 1 ┆ 446 ┆ LEU ┆ … ┆ LEU ┆ B ┆ false ┆ n │
│ B ┆ 1 ┆ 447 ┆ GLY ┆ … ┆ GLY ┆ B ┆ false ┆ n │
│ B ┆ 1 ┆ 448 ┆ LEU ┆ … ┆ LEU ┆ B ┆ false ┆ n │
│ B ┆ 1 ┆ 449 ┆ LYS ┆ … ┆ LYS ┆ B ┆ false ┆ n │
└─────────┴───────────┴────────┴────────┴───┴─────────────┴───────────────┴──────────────┴────────┘
*****_pdbx_nonpoly_scheme.*****
shape: (14, 10)
┌─────────┬───────────┬────────┬────────────┬───┬────────────┬────────────┬────────────┬───────────┐
│ asym_id ┆ entity_id ┆ mon_id ┆ ndb_seq_nu ┆ … ┆ pdb_mon_id ┆ auth_mon_i ┆ pdb_strand ┆ pdb_ins_c │
│ --- ┆ --- ┆ --- ┆ m ┆ ┆ --- ┆ d ┆ _id ┆ ode │
│ str ┆ str ┆ str ┆ --- ┆ ┆ str ┆ --- ┆ --- ┆ --- │
│ ┆ ┆ ┆ str ┆ ┆ ┆ str ┆ str ┆ bool │
╞═════════╪═══════════╪════════╪════════════╪═══╪════════════╪════════════╪════════════╪═══════════╡
│ C ┆ 2 ┆ ZN ┆ 1 ┆ … ┆ ZN ┆ ZN ┆ A ┆ false │
│ D ┆ 2 ┆ ZN ┆ 1 ┆ … ┆ ZN ┆ ZN ┆ A ┆ false │
│ E ┆ 3 ┆ MG ┆ 1 ┆ … ┆ MG ┆ MG ┆ A ┆ false │
│ F ┆ 4 ┆ PO4 ┆ 1 ┆ … ┆ PO4 ┆ PO4 ┆ A ┆ false │
│ G ┆ 2 ┆ ZN ┆ 1 ┆ … ┆ ZN ┆ ZN ┆ B ┆ false │
│ … ┆ … ┆ … ┆ … ┆ … ┆ … ┆ … ┆ … ┆ … │
│ K ┆ 5 ┆ HOH ┆ 2 ┆ … ┆ HOH ┆ HOH ┆ A ┆ false │
│ K ┆ 5 ┆ HOH ┆ 3 ┆ … ┆ HOH ┆ HOH ┆ A ┆ false │
│ L ┆ 5 ┆ HOH ┆ 1 ┆ … ┆ HOH ┆ HOH ┆ B ┆ false │
│ L ┆ 5 ┆ HOH ┆ 2 ┆ … ┆ HOH ┆ HOH ┆ B ┆ false │
│ L ┆ 5 ┆ HOH ┆ 3 ┆ … ┆ HOH ┆ HOH ┆ B ┆ false │
└─────────┴───────────┴────────┴────────────┴───┴────────────┴────────────┴────────────┴───────────┘
*****_software.*****
shape: (4, 5)
┌────────┬────────────────┬─────────┬─────────────┬──────────────┐
│ name ┆ classification ┆ version ┆ citation_id ┆ pdbx_ordinal │
│ --- ┆ --- ┆ --- ┆ --- ┆ --- │
│ str ┆ str ┆ bool ┆ null ┆ str │
╞════════╪════════════════╪═════════╪═════════════╪══════════════╡
│ X-PLOR ┆ model building ┆ false ┆ null ┆ 1 │
│ PROFFT ┆ refinement ┆ false ┆ null ┆ 2 │
│ X-PLOR ┆ refinement ┆ false ┆ null ┆ 3 │
│ X-PLOR ┆ phasing ┆ false ┆ null ┆ 4 │
└────────┴────────────────┴─────────┴─────────────┴──────────────┘
*****_cell.*****
shape: (1, 9)
┌──────────┬──────────┬──────────┬──────────┬───┬────────────┬─────────────┬───────┬───────────────┐
│ entry_id ┆ length_a ┆ length_b ┆ length_c ┆ … ┆ angle_beta ┆ angle_gamma ┆ Z_PDB ┆ pdbx_unique_a │
│ --- ┆ --- ┆ --- ┆ --- ┆ ┆ --- ┆ --- ┆ --- ┆ xis │
│ str ┆ str ┆ str ┆ str ┆ ┆ str ┆ str ┆ str ┆ --- │
│ ┆ ┆ ┆ ┆ ┆ ┆ ┆ ┆ null │
╞══════════╪══════════╪══════════╪══════════╪═══╪════════════╪═════════════╪═══════╪═══════════════╡
│ 1ALK ┆ 195.300 ┆ 167.400 ┆ 76.700 ┆ … ┆ 90.00 ┆ 90.00 ┆ 16 ┆ null │
└──────────┴──────────┴──────────┴──────────┴───┴────────────┴─────────────┴───────┴───────────────┘
*****_symmetry.*****
shape: (1, 5)
┌──────────┬──────────────────────┬─────────────────────────────┬──────────────┬───────────────────┐
│ entry_id ┆ space_group_name_H-M ┆ pdbx_full_space_group_name_ ┆ cell_setting ┆ Int_Tables_number │
│ --- ┆ --- ┆ H-M ┆ --- ┆ --- │
│ str ┆ str ┆ --- ┆ null ┆ str │
│ ┆ ┆ null ┆ ┆ │
╞══════════╪══════════════════════╪═════════════════════════════╪══════════════╪═══════════════════╡
│ 1ALK ┆ I 2 2 2 ┆ null ┆ null ┆ 23 │
└──────────┴──────────────────────┴─────────────────────────────┴──────────────┴───────────────────┘
*****_exptl.*****
shape: (1, 3)
┌──────────┬───────────────────┬─────────────────┐
│ entry_id ┆ method ┆ crystals_number │
│ --- ┆ --- ┆ --- │
│ str ┆ str ┆ null │
╞══════════╪═══════════════════╪═════════════════╡
│ 1ALK ┆ X-RAY DIFFRACTION ┆ null │
└──────────┴───────────────────┴─────────────────┘
*****_exptl_crystal.*****
shape: (1, 5)
┌─────┬──────────────┬──────────────────┬─────────────────────┬─────────────┐
│ id ┆ density_meas ┆ density_Matthews ┆ density_percent_sol ┆ description │
│ --- ┆ --- ┆ --- ┆ --- ┆ --- │
│ str ┆ null ┆ str ┆ str ┆ null │
╞═════╪══════════════╪══════════════════╪═════════════════════╪═════════════╡
│ 1 ┆ null ┆ 3.33 ┆ 63.02 ┆ null │
└─────┴──────────────┴──────────────────┴─────────────────────┴─────────────┘
*****_diffrn.*****
shape: (1, 4)
┌─────┬──────────────┬──────────────────────┬────────────┐
│ id ┆ ambient_temp ┆ ambient_temp_details ┆ crystal_id │
│ --- ┆ --- ┆ --- ┆ --- │
│ str ┆ null ┆ null ┆ str │
╞═════╪══════════════╪══════════════════════╪════════════╡
│ 1 ┆ null ┆ null ┆ 1 │
└─────┴──────────────┴──────────────────────┴────────────┘
*****_diffrn_radiation.*****
shape: (1, 6)
┌───────────┬───────────────┬──────────────────┬───────────────┬─────────────────┬─────────────────┐
│ diffrn_id ┆ wavelength_id ┆ pdbx_monochromat ┆ monochromator ┆ pdbx_diffrn_pro ┆ pdbx_scattering │
│ --- ┆ --- ┆ ic_or_laue_m_l ┆ --- ┆ tocol ┆ _type │
│ str ┆ str ┆ --- ┆ null ┆ --- ┆ --- │
│ ┆ ┆ null ┆ ┆ null ┆ str │
╞═══════════╪═══════════════╪══════════════════╪═══════════════╪═════════════════╪═════════════════╡
│ 1 ┆ 1 ┆ null ┆ null ┆ null ┆ x-ray │
└───────────┴───────────────┴──────────────────┴───────────────┴─────────────────┴─────────────────┘
*****_diffrn_radiation_wavelength.*****
shape: (1, 3)
┌─────┬────────────┬─────┐
│ id ┆ wavelength ┆ wt │
│ --- ┆ --- ┆ --- │
│ str ┆ bool ┆ str │
╞═════╪════════════╪═════╡
│ 1 ┆ false ┆ 1.0 │
└─────┴────────────┴─────┘
*****_refine.*****
shape: (1, 58)
┌──────────┬───────────┬───────────┬───────────┬───┬───────────┬───────────┬───────────┬───────────┐
│ entry_id ┆ ls_number ┆ ls_number ┆ pdbx_ls_s ┆ … ┆ overall_S ┆ pdbx_over ┆ pdbx_over ┆ pdbx_over │
│ --- ┆ _reflns_o ┆ _reflns_a ┆ igma_I ┆ ┆ U_R_Cruic ┆ all_SU_R_ ┆ all_SU_R_ ┆ all_SU_R_ │
│ str ┆ bs ┆ ll ┆ --- ┆ ┆ kshank_DP ┆ free_Crui ┆ Blow_DPI ┆ free_Blow │
│ ┆ --- ┆ --- ┆ null ┆ ┆ I ┆ cks… ┆ --- ┆ _DP… │
│ ┆ null ┆ null ┆ ┆ ┆ --- ┆ --- ┆ null ┆ --- │
│ ┆ ┆ ┆ ┆ ┆ null ┆ null ┆ ┆ null │
╞══════════╪═══════════╪═══════════╪═══════════╪═══╪═══════════╪═══════════╪═══════════╪═══════════╡
│ 1ALK ┆ null ┆ null ┆ null ┆ … ┆ null ┆ null ┆ null ┆ null │
└──────────┴───────────┴───────────┴───────────┴───┴───────────┴───────────┴───────────┴───────────┘
*****_refine_hist.*****
shape: (1, 9)
┌───────────┬──────────┬───────────┬───────────┬───┬───────────┬───────────┬───────────┬───────────┐
│ pdbx_refi ┆ cycle_id ┆ pdbx_numb ┆ pdbx_numb ┆ … ┆ number_at ┆ number_at ┆ d_res_hig ┆ d_res_low │
│ ne_id ┆ --- ┆ er_atoms_ ┆ er_atoms_ ┆ ┆ oms_solve ┆ oms_total ┆ h ┆ --- │
│ --- ┆ str ┆ protein ┆ nucleic_a ┆ ┆ nt ┆ --- ┆ --- ┆ bool │
│ str ┆ ┆ --- ┆ cid ┆ ┆ --- ┆ str ┆ str ┆ │
│ ┆ ┆ str ┆ --- ┆ ┆ str ┆ ┆ ┆ │
│ ┆ ┆ ┆ str ┆ ┆ ┆ ┆ ┆ │
╞═══════════╪══════════╪═══════════╪═══════════╪═══╪═══════════╪═══════════╪═══════════╪═══════════╡
│ X-RAY DIF ┆ LAST ┆ 6608 ┆ 0 ┆ … ┆ 6 ┆ 6630 ┆ 2.0 ┆ false │
│ FRACTION ┆ ┆ ┆ ┆ ┆ ┆ ┆ ┆ │
└───────────┴──────────┴───────────┴───────────┴───┴───────────┴───────────┴───────────┴───────────┘
*****_database_PDB_matrix.*****
shape: (1, 13)
┌──────────┬───────────┬───────────┬───────────┬───┬───────────┬───────────┬───────────┬───────────┐
│ entry_id ┆ origx[1][ ┆ origx[1][ ┆ origx[1][ ┆ … ┆ origx[3][ ┆ origx_vec ┆ origx_vec ┆ origx_vec │
│ --- ┆ 1] ┆ 2] ┆ 3] ┆ ┆ 3] ┆ tor[1] ┆ tor[2] ┆ tor[3] │
│ str ┆ --- ┆ --- ┆ --- ┆ ┆ --- ┆ --- ┆ --- ┆ --- │
│ ┆ str ┆ str ┆ str ┆ ┆ str ┆ str ┆ str ┆ str │
╞══════════╪═══════════╪═══════════╪═══════════╪═══╪═══════════╪═══════════╪═══════════╪═══════════╡
│ 1ALK ┆ 1.000000 ┆ 0.000000 ┆ 0.000000 ┆ … ┆ 1.000000 ┆ 0.00000 ┆ 0.00000 ┆ 0.00000 │
└──────────┴───────────┴───────────┴───────────┴───┴───────────┴───────────┴───────────┴───────────┘
*****_struct.*****
shape: (1, 5)
┌──────────┬────────────────────────┬────────────────────┬────────────────┬────────────────────────┐
│ entry_id ┆ title ┆ pdbx_model_details ┆ pdbx_CASP_flag ┆ pdbx_model_type_detail │
│ --- ┆ --- ┆ --- ┆ --- ┆ s │
│ str ┆ str ┆ null ┆ null ┆ --- │
│ ┆ ┆ ┆ ┆ null │
╞══════════╪════════════════════════╪════════════════════╪════════════════╪════════════════════════╡
│ 1ALK ┆ REACTION MECHANISM OF ┆ null ┆ null ┆ null │
│ ┆ ALKALINE… ┆ ┆ ┆ │
└──────────┴────────────────────────┴────────────────────┴────────────────┴────────────────────────┘
*****_struct_keywords.*****
shape: (1, 3)
┌──────────┬──────────────────────┬──────────────────────┐
│ entry_id ┆ pdbx_keywords ┆ text │
│ --- ┆ --- ┆ --- │
│ str ┆ str ┆ str │
╞══════════╪══════════════════════╪══════════════════════╡
│ 1ALK ┆ ALKALINE PHOSPHATASE ┆ ALKALINE PHOSPHATASE │
└──────────┴──────────────────────┴──────────────────────┘
*****_struct_asym.*****
shape: (12, 5)
┌─────┬─────────────────────────────┬───────────────┬───────────┬─────────┐
│ id ┆ pdbx_blank_PDB_chainid_flag ┆ pdbx_modified ┆ entity_id ┆ details │
│ --- ┆ --- ┆ --- ┆ --- ┆ --- │
│ str ┆ str ┆ str ┆ str ┆ null │
╞═════╪═════════════════════════════╪═══════════════╪═══════════╪═════════╡
│ A ┆ N ┆ N ┆ 1 ┆ null │
│ B ┆ N ┆ N ┆ 1 ┆ null │
│ C ┆ N ┆ N ┆ 2 ┆ null │
│ D ┆ N ┆ N ┆ 2 ┆ null │
│ E ┆ N ┆ N ┆ 3 ┆ null │
│ … ┆ … ┆ … ┆ … ┆ … │
│ H ┆ N ┆ N ┆ 2 ┆ null │
│ I ┆ N ┆ N ┆ 3 ┆ null │
│ J ┆ N ┆ N ┆ 4 ┆ null │
│ K ┆ N ┆ N ┆ 5 ┆ null │
│ L ┆ N ┆ N ┆ 5 ┆ null │
└─────┴─────────────────────────────┴───────────────┴───────────┴─────────┘
*****_struct_ref.*****
shape: (1, 8)
┌─────┬─────────┬───────────┬───────────┬──────────────┬──────────────┬──────────────┬─────────────┐
│ id ┆ db_name ┆ db_code ┆ entity_id ┆ pdbx_db_acce ┆ pdbx_align_b ┆ pdbx_seq_one ┆ pdbx_db_iso │
│ --- ┆ --- ┆ --- ┆ --- ┆ ssion ┆ egin ┆ _letter_code ┆ form │
│ str ┆ str ┆ str ┆ str ┆ --- ┆ --- ┆ --- ┆ --- │
│ ┆ ┆ ┆ ┆ str ┆ str ┆ str ┆ null │
╞═════╪═════════╪═══════════╪═══════════╪══════════════╪══════════════╪══════════════╪═════════════╡
│ 1 ┆ UNP ┆ PPB_ECOLI ┆ 1 ┆ P00634 ┆ 1 ┆ MKQSTIALALLP ┆ null │
│ ┆ ┆ ┆ ┆ ┆ ┆ LLFTPVTKARTP ┆ │
│ ┆ ┆ ┆ ┆ ┆ ┆ EMPVLE… ┆ │
└─────┴─────────┴───────────┴───────────┴──────────────┴──────────────┴──────────────┴─────────────┘
*****_struct_ref_seq.*****
shape: (2, 15)
┌──────────┬────────┬────────────┬────────────┬───┬────────────┬───────────┬───────────┬───────────┐
│ align_id ┆ ref_id ┆ pdbx_PDB_i ┆ pdbx_stran ┆ … ┆ db_align_e ┆ pdbx_db_a ┆ pdbx_auth ┆ pdbx_auth │
│ --- ┆ --- ┆ d_code ┆ d_id ┆ ┆ nd ┆ lign_end_ ┆ _seq_alig ┆ _seq_alig │
│ str ┆ str ┆ --- ┆ --- ┆ ┆ --- ┆ ins_code ┆ n_beg ┆ n_end │
│ ┆ ┆ str ┆ str ┆ ┆ str ┆ --- ┆ --- ┆ --- │
│ ┆ ┆ ┆ ┆ ┆ ┆ null ┆ str ┆ str │
╞══════════╪════════╪════════════╪════════════╪═══╪════════════╪═══════════╪═══════════╪═══════════╡
│ 1 ┆ 1 ┆ 1ALK ┆ A ┆ … ┆ 471 ┆ null ┆ 1 ┆ 449 │
│ 2 ┆ 1 ┆ 1ALK ┆ B ┆ … ┆ 471 ┆ null ┆ 1 ┆ 449 │
└──────────┴────────┴────────────┴────────────┴───┴────────────┴───────────┴───────────┴───────────┘
*****_struct_ref_seq_dif.*****
shape: (10, 13)
┌──────────┬────────────┬────────┬────────────┬───┬────────────┬──────────┬────────────┬───────────┐
│ align_id ┆ pdbx_pdb_i ┆ mon_id ┆ pdbx_pdb_s ┆ … ┆ pdbx_seq_d ┆ details ┆ pdbx_auth_ ┆ pdbx_ordi │
│ --- ┆ d_code ┆ --- ┆ trand_id ┆ ┆ b_seq_num ┆ --- ┆ seq_num ┆ nal │
│ str ┆ --- ┆ str ┆ --- ┆ ┆ --- ┆ str ┆ --- ┆ --- │
│ ┆ str ┆ ┆ str ┆ ┆ str ┆ ┆ str ┆ str │
╞══════════╪════════════╪════════╪════════════╪═══╪════════════╪══════════╪════════════╪═══════════╡
│ 1 ┆ 1ALK ┆ ASN ┆ A ┆ … ┆ 37 ┆ conflict ┆ 15 ┆ 1 │
│ 1 ┆ 1ALK ┆ ASN ┆ A ┆ … ┆ 57 ┆ conflict ┆ 35 ┆ 2 │
│ 1 ┆ 1ALK ┆ GLN ┆ A ┆ … ┆ 198 ┆ conflict ┆ 176 ┆ 3 │
│ 1 ┆ 1ALK ┆ GLU ┆ A ┆ … ┆ 250 ┆ conflict ┆ 228 ┆ 4 │
│ 1 ┆ 1ALK ┆ GLU ┆ A ┆ … ┆ 252 ┆ conflict ┆ 230 ┆ 5 │
│ 2 ┆ 1ALK ┆ ASN ┆ B ┆ … ┆ 37 ┆ conflict ┆ 15 ┆ 6 │
│ 2 ┆ 1ALK ┆ ASN ┆ B ┆ … ┆ 57 ┆ conflict ┆ 35 ┆ 7 │
│ 2 ┆ 1ALK ┆ GLN ┆ B ┆ … ┆ 198 ┆ conflict ┆ 176 ┆ 8 │
│ 2 ┆ 1ALK ┆ GLU ┆ B ┆ … ┆ 250 ┆ conflict ┆ 228 ┆ 9 │
│ 2 ┆ 1ALK ┆ GLU ┆ B ┆ … ┆ 252 ┆ conflict ┆ 230 ┆ 10 │
└──────────┴────────────┴────────┴────────────┴───┴────────────┴──────────┴────────────┴───────────┘
*****_pdbx_struct_assembly.*****
shape: (1, 5)
┌─────┬─────────────────────────────────┬────────────────┬────────────────────┬──────────────────┐
│ id ┆ details ┆ method_details ┆ oligomeric_details ┆ oligomeric_count │
│ --- ┆ --- ┆ --- ┆ --- ┆ --- │
│ str ┆ str ┆ str ┆ str ┆ str │
╞═════╪═════════════════════════════════╪════════════════╪════════════════════╪══════════════════╡
│ 1 ┆ author_and_software_defined_as… ┆ PISA ┆ dimeric ┆ 2 │
└─────┴─────────────────────────────────┴────────────────┴────────────────────┴──────────────────┘
*****_pdbx_struct_assembly_prop.*****
shape: (3, 4)
┌─────────┬────────────┬───────┬─────────┐
│ biol_id ┆ type ┆ value ┆ details │
│ --- ┆ --- ┆ --- ┆ --- │
│ str ┆ str ┆ str ┆ null │
╞═════════╪════════════╪═══════╪═════════╡
│ 1 ┆ ABSA (A^2) ┆ 8720 ┆ null │
│ 1 ┆ MORE ┆ -205 ┆ null │
│ 1 ┆ SSA (A^2) ┆ 27920 ┆ null │
└─────────┴────────────┴───────┴─────────┘
*****_pdbx_struct_assembly_gen.*****
shape: (1, 3)
┌─────────────┬─────────────────┬─────────────────────────┐
│ assembly_id ┆ oper_expression ┆ asym_id_list │
│ --- ┆ --- ┆ --- │
│ str ┆ str ┆ str │
╞═════════════╪═════════════════╪═════════════════════════╡
│ 1 ┆ 1 ┆ A,B,C,D,E,F,G,H,I,J,K,L │
└─────────────┴─────────────────┴─────────────────────────┘
*****_pdbx_struct_oper_list.*****
shape: (1, 16)
┌─────┬─────────────┬───────┬─────────────┬───┬─────────────┬────────────┬────────────┬────────────┐
│ id ┆ type ┆ name ┆ symmetry_op ┆ … ┆ matrix[3][1 ┆ matrix[3][ ┆ matrix[3][ ┆ vector[3] │
│ --- ┆ --- ┆ --- ┆ eration ┆ ┆ ] ┆ 2] ┆ 3] ┆ --- │
│ str ┆ str ┆ str ┆ --- ┆ ┆ --- ┆ --- ┆ --- ┆ str │
│ ┆ ┆ ┆ str ┆ ┆ str ┆ str ┆ str ┆ │
╞═════╪═════════════╪═══════╪═════════════╪═══╪═════════════╪════════════╪════════════╪════════════╡
│ 1 ┆ identity ┆ 1_555 ┆ x,y,z ┆ … ┆ 0.000000000 ┆ 0.00000000 ┆ 1.00000000 ┆ 0.00000000 │
│ ┆ operation ┆ ┆ ┆ ┆ 0 ┆ 00 ┆ 00 ┆ 00 │
└─────┴─────────────┴───────┴─────────────┴───┴─────────────┴────────────┴────────────┴────────────┘
*****_struct_biol.*****
shape: (1, 3)
┌─────┬─────────────────────────────────┬─────────────────────┐
│ id ┆ details ┆ pdbx_parent_biol_id │
│ --- ┆ --- ┆ --- │
│ str ┆ str ┆ null │
╞═════╪═════════════════════════════════╪═════════════════════╡
│ 1 ┆ THERE IS A DIMER (IDENTICAL CH… ┆ null │
└─────┴─────────────────────────────────┴─────────────────────┘
*****_struct_conf.*****
shape: (32, 20)
┌────────────┬──────────┬────────────┬───────────┬───┬───────────┬───────────┬─────────┬───────────┐
│ conf_type_ ┆ id ┆ pdbx_PDB_h ┆ beg_label ┆ … ┆ end_auth_ ┆ pdbx_PDB_ ┆ details ┆ pdbx_PDB_ │
│ id ┆ --- ┆ elix_id ┆ _comp_id ┆ ┆ seq_id ┆ helix_cla ┆ --- ┆ helix_len │
│ --- ┆ str ┆ --- ┆ --- ┆ ┆ --- ┆ ss ┆ null ┆ gth │
│ str ┆ ┆ str ┆ str ┆ ┆ str ┆ --- ┆ ┆ --- │
│ ┆ ┆ ┆ ┆ ┆ ┆ str ┆ ┆ str │
╞════════════╪══════════╪════════════╪═══════════╪═══╪═══════════╪═══════════╪═════════╪═══════════╡
│ HELX_P ┆ HELX_P1 ┆ H1 ┆ GLN ┆ … ┆ 37 ┆ 1 ┆ null ┆ 9 │
│ HELX_P ┆ HELX_P2 ┆ H2 ┆ GLY ┆ … ┆ 66 ┆ 1 ┆ null ┆ 13 │
│ HELX_P ┆ HELX_P3 ┆ H3 ┆ PHE ┆ … ┆ 78 ┆ 1 ┆ null ┆ 7 │
│ HELX_P ┆ HELX_P4 ┆ H4 ┆ ASP ┆ … ┆ 112 ┆ 1 ┆ null ┆ 12 │
│ HELX_P ┆ HELX_P5 ┆ H5 ┆ THR ┆ … ┆ 140 ┆ 1 ┆ null ┆ 10 │
│ … ┆ … ┆ … ┆ … ┆ … ┆ … ┆ … ┆ … ┆ … │
│ HELX_P ┆ HELX_P28 ┆ G12 ┆ HIS ┆ … ┆ 281 ┆ 1 ┆ null ┆ 6 │
│ HELX_P ┆ HELX_P29 ┆ G13 ┆ THR ┆ … ┆ 313 ┆ 1 ┆ null ┆ 16 │
│ HELX_P ┆ HELX_P30 ┆ G14 ┆ SER ┆ … ┆ 333 ┆ 1 ┆ null ┆ 9 │
│ HELX_P ┆ HELX_P31 ┆ G15 ┆ ASN ┆ … ┆ 360 ┆ 1 ┆ null ┆ 27 │
│ HELX_P ┆ HELX_P32 ┆ G16 ┆ ASP ┆ … ┆ 447 ┆ 1 ┆ null ┆ 14 │
└────────────┴──────────┴────────────┴───────────┴───┴───────────┴───────────┴─────────┴───────────┘
*****_struct_conf_type.*****
shape: (1, 3)
┌────────┬──────────┬───────────┐
│ id ┆ criteria ┆ reference │
│ --- ┆ --- ┆ --- │
│ str ┆ null ┆ null │
╞════════╪══════════╪═══════════╡
│ HELX_P ┆ null ┆ null │
└────────┴──────────┴───────────┘
*****_struct_conn.*****
shape: (34, 35)
┌──────────┬────────────┬────────────┬───────────┬───┬─────────┬───────────┬───────────┬───────────┐
│ id ┆ conn_type_ ┆ pdbx_leavi ┆ pdbx_PDB_ ┆ … ┆ details ┆ pdbx_dist ┆ pdbx_valu ┆ pdbx_role │
│ --- ┆ id ┆ ng_atom_fl ┆ id ┆ ┆ --- ┆ _value ┆ e_order ┆ --- │
│ str ┆ --- ┆ ag ┆ --- ┆ ┆ null ┆ --- ┆ --- ┆ null │
│ ┆ str ┆ --- ┆ null ┆ ┆ ┆ str ┆ null ┆ │
│ ┆ ┆ null ┆ ┆ ┆ ┆ ┆ ┆ │
╞══════════╪════════════╪════════════╪═══════════╪═══╪═════════╪═══════════╪═══════════╪═══════════╡
│ disulf1 ┆ disulf ┆ null ┆ null ┆ … ┆ null ┆ 1.975 ┆ null ┆ null │
│ disulf2 ┆ disulf ┆ null ┆ null ┆ … ┆ null ┆ 1.995 ┆ null ┆ null │
│ disulf3 ┆ disulf ┆ null ┆ null ┆ … ┆ null ┆ 1.996 ┆ null ┆ null │
│ disulf4 ┆ disulf ┆ null ┆ null ┆ … ┆ null ┆ 2.032 ┆ null ┆ null │
│ metalc1 ┆ metalc ┆ null ┆ null ┆ … ┆ null ┆ 1.996 ┆ null ┆ null │
│ … ┆ … ┆ … ┆ … ┆ … ┆ … ┆ … ┆ … ┆ … │
│ metalc26 ┆ metalc ┆ null ┆ null ┆ … ┆ null ┆ 2.083 ┆ null ┆ null │
│ metalc27 ┆ metalc ┆ null ┆ null ┆ … ┆ null ┆ 2.059 ┆ null ┆ null │
│ metalc28 ┆ metalc ┆ null ┆ null ┆ … ┆ null ┆ 1.926 ┆ null ┆ null │
│ metalc29 ┆ metalc ┆ null ┆ null ┆ … ┆ null ┆ 2.007 ┆ null ┆ null │
│ metalc30 ┆ metalc ┆ null ┆ null ┆ … ┆ null ┆ 2.027 ┆ null ┆ null │
└──────────┴────────────┴────────────┴───────────┴───┴─────────┴───────────┴───────────┴───────────┘
*****_struct_conn_type.*****
shape: (2, 3)
┌────────┬──────────┬───────────┐
│ id ┆ criteria ┆ reference │
│ --- ┆ --- ┆ --- │
│ str ┆ null ┆ null │
╞════════╪══════════╪═══════════╡
│ disulf ┆ null ┆ null │
│ metalc ┆ null ┆ null │
└────────┴──────────┴───────────┘
*****_pdbx_struct_conn_angle.*****
shape: (61, 36)
┌─────┬─────────────┬─────────────┬─────────────┬───┬─────────────┬────────────┬───────┬───────────┐
│ id ┆ ptnr1_label ┆ ptnr1_label ┆ ptnr1_label ┆ … ┆ ptnr3_PDB_i ┆ ptnr3_symm ┆ value ┆ value_esd │
│ --- ┆ _atom_id ┆ _alt_id ┆ _asym_id ┆ ┆ ns_code ┆ etry ┆ --- ┆ --- │
│ str ┆ --- ┆ --- ┆ --- ┆ ┆ --- ┆ --- ┆ str ┆ null │
│ ┆ str ┆ null ┆ str ┆ ┆ null ┆ str ┆ ┆ │
╞═════╪═════════════╪═════════════╪═════════════╪═══╪═════════════╪════════════╪═══════╪═══════════╡
│ 1 ┆ OD1 ┆ null ┆ A ┆ … ┆ null ┆ 1_555 ┆ 117.7 ┆ null │
│ 2 ┆ OD1 ┆ null ┆ A ┆ … ┆ null ┆ 1_555 ┆ 106.5 ┆ null │
│ 3 ┆ OG ┆ null ┆ A ┆ … ┆ null ┆ 1_555 ┆ 73.3 ┆ null │
│ 4 ┆ OD1 ┆ null ┆ A ┆ … ┆ null ┆ 1_555 ┆ 119.4 ┆ null │
│ 5 ┆ OG ┆ null ┆ A ┆ … ┆ null ┆ 1_555 ┆ 122.8 ┆ null │
│ … ┆ … ┆ … ┆ … ┆ … ┆ … ┆ … ┆ … ┆ … │
│ 57 ┆ OD1 ┆ null ┆ B ┆ … ┆ null ┆ 1_555 ┆ 96.9 ┆ null │
│ 58 ┆ NE2 ┆ null ┆ B ┆ … ┆ null ┆ 1_555 ┆ 93.7 ┆ null │
│ 59 ┆ OD1 ┆ null ┆ B ┆ … ┆ null ┆ 1_555 ┆ 137.8 ┆ null │
│ 60 ┆ NE2 ┆ null ┆ B ┆ … ┆ null ┆ 1_555 ┆ 107.0 ┆ null │
│ 61 ┆ NE2 ┆ null ┆ B ┆ … ┆ null ┆ 1_555 ┆ 109.3 ┆ null │
└─────┴─────────────┴─────────────┴─────────────┴───┴─────────────┴────────────┴───────┴───────────┘
*****_struct_sheet.*****
shape: (4, 4)
┌─────┬──────┬────────────────┬─────────┐
│ id ┆ type ┆ number_strands ┆ details │
│ --- ┆ --- ┆ --- ┆ --- │
│ str ┆ null ┆ str ┆ null │
╞═════╪══════╪════════════════╪═════════╡
│ S1A ┆ null ┆ 10 ┆ null │
│ S2A ┆ null ┆ 3 ┆ null │
│ S1B ┆ null ┆ 10 ┆ null │
│ S2B ┆ null ┆ 3 ┆ null │
└─────┴──────┴────────────────┴─────────┘
*****_struct_sheet_order.*****
shape: (22, 5)
┌──────────┬────────────┬────────────┬────────┬───────────────┐
│ sheet_id ┆ range_id_1 ┆ range_id_2 ┆ offset ┆ sense │
│ --- ┆ --- ┆ --- ┆ --- ┆ --- │
│ str ┆ str ┆ str ┆ null ┆ str │
╞══════════╪════════════╪════════════╪════════╪═══════════════╡
│ S1A ┆ 1 ┆ 2 ┆ null ┆ parallel │
│ S1A ┆ 2 ┆ 3 ┆ null ┆ parallel │
│ S1A ┆ 3 ┆ 4 ┆ null ┆ parallel │
│ S1A ┆ 4 ┆ 5 ┆ null ┆ parallel │
│ S1A ┆ 5 ┆ 6 ┆ null ┆ parallel │
│ … ┆ … ┆ … ┆ … ┆ … │
│ S1B ┆ 7 ┆ 8 ┆ null ┆ parallel │
│ S1B ┆ 8 ┆ 9 ┆ null ┆ anti-parallel │
│ S1B ┆ 9 ┆ 10 ┆ null ┆ parallel │
│ S2B ┆ 1 ┆ 2 ┆ null ┆ parallel │
│ S2B ┆ 2 ┆ 3 ┆ null ┆ anti-parallel │
└──────────┴────────────┴────────────┴────────┴───────────────┘
*****_struct_sheet_range.*****
shape: (26, 16)
┌──────────┬─────┬────────────┬────────────┬───┬────────────┬────────────┬────────────┬────────────┐
│ sheet_id ┆ id ┆ beg_label_ ┆ beg_label_ ┆ … ┆ beg_auth_s ┆ end_auth_c ┆ end_auth_a ┆ end_auth_s │
│ --- ┆ --- ┆ comp_id ┆ asym_id ┆ ┆ eq_id ┆ omp_id ┆ sym_id ┆ eq_id │
│ str ┆ str ┆ --- ┆ --- ┆ ┆ --- ┆ --- ┆ --- ┆ --- │
│ ┆ ┆ str ┆ str ┆ ┆ str ┆ str ┆ str ┆ str │
╞══════════╪═════╪════════════╪════════════╪═══╪════════════╪════════════╪════════════╪════════════╡
│ S1A ┆ 1 ┆ ASN ┆ A ┆ … ┆ 44 ┆ GLY ┆ A ┆ 50 │
│ S1A ┆ 2 ┆ LEU ┆ A ┆ … ┆ 80 ┆ THR ┆ A ┆ 85 │
│ S1A ┆ 3 ┆ ALA ┆ A ┆ … ┆ 142 ┆ THR ┆ A ┆ 148 │
│ S1A ┆ 4 ┆ ASP ┆ A ┆ … ┆ 201 ┆ GLY ┆ A ┆ 205 │
│ S1A ┆ 5 ┆ GLN ┆ A ┆ … ┆ 235 ┆ VAL ┆ A ┆ 237 │
│ … ┆ … ┆ … ┆ … ┆ … ┆ … ┆ … ┆ … ┆ … │
│ S1B ┆ 9 ┆ LEU ┆ B ┆ … ┆ 417 ┆ TYR ┆ B ┆ 422 │
│ S1B ┆ 10 ┆ GLY ┆ B ┆ … ┆ 431 ┆ ASP ┆ B ┆ 434 │
│ S2B ┆ 1 ┆ GLN ┆ B ┆ … ┆ 375 ┆ VAL ┆ B ┆ 377 │
│ S2B ┆ 2 ┆ LEU ┆ B ┆ … ┆ 386 ┆ ASN ┆ B ┆ 391 │
│ S2B ┆ 3 ┆ VAL ┆ B ┆ … ┆ 397 ┆ TYR ┆ B ┆ 402 │
└──────────┴─────┴────────────┴────────────┴───┴────────────┴────────────┴────────────┴────────────┘
*****_struct_site.*****
shape: (8, 8)
┌─────┬─────────────┬─────────────┬────────────┬────────────┬────────────┬────────────┬────────────┐
│ id ┆ pdbx_eviden ┆ pdbx_auth_a ┆ pdbx_auth_ ┆ pdbx_auth_ ┆ pdbx_auth_ ┆ pdbx_num_r ┆ details │
│ --- ┆ ce_code ┆ sym_id ┆ comp_id ┆ seq_id ┆ ins_code ┆ esidues ┆ --- │
│ str ┆ --- ┆ --- ┆ --- ┆ --- ┆ --- ┆ --- ┆ str │
│ ┆ str ┆ str ┆ str ┆ str ┆ null ┆ str ┆ │
╞═════╪═════════════╪═════════════╪════════════╪════════════╪════════════╪════════════╪════════════╡
│ AC1 ┆ Software ┆ A ┆ ZN ┆ 450 ┆ null ┆ 4 ┆ BINDING │
│ ┆ ┆ ┆ ┆ ┆ ┆ ┆ SITE FOR │
│ ┆ ┆ ┆ ┆ ┆ ┆ ┆ RESIDUE ZN │
│ ┆ ┆ ┆ ┆ ┆ ┆ ┆ A … │
│ AC2 ┆ Software ┆ A ┆ ZN ┆ 451 ┆ null ┆ 6 ┆ BINDING │
│ ┆ ┆ ┆ ┆ ┆ ┆ ┆ SITE FOR │
│ ┆ ┆ ┆ ┆ ┆ ┆ ┆ RESIDUE ZN │
│ ┆ ┆ ┆ ┆ ┆ ┆ ┆ A … │
│ AC3 ┆ Software ┆ A ┆ MG ┆ 452 ┆ null ┆ 5 ┆ BINDING │
│ ┆ ┆ ┆ ┆ ┆ ┆ ┆ SITE FOR │
│ ┆ ┆ ┆ ┆ ┆ ┆ ┆ RESIDUE MG │
│ ┆ ┆ ┆ ┆ ┆ ┆ ┆ A … │
│ AC4 ┆ Software ┆ A ┆ PO4 ┆ 453 ┆ null ┆ 11 ┆ BINDING │
│ ┆ ┆ ┆ ┆ ┆ ┆ ┆ SITE FOR │
│ ┆ ┆ ┆ ┆ ┆ ┆ ┆ RESIDUE │
│ ┆ ┆ ┆ ┆ ┆ ┆ ┆ PO4 A… │
│ AC5 ┆ Software ┆ B ┆ ZN ┆ 450 ┆ null ┆ 4 ┆ BINDING │
│ ┆ ┆ ┆ ┆ ┆ ┆ ┆ SITE FOR │
│ ┆ ┆ ┆ ┆ ┆ ┆ ┆ RESIDUE ZN │
│ ┆ ┆ ┆ ┆ ┆ ┆ ┆ B … │
│ AC6 ┆ Software ┆ B ┆ ZN ┆ 451 ┆ null ┆ 6 ┆ BINDING │
│ ┆ ┆ ┆ ┆ ┆ ┆ ┆ SITE FOR │
│ ┆ ┆ ┆ ┆ ┆ ┆ ┆ RESIDUE ZN │
│ ┆ ┆ ┆ ┆ ┆ ┆ ┆ B … │
│ AC7 ┆ Software ┆ B ┆ MG ┆ 452 ┆ null ┆ 6 ┆ BINDING │
│ ┆ ┆ ┆ ┆ ┆ ┆ ┆ SITE FOR │
│ ┆ ┆ ┆ ┆ ┆ ┆ ┆ RESIDUE MG │
│ ┆ ┆ ┆ ┆ ┆ ┆ ┆ B … │
│ AC8 ┆ Software ┆ B ┆ PO4 ┆ 453 ┆ null ┆ 12 ┆ BINDING │
│ ┆ ┆ ┆ ┆ ┆ ┆ ┆ SITE FOR │
│ ┆ ┆ ┆ ┆ ┆ ┆ ┆ RESIDUE │
│ ┆ ┆ ┆ ┆ ┆ ┆ ┆ PO4 B… │
└─────┴─────────────┴─────────────┴────────────┴────────────┴────────────┴────────────┴────────────┘
*****_struct_site_gen.*****
shape: (54, 14)
┌─────┬─────────┬──────────────┬──────────────┬───┬─────────────┬─────────────┬──────────┬─────────┐
│ id ┆ site_id ┆ pdbx_num_res ┆ label_comp_i ┆ … ┆ label_atom_ ┆ label_alt_i ┆ symmetry ┆ details │
│ --- ┆ --- ┆ --- ┆ d ┆ ┆ id ┆ d ┆ --- ┆ --- │
│ str ┆ str ┆ str ┆ --- ┆ ┆ --- ┆ --- ┆ str ┆ null │
│ ┆ ┆ ┆ str ┆ ┆ bool ┆ null ┆ ┆ │
╞═════╪═════════╪══════════════╪══════════════╪═══╪═════════════╪═════════════╪══════════╪═════════╡
│ 1 ┆ AC1 ┆ 4 ┆ ASP ┆ … ┆ false ┆ null ┆ 1_555 ┆ null │
│ 2 ┆ AC1 ┆ 4 ┆ HIS ┆ … ┆ false ┆ null ┆ 1_555 ┆ null │
│ 3 ┆ AC1 ┆ 4 ┆ HIS ┆ … ┆ false ┆ null ┆ 1_555 ┆ null │
│ 4 ┆ AC1 ┆ 4 ┆ PO4 ┆ … ┆ false ┆ null ┆ 1_555 ┆ null │
│ 5 ┆ AC2 ┆ 6 ┆ ASP ┆ … ┆ false ┆ null ┆ 1_555 ┆ null │
│ … ┆ … ┆ … ┆ … ┆ … ┆ … ┆ … ┆ … ┆ … │
│ 50 ┆ AC8 ┆ 12 ┆ HIS ┆ … ┆ false ┆ null ┆ 1_555 ┆ null │
│ 51 ┆ AC8 ┆ 12 ┆ HIS ┆ … ┆ false ┆ null ┆ 1_555 ┆ null │
│ 52 ┆ AC8 ┆ 12 ┆ ZN ┆ … ┆ false ┆ null ┆ 1_555 ┆ null │
│ 53 ┆ AC8 ┆ 12 ┆ ZN ┆ … ┆ false ┆ null ┆ 1_555 ┆ null │
│ 54 ┆ AC8 ┆ 12 ┆ HOH ┆ … ┆ false ┆ null ┆ 1_555 ┆ null │
└─────┴─────────┴──────────────┴──────────────┴───┴─────────────┴─────────────┴──────────┴─────────┘
*****_pdbx_validate_close_contact.*****
shape: (2, 15)
┌─────┬─────────────┬─────────────┬─────────────┬───┬─────────────┬────────────┬────────────┬──────┐
│ id ┆ PDB_model_n ┆ auth_atom_i ┆ auth_asym_i ┆ … ┆ auth_seq_id ┆ PDB_ins_co ┆ label_alt_ ┆ dist │
│ --- ┆ um ┆ d_1 ┆ d_1 ┆ ┆ _2 ┆ de_2 ┆ id_2 ┆ --- │
│ str ┆ --- ┆ --- ┆ --- ┆ ┆ --- ┆ --- ┆ --- ┆ str │
│ ┆ str ┆ str ┆ str ┆ ┆ str ┆ null ┆ null ┆ │
╞═════╪═════════════╪═════════════╪═════════════╪═══╪═════════════╪════════════╪════════════╪══════╡
│ 1 ┆ 1 ┆ OG ┆ A ┆ … ┆ 453 ┆ null ┆ null ┆ 1.92 │
│ 2 ┆ 1 ┆ OG ┆ B ┆ … ┆ 453 ┆ null ┆ null ┆ 1.97 │
└─────┴─────────────┴─────────────┴─────────────┴───┴─────────────┴────────────┴────────────┴──────┘
*****_pdbx_validate_rmsd_bond.*****
shape: (2, 19)
┌─────┬────────────┬────────────┬────────────┬───┬────────────┬────────────┬───────────┬───────────┐
│ id ┆ PDB_model_ ┆ auth_atom_ ┆ auth_asym_ ┆ … ┆ bond_targe ┆ bond_devia ┆ bond_stan ┆ linker_fl │
│ --- ┆ num ┆ id_1 ┆ id_1 ┆ ┆ t_value ┆ tion ┆ dard_devi ┆ ag │
│ str ┆ --- ┆ --- ┆ --- ┆ ┆ --- ┆ --- ┆ ation ┆ --- │
│ ┆ str ┆ str ┆ str ┆ ┆ str ┆ str ┆ --- ┆ str │
│ ┆ ┆ ┆ ┆ ┆ ┆ ┆ str ┆ │
╞═════╪════════════╪════════════╪════════════╪═══╪════════════╪════════════╪═══════════╪═══════════╡
│ 1 ┆ 1 ┆ CD ┆ A ┆ … ┆ 1.252 ┆ -0.074 ┆ 0.011 ┆ N │
│ 2 ┆ 1 ┆ CD ┆ A ┆ … ┆ 1.235 ┆ 0.149 ┆ 0.022 ┆ N │
└─────┴────────────┴────────────┴────────────┴───┴────────────┴────────────┴───────────┴───────────┘
*****_pdbx_validate_rmsd_angle.*****
shape: (29, 25)
┌─────┬────────────┬────────────┬────────────┬───┬────────────┬────────────┬───────────┬───────────┐
│ id ┆ PDB_model_ ┆ auth_atom_ ┆ auth_asym_ ┆ … ┆ angle_targ ┆ angle_devi ┆ angle_sta ┆ linker_fl │
│ --- ┆ num ┆ id_1 ┆ id_1 ┆ ┆ et_value ┆ ation ┆ ndard_dev ┆ ag │
│ str ┆ --- ┆ --- ┆ --- ┆ ┆ --- ┆ --- ┆ iation ┆ --- │
│ ┆ str ┆ str ┆ str ┆ ┆ str ┆ str ┆ --- ┆ str │
│ ┆ ┆ ┆ ┆ ┆ ┆ ┆ str ┆ │
╞═════╪════════════╪════════════╪════════════╪═══╪════════════╪════════════╪═══════════╪═══════════╡
│ 1 ┆ 1 ┆ NE ┆ A ┆ … ┆ 120.30 ┆ -4.27 ┆ 0.50 ┆ N │
│ 2 ┆ 1 ┆ NE ┆ A ┆ … ┆ 120.30 ┆ 3.22 ┆ 0.50 ┆ N │
│ 3 ┆ 1 ┆ CA ┆ A ┆ … ┆ 115.30 ┆ 28.82 ┆ 2.30 ┆ N │
│ 4 ┆ 1 ┆ CA ┆ A ┆ … ┆ 113.40 ┆ 13.53 ┆ 1.90 ┆ N │
│ 5 ┆ 1 ┆ CB ┆ A ┆ … ┆ 121.00 ┆ 5.55 ┆ 0.60 ┆ N │
│ … ┆ … ┆ … ┆ … ┆ … ┆ … ┆ … ┆ … ┆ … │
│ 25 ┆ 1 ┆ CB ┆ B ┆ … ┆ 118.30 ┆ 6.71 ┆ 0.90 ┆ N │
│ 26 ┆ 1 ┆ CD ┆ B ┆ … ┆ 123.60 ┆ 31.22 ┆ 1.40 ┆ N │
│ 27 ┆ 1 ┆ NH1 ┆ B ┆ … ┆ 119.40 ┆ -11.67 ┆ 1.10 ┆ N │
│ 28 ┆ 1 ┆ NE ┆ B ┆ … ┆ 120.30 ┆ 4.10 ┆ 0.50 ┆ N │
│ 29 ┆ 1 ┆ NE ┆ B ┆ … ┆ 120.30 ┆ 7.41 ┆ 0.50 ┆ N │
└─────┴────────────┴────────────┴────────────┴───┴────────────┴────────────┴───────────┴───────────┘
*****_pdbx_validate_torsion.*****
shape: (19, 9)
┌─────┬─────────────┬─────────────┬─────────────┬───┬─────────────┬────────────┬─────────┬─────────┐
│ id ┆ PDB_model_n ┆ auth_comp_i ┆ auth_asym_i ┆ … ┆ PDB_ins_cod ┆ label_alt_ ┆ phi ┆ psi │
│ --- ┆ um ┆ d ┆ d ┆ ┆ e ┆ id ┆ --- ┆ --- │
│ str ┆ --- ┆ --- ┆ --- ┆ ┆ --- ┆ --- ┆ str ┆ str │
│ ┆ str ┆ str ┆ str ┆ ┆ null ┆ null ┆ ┆ │
╞═════╪═════════════╪═════════════╪═════════════╪═══╪═════════════╪════════════╪═════════╪═════════╡
│ 1 ┆ 1 ┆ PRO ┆ A ┆ … ┆ null ┆ null ┆ -52.81 ┆ -177.76 │
│ 2 ┆ 1 ┆ GLU ┆ A ┆ … ┆ null ┆ null ┆ 145.31 ┆ 140.53 │
│ 3 ┆ 1 ┆ ALA ┆ A ┆ … ┆ null ┆ null ┆ -110.12 ┆ -168.33 │
│ 4 ┆ 1 ┆ SER ┆ A ┆ … ┆ null ┆ null ┆ -143.49 ┆ 23.75 │
│ 5 ┆ 1 ┆ ASN ┆ A ┆ … ┆ null ┆ null ┆ 90.00 ┆ 158.97 │
│ … ┆ … ┆ … ┆ … ┆ … ┆ … ┆ … ┆ … ┆ … │
│ 15 ┆ 1 ┆ GLN ┆ B ┆ … ┆ null ┆ null ┆ -114.48 ┆ 57.35 │
│ 16 ┆ 1 ┆ LYS ┆ B ┆ … ┆ null ┆ null ┆ 159.84 ┆ 42.87 │
│ 17 ┆ 1 ┆ ASN ┆ B ┆ … ┆ null ┆ null ┆ 56.70 ┆ 163.76 │
│ 18 ┆ 1 ┆ THR ┆ B ┆ … ┆ null ┆ null ┆ -160.71 ┆ -166.30 │
│ 19 ┆ 1 ┆ GLU ┆ B ┆ … ┆ null ┆ null ┆ -78.73 ┆ -154.20 │
└─────┴─────────────┴─────────────┴─────────────┴───┴─────────────┴────────────┴─────────┴─────────┘
*****_pdbx_validate_planes.*****
shape: (1, 9)
┌─────┬─────────────┬─────────────┬─────────────┬───┬────────────┬────────────┬───────┬────────────┐
│ id ┆ PDB_model_n ┆ auth_comp_i ┆ auth_asym_i ┆ … ┆ PDB_ins_co ┆ label_alt_ ┆ rmsd ┆ type │
│ --- ┆ um ┆ d ┆ d ┆ ┆ de ┆ id ┆ --- ┆ --- │
│ str ┆ --- ┆ --- ┆ --- ┆ ┆ --- ┆ --- ┆ str ┆ str │
│ ┆ str ┆ str ┆ str ┆ ┆ null ┆ null ┆ ┆ │
╞═════╪═════════════╪═════════════╪═════════════╪═══╪════════════╪════════════╪═══════╪════════════╡
│ 1 ┆ 1 ┆ ARG ┆ A ┆ … ┆ null ┆ null ┆ 0.081 ┆ SIDE CHAIN │
└─────┴─────────────┴─────────────┴─────────────┴───┴────────────┴────────────┴───────┴────────────┘
*****_pdbx_entry_details.*****
shape: (1, 6)
┌──────────┬─────────────────┬────────────────┬─────────────────┬─────────────────┬────────────────┐
│ entry_id ┆ compound_detail ┆ source_details ┆ nonpolymer_deta ┆ sequence_detail ┆ has_ligand_of_ │
│ --- ┆ s ┆ --- ┆ ils ┆ s ┆ interest │
│ str ┆ --- ┆ null ┆ --- ┆ --- ┆ --- │
│ ┆ null ┆ ┆ str ┆ str ┆ null │
╞══════════╪═════════════════╪════════════════╪═════════════════╪═════════════════╪════════════════╡
│ 1ALK ┆ null ┆ null ┆ THERE ARE THREE ┆ SEQUENCE ┆ null │
│ ┆ ┆ ┆ WATER MOLECULE… ┆ ADVISORY ┆ │
│ ┆ ┆ ┆ ┆ NOTICE: ┆ │
│ ┆ ┆ ┆ ┆ … ┆ │
└──────────┴─────────────────┴────────────────┴─────────────────┴─────────────────┴────────────────┘
*****_chem_comp_atom.*****
shape: (397, 6)
┌─────────┬─────────┬─────────────┬────────────────────┬────────────────────┬──────────────┐
│ comp_id ┆ atom_id ┆ type_symbol ┆ pdbx_aromatic_flag ┆ pdbx_stereo_config ┆ pdbx_ordinal │
│ --- ┆ --- ┆ --- ┆ --- ┆ --- ┆ --- │
│ str ┆ str ┆ str ┆ str ┆ str ┆ str │
╞═════════╪═════════╪═════════════╪════════════════════╪════════════════════╪══════════════╡
│ ALA ┆ N ┆ N ┆ N ┆ N ┆ 1 │
│ ALA ┆ CA ┆ C ┆ N ┆ S ┆ 2 │
│ ALA ┆ C ┆ C ┆ N ┆ N ┆ 3 │
│ ALA ┆ O ┆ O ┆ N ┆ N ┆ 4 │
│ ALA ┆ CB ┆ C ┆ N ┆ N ┆ 5 │
│ … ┆ … ┆ … ┆ … ┆ … ┆ … │
│ VAL ┆ HG21 ┆ H ┆ N ┆ N ┆ 393 │
│ VAL ┆ HG22 ┆ H ┆ N ┆ N ┆ 394 │
│ VAL ┆ HG23 ┆ H ┆ N ┆ N ┆ 395 │
│ VAL ┆ HXT ┆ H ┆ N ┆ N ┆ 396 │
│ ZN ┆ ZN ┆ ZN ┆ N ┆ N ┆ 397 │
└─────────┴─────────┴─────────────┴────────────────────┴────────────────────┴──────────────┘
*****_chem_comp_bond.*****
shape: (379, 7)
┌─────────┬───────────┬───────────┬─────────────┬─────────────────┬─────────────────┬──────────────┐
│ comp_id ┆ atom_id_1 ┆ atom_id_2 ┆ value_order ┆ pdbx_aromatic_f ┆ pdbx_stereo_con ┆ pdbx_ordinal │
│ --- ┆ --- ┆ --- ┆ --- ┆ lag ┆ fig ┆ --- │
│ str ┆ str ┆ str ┆ str ┆ --- ┆ --- ┆ str │
│ ┆ ┆ ┆ ┆ str ┆ str ┆ │
╞═════════╪═══════════╪═══════════╪═════════════╪═════════════════╪═════════════════╪══════════════╡
│ ALA ┆ N ┆ CA ┆ sing ┆ N ┆ N ┆ 1 │
│ ALA ┆ N ┆ H ┆ sing ┆ N ┆ N ┆ 2 │
│ ALA ┆ N ┆ H2 ┆ sing ┆ N ┆ N ┆ 3 │
│ ALA ┆ CA ┆ C ┆ sing ┆ N ┆ N ┆ 4 │
│ ALA ┆ CA ┆ CB ┆ sing ┆ N ┆ N ┆ 5 │
│ … ┆ … ┆ … ┆ … ┆ … ┆ … ┆ … │
│ VAL ┆ CG1 ┆ HG13 ┆ sing ┆ N ┆ N ┆ 375 │
│ VAL ┆ CG2 ┆ HG21 ┆ sing ┆ N ┆ N ┆ 376 │
│ VAL ┆ CG2 ┆ HG22 ┆ sing ┆ N ┆ N ┆ 377 │
│ VAL ┆ CG2 ┆ HG23 ┆ sing ┆ N ┆ N ┆ 378 │
│ VAL ┆ OXT ┆ HXT ┆ sing ┆ N ┆ N ┆ 379 │
└─────────┴───────────┴───────────┴─────────────┴─────────────────┴─────────────────┴──────────────┘
*****_atom_sites.*****
shape: (1, 13)
┌──────────┬───────────┬───────────┬───────────┬───┬───────────┬───────────┬───────────┬───────────┐
│ entry_id ┆ fract_tra ┆ fract_tra ┆ fract_tra ┆ … ┆ fract_tra ┆ fract_tra ┆ fract_tra ┆ fract_tra │
│ --- ┆ nsf_matri ┆ nsf_matri ┆ nsf_matri ┆ ┆ nsf_matri ┆ nsf_vecto ┆ nsf_vecto ┆ nsf_vecto │
│ str ┆ x[1][1] ┆ x[1][2] ┆ x[1][3] ┆ ┆ x[3][3] ┆ r[1] ┆ r[2] ┆ r[3] │
│ ┆ --- ┆ --- ┆ --- ┆ ┆ --- ┆ --- ┆ --- ┆ --- │
│ ┆ str ┆ str ┆ str ┆ ┆ str ┆ str ┆ str ┆ str │
╞══════════╪═══════════╪═══════════╪═══════════╪═══╪═══════════╪═══════════╪═══════════╪═══════════╡
│ 1ALK ┆ 0.005120 ┆ 0.000000 ┆ 0.000000 ┆ … ┆ 0.013038 ┆ 0.00000 ┆ 0.00000 ┆ 0.00000 │
└──────────┴───────────┴───────────┴───────────┴───┴───────────┴───────────┴───────────┴───────────┘
*****_atom_sites_footnote.*****
shape: (1, 2)
┌─────┬─────────────────────────────────┐
│ id ┆ text │
│ --- ┆ --- │
│ str ┆ str │
╞═════╪═════════════════════════════════╡
│ 1 ┆ RESIDUES 1-4 AND 404-409 ARE N… │
└─────┴─────────────────────────────────┘
*****_atom_type.*****
shape: (7, 1)
┌────────┐
│ symbol │
│ --- │
│ str │
╞════════╡
│ C │
│ MG │
│ N │
│ O │
│ P │
│ S │
│ ZN │
└────────┘
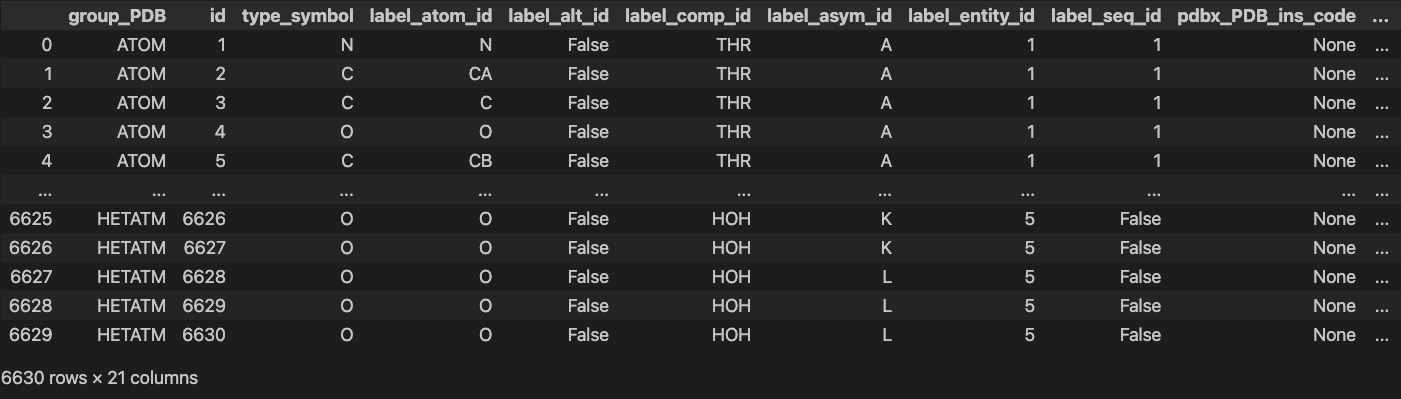
*****_atom_site.*****
shape: (6_630, 21)
┌───────────┬──────┬────────────┬────────────┬───┬────────────┬────────────┬───────────┬───────────┐
│ group_PDB ┆ id ┆ type_symbo ┆ label_atom ┆ … ┆ auth_comp_ ┆ auth_asym_ ┆ auth_atom ┆ pdbx_PDB_ │
│ --- ┆ --- ┆ l ┆ _id ┆ ┆ id ┆ id ┆ _id ┆ model_num │
│ str ┆ str ┆ --- ┆ --- ┆ ┆ --- ┆ --- ┆ --- ┆ --- │
│ ┆ ┆ str ┆ str ┆ ┆ str ┆ str ┆ str ┆ str │
╞═══════════╪══════╪════════════╪════════════╪═══╪════════════╪════════════╪═══════════╪═══════════╡
│ ATOM ┆ 1 ┆ N ┆ N ┆ … ┆ THR ┆ A ┆ N ┆ 1 │
│ ATOM ┆ 2 ┆ C ┆ CA ┆ … ┆ THR ┆ A ┆ CA ┆ 1 │
│ ATOM ┆ 3 ┆ C ┆ C ┆ … ┆ THR ┆ A ┆ C ┆ 1 │
│ ATOM ┆ 4 ┆ O ┆ O ┆ … ┆ THR ┆ A ┆ O ┆ 1 │
│ ATOM ┆ 5 ┆ C ┆ CB ┆ … ┆ THR ┆ A ┆ CB ┆ 1 │
│ … ┆ … ┆ … ┆ … ┆ … ┆ … ┆ … ┆ … ┆ … │
│ HETATM ┆ 6626 ┆ O ┆ O ┆ … ┆ HOH ┆ A ┆ O ┆ 1 │
│ HETATM ┆ 6627 ┆ O ┆ O ┆ … ┆ HOH ┆ A ┆ O ┆ 1 │
│ HETATM ┆ 6628 ┆ O ┆ O ┆ … ┆ HOH ┆ B ┆ O ┆ 1 │
│ HETATM ┆ 6629 ┆ O ┆ O ┆ … ┆ HOH ┆ B ┆ O ┆ 1 │
│ HETATM ┆ 6630 ┆ O ┆ O ┆ … ┆ HOH ┆ B ┆ O ┆ 1 │
└───────────┴──────┴────────────┴────────────┴───┴────────────┴────────────┴───────────┴───────────┘
もしどのカテゴリにどんなことが書かれているか知りたかったらPDBx/mmCIF Dictionary Resourcesで調べればOK!!
Pandas/PolarsでCIFファイルのデータ解析
dataframeとして扱えるようになったので座標情報が書かれている_atom_siteを中心にデータ解析もしてみましょう!
atom_site
データ解析する前に、atom_siteのitemに何が書かれているか簡単に解説していきましょう。
詳しく知りたい人はこちらをチェック
| 項目名 | 説明 |
|---|---|
| group_PDB | 原子群の種類(ATOM/HETATM) |
| id | 各原子の一意の識別子 |
| type_symbol | 原子の元素記号 |
| label_atom_id | PDBがつけた原子の名前 |
| label_alt_id | 代替位置の識別子 |
| label_comp_id | 残基名 |
| label_asym_id | ラベリングによるChain ID |
| label_entity_id | 分子実体の識別子 |
| label_seq_id | ラベリングによる残基番号 |
| pdbx_PDB_ins_code | PDB insertion code |
| Cartn_x | X座標 |
| Cartn_y | Y座標 |
| Cartn_z | Z座標 |
| occupancy | 原子の占有率 |
| B_iso_or_equiv | B factor |
| pdbx_formal_charge | 形式電荷 |
| auth_seq_id | 著者指定の残基番号 |
| auth_comp_id | 著者指定の残基名 |
| auth_asym_id | 著者指定の鎖ID |
| auth_atom_id | 著者指定の原子名 |
| pdbx_PDB_model_num | モデル番号 |
label vs auth
基本的にlabel_となっているitemは機械的なラベリングによる識別子で、auth_は著者側がつけた識別子になります。
特に注意が必要なのはasym_id(所謂ChainID)です。論文やChimeraXなどのソフトウェアなどでは多くの場合auth_asym_idがChainIDとして扱われています。ただ一方で、バイオインフォマティクスなどのシステマチックな処理の場合、label_asym_idを参照した方がいい場合もあります。以下にその違いをまとめてみた。参考
auth(著者/PDB割り当てシステム)
- 由来:
- ポリマー鎖(タンパク質/DNA/RNA)→ 著者が提供
- 多糖類鎖 → PDBが割り当て
- 非ポリマー/溶媒 → PDBが割り当て
- 特徴:
- 近接する分子同士で同じチェインIDを共有することがある
- 特に溶媒やリガンドは、近くのポリマー鎖のIDに合わせて割り当てられる
- 多くの可視化プログラムで使用される
- PDBフォーマットの座標ファイルで使用される標準的な方式
label(連続ラベリングシステム)
- 由来:
- すべての成分に対してシステマティックに割り当て
- 特徴:
- アルファベット順の連続的な割り当て(A→Z→AA→BA...)
- 各成分(ポリマー/非ポリマー)に独自のIDを付与
- 溶媒とリガンドも独立したIDを持つ
- バイオインフォマティクス applications向けに最適化
- より体系的で一貫性のある識別が可能
せっかくなので、この違いをDataFrameを使って可視化してみましょう
from gemmi import cif
import pandas as pd
doc = cif.read('1alk.cif.gz')
block = doc.sole_block()
df = pd.DataFrame(block.get_mmcif_category('_atom_site.'))
diff_asym = df[df['label_asym_id'] != df['auth_asym_id']] # label vs auth
diff_asym[[ #表示するカラムを制限
'id',
'label_comp_id',
'label_asym_id',
'auth_asym_id',
'label_seq_id',
'auth_seq_id',
'label_atom_id',
'auth_atom_id'
]]
polarsの場合
from gemmi import cif
import polars as pl
doc = cif.read('1alk.cif.gz')
block = doc.sole_block()
df = (pl.DataFrame(block.get_mmcif_category('_atom_site.'), strict=False)
.filter(pl.col('label_asym_id') != pl.col('auth_asym_id'))
.select(['id',
'label_comp_id',
'label_asym_id',
'auth_asym_id',
'label_seq_id',
'auth_seq_id',
'label_atom_id',
'auth_atom_id'])
)
df

ご覧の通りイオンや溶媒、水分子などは、authの場合近くのChainに属してますが、labelの場合個々のインスタンスに対して割り当てられています。他にも、seq_idはlabelの場合、polymerでないと割り当てられていません。
viewerなどで見ると一目瞭然です。

Chainごとに色付けした1alk(左がauth, 右がauthをlabelで置き換えたもの)
水分子や溶媒などの分け方が異なっているのがわかります。
atom_siteのitemについてなんとなくわかってきたと思いますので実際にデータの解析をしてみましょう。
AlphaFold Serverの予測結果CIFファイルを解析してみる
AlphaFold3/AlphaFold Server の結果もCIFファイルで出力されます。このデータ参照してデータ解析をしてみよう。
ちなみに、AF ServerやAFDBのCIFファイルはModelCIFと呼ばれるdictで書かれています。_maで始まるカテゴリが ModelCIF特有のカラムになっています。
今回は例として、AF ServerのexampleになっているProtein-RNA-Ion: PDB 8AW3 を使ってみましょう。
exampleを押して実行後、結果のzipファイルをダウンロード&解凍して、解析を実行するディレクトリに解凍しておいてください。解凍後のdir名はexamplefold_pdb_8aw3になっていると思います。結果フォルダに入っているjosnファイルを使っても解析はできますが、今回はあえてcifファイルで解析していきましょう。
global plDDT
まず、global plDDTを取得してみましょう。global plDDTは_ma_qa_metric_global.metric_value (key-value型)に書かれていますので、今回はfind_mmcif_categoryではなくfind_valueを使って取り出してみましょう。
import pandas as pd
from gemmi import cif
def block2df(block:cif.Block, category_name:str) -> pd.DataFrame:
return pd.DataFrame(block.get_mmcif_category(category_name))
path = 'fold_protein_rna_ion_pdb_8aw3/fold_protein_rna_ion_pdb_8aw3_model_0.cif'
block = cif.read(path).sole_block()
block.find_value('_ma_qa_metric_global.metric_value')
'71.97'
model_0のglobal plDDTは71.97みたいです。ちなみにAF serverの場合は_ma_qa_metric_local はありません(AFDBのCIFファイルにはあります)。
local plDDT
各原子(AFDBの場合は各残基)のplDDTは座標情報が入ってる_atom_siteの、B_iso_or_equiv のカラムに入っています(本来はB factorが入ってるカラム)。
とりあえず、結果のmodel_0.cifを読み込んで、_atom_siteと_entity_polyのdataframeを作りましょう。ついでに面倒なので、block2dfという関数もつくちゃいましょう。
import pandas as pd
from gemmi import cif
def block2df(block:cif.Block, category_name:str) -> pd.DataFrame:
return pd.DataFrame(block.get_mmcif_category(category_name))
path = 'fold_protein_rna_ion_pdb_8aw3/fold_protein_rna_ion_pdb_8aw3_model_0.cif'
block = cif.read(path).sole_block()
atoms = block2df(block, '_atom_site.')
entity_poly = block2df(block, '_entity_poly.')
entity_polyの中身を見てpolymerを探してみます。
entity_poly
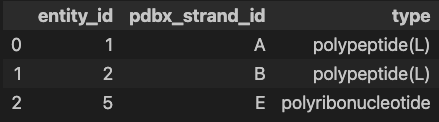
pdbx_strand_id はatom_siteのauth_asym_idと同じ意味でPDBが割り当てたChainIDになります(参考)。
A, B にポリペプチド鎖(タンパク質)Eにポリヌクレオチド鎖(今回はRNA)が入っていることがわかります。今回はChain A(auth_asym_idがA) の鎖のplDDTを解析してみましょう。
AF serverの場合、label_asym_idとauth_asym_idは同一のidが当てられています(多分)のでどちらでフィルターしても大丈夫です。
atoms[atoms['label_asym_id'] != atoms['auth_asym_id']].empty # 一応確認
>> True
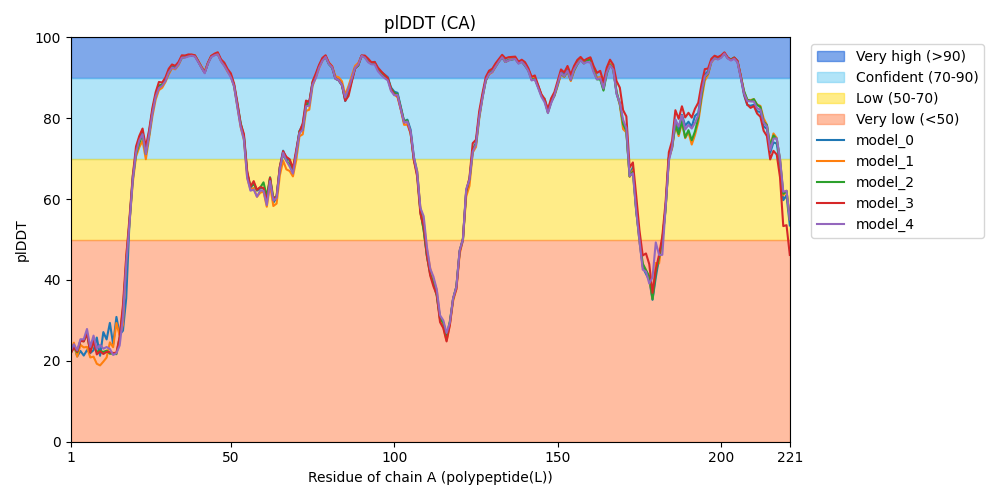
Chain AのCA原子を取り出して、残基番号ごとのplDDTの推移を折れ線グラフで可視化してみましょう。
chainA = atoms[(atoms['label_asym_id'] == 'A') & (atoms['label_atom_id'] == 'CA')]
chainA = chainA.astype({# str型になっているので型変更
'B_iso_or_equiv': float,
'label_seq_id': int
})
import matplotlib.pyplot as plt
import seaborn as sns
fig, ax = plt.subplots(figsize=(10, 5))
# 背景をplDDTカラーにしてわかりやすく
ax.axhspan(90, 100, color='#0053D6', alpha=0.5, label='Very high (>90)')
ax.axhspan(70, 90, color='#65CBF3', alpha=0.5, label='Confident (70-90)')
ax.axhspan(50, 70, color='#FFDB13', alpha=0.5, label='Low (50-70)')
ax.axhspan(0, 50, color='#FF7D45', alpha=0.5, label='Very low (<50)')
# plDDTをプロット
sns.lineplot(data=chainA, x='label_seq_id', y='B_iso_or_equiv', ax=ax, lw=2)
# この辺はお好みで
ax.set_ylim(0, 100)
ticks = np.unique(np.append(ax.get_xticks(), [chainA['label_seq_id'].min(), chainA['label_seq_id'].max()]))
ax.set_xticks(ticks)
ax.set_xlim(chainA['label_seq_id'].min(), chainA['label_seq_id'].max())
ax.set_xlabel('Residue of chain A')
ax.set_ylabel('plDDT')
ax.set_title('fold_protein_rna_ion_pdb_8aw3_model_0')
plt.tight_layout()
plt.show()

おおおお!!なんかいい感じのグラフができました!!こうしてみると予測信頼度の高い部分が4~5つくらいの山になっていますね!!ChimeraXなどを使って実際の構造と比べてみましょう!
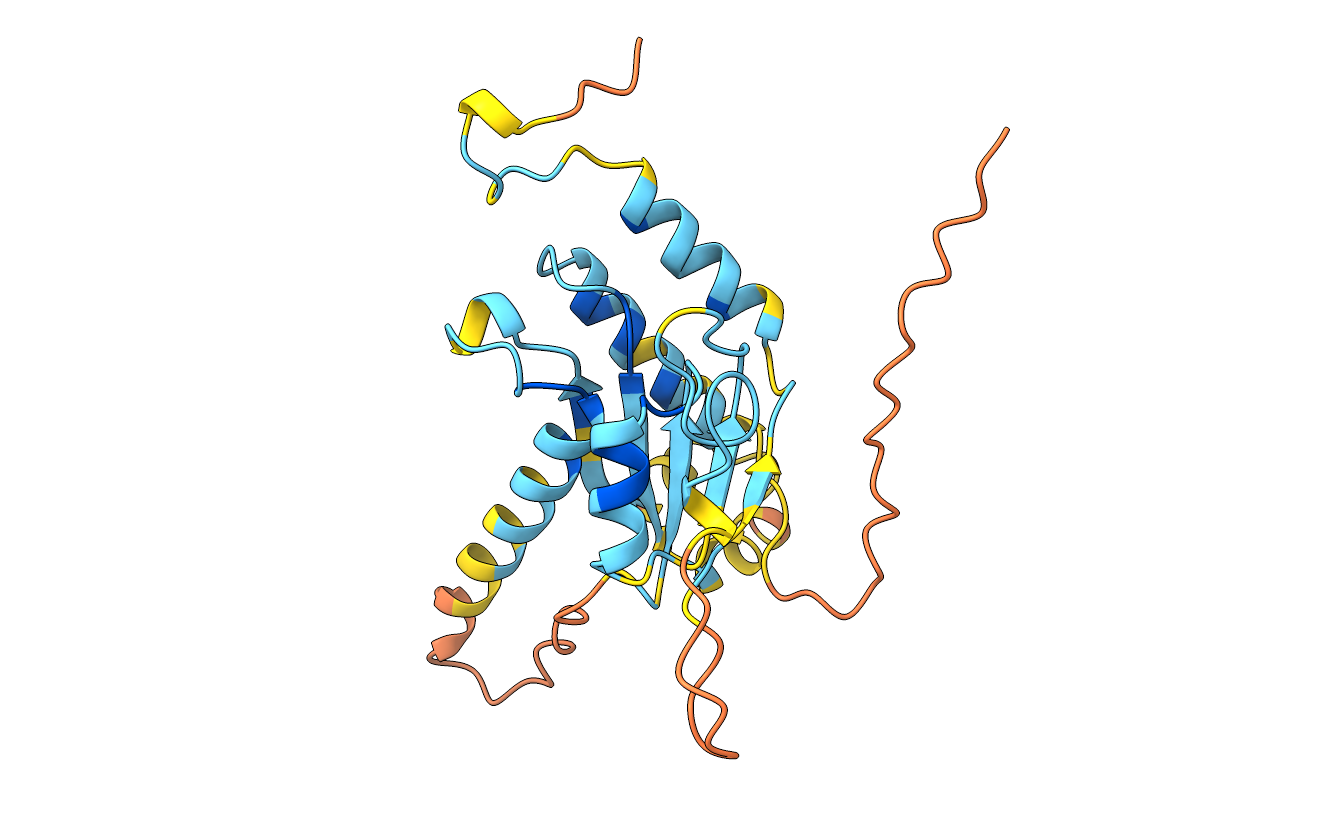
plDDTカラーコマンドに関してはこちらの記事を参照してください。
実際の構造をみると水色以上の領域がちょうど4~5つあるのがわかります。しかもhelixやsheetの部分に対応していることもわかりますね!他にも色々考察ができそうです。
これをみて分かると思いますがグラフで得られる情報と構造を見て分かる情報は結構印象が異なります。 構造だけ見て満足せずに、グラフ等をプロットしてあげることも重要です。
さて、AF3からは残基ごとではなく、atomごとにplDDTが割り当てられています。
なので、
- 各残基の原子のうち一番小さいplDDTの原子のplDDT:plDDT_min
- 各残基の原子のうち一番大きいplDDTの原子のplDDT:plDDT_max
- 各残基各原子のplDDTの平均値:plDDT_mean
- CAのplDDT:plDDT_ca
の4つをプロットして違いを見てみましょう。
def block2df(block:cif.Block, category_name:str) -> pd.DataFrame:
return pd.DataFrame(block.get_mmcif_category(category_name))
path = 'fold_protein_rna_ion_pdb_8aw3/fold_protein_rna_ion_pdb_8aw3_model_0.cif'
block = cif.read(path).sole_block()
atoms = block2df(block, '_atom_site.'
).astype({'B_iso_or_equiv': float, 'label_seq_id': int}
).rename(columns={'B_iso_or_equiv': 'plDDT'})
chainA = atoms[atoms['label_asym_id'] == 'A']
chainA = chainA.groupby('label_seq_id').agg( #groupbyを使って集計
plDDT_mean=('plDDT', 'mean'),
plDDT_min=('plDDT', 'min'),
plDDT_max=('plDDT', 'max'),
plDDT_CA=(
'plDDT',
lambda x: x[chainA['label_atom_id'] == 'CA'].iloc[0] if len(x[chainA['label_atom_id'] == 'CA']) > 0 else None
)
).reset_index()
chainA
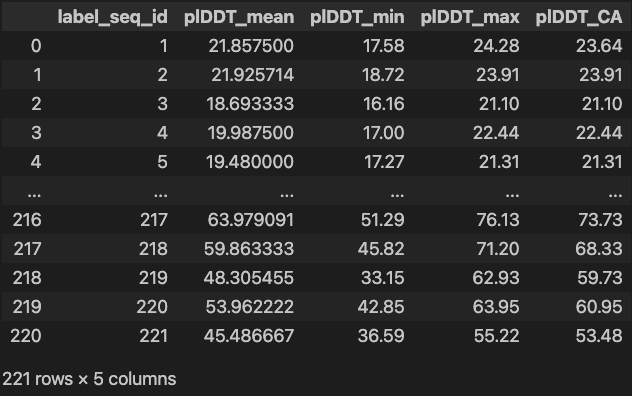
いい感じに集計できていますね、これを先ほどの要領でplotしてみましょう。
import matplotlib.pyplot as plt
import seaborn as sns
import numpy as np
# Create the plot
fig, ax = plt.subplots(figsize=(10, 5))
# Add colored background regions
ax.axhspan(90, 100, color='#0053D6', alpha=0.5, label='Very high (>90)')
ax.axhspan(70, 90, color='#65CBF3', alpha=0.5, label='Confident (70-90)')
ax.axhspan(50, 70, color='#FFDB13', alpha=0.5, label='Low (50-70)')
ax.axhspan(0, 50, color='#FF7D45', alpha=0.5, label='Very low (<50)')
# Plot the line
sns.lineplot(data=chainA, x='label_seq_id', y='plDDT_mean', ax=ax, lw=1, label='Mean')
sns.lineplot(data=chainA, x='label_seq_id', y='plDDT_min', ax=ax, lw=1, label='Min')
sns.lineplot(data=chainA, x='label_seq_id', y='plDDT_max', ax=ax, lw=1, label='Max')
sns.lineplot(data=chainA, x='label_seq_id', y='plDDT_CA', ax=ax, lw=1, label='CA')
# Customize the plot
ax.set_ylim(0, 100)
ticks = np.unique(np.append(ax.get_xticks(), [chainA['label_seq_id'].min(), chainA['label_seq_id'].max()]))
ax.set_xticks(ticks)
ax.set_xlim(chainA['label_seq_id'].min(), chainA['label_seq_id'].max())
ax.set_xlabel('Residue')
ax.set_ylabel('plDDT')
ax.set_title('plDDT per residue of chain A')
# Add legend
ax.legend(bbox_to_anchor=(1.02, 1))
# Adjust layout to prevent legend cutoff
plt.tight_layout()
plt.show()

できました!重なっててみづらいですが、maxとCAがほとんど同じだったり、CAは良くてもminになると結構落ち込んでいるところがあるのがわかりますね。
最後に特定のchainのmodelごとのplddtの推移を出力するプログラムを作成してみましょう。
import pandas as pd
import gemmi
from gemmi import cif
import matplotlib.pyplot as plt
import seaborn as sns
import numpy as np
from typing import Literal
import typer
def plot_init(figsize=(10, 5)):
fig, ax = plt.subplots(figsize=figsize)
ax.axhspan(90, 100, color='#0053D6', alpha=0.5, label='Very high (>90)')
ax.axhspan(70, 90, color='#65CBF3', alpha=0.5, label='Confident (70-90)')
ax.axhspan(50, 70, color='#FFDB13', alpha=0.5, label='Low (50-70)')
ax.axhspan(0, 50, color='#FF7D45', alpha=0.5, label='Very low (<50)')
return fig, ax
def plot_customize(data:pd.DataFrame, x:str, ax:plt.Axes, filtering:str, chain_id:str, asym2entity:dict=None):
if x == 'label_seq_id':
ax.set_xticks(np.unique(np.append(ax.get_xticks(), [data[x].min(), data[x].max()])))
ax.set_xlim(data[x].min(), data[x].max())
ax.set_xlabel(f'Residue of chain {chain_id} ({asym2entity["poly_type"]})')
ax.set_title(f'plDDT ({filtering})')
else:
ax.set_xlabel(f'Atom of chain {chain_id} ({asym2entity["entity_type"]})')
ax.set_title(f'plDDT')
ax.set_ylim(0, 100)
ax.set_ylabel(f'plDDT')
ax.legend(bbox_to_anchor=(1.02, 1))
plt.tight_layout()
return ax
def block2df(block:cif.Block, category:str):
return pd.DataFrame(block.get_mmcif_category(category))
def get_asym2entity(block:cif.Block):
# {'A': {'entity_type': 'polymer', 'poly_type': 'polypeptide(L)'},
# 'B': {'entity_type': 'non-polymer', 'poly_type': nan},
# 'C': {'entity_type': 'branched', 'poly_type': nan},
# 'D': {'entity_type': 'branched', 'poly_type': nan},
# 'E': {'entity_type': 'branched', 'poly_type': nan},
# 'F': {'entity_type': 'branched', 'poly_type': nan},
# 'G': {'entity_type': 'branched', 'poly_type': nan},
# 'H': {'entity_type': 'branched', 'poly_type': nan}}
asym2entity = (
block2df(block, '_entity.')
.rename(columns={'id': 'entity_id', 'type': 'entity_type'})
.drop(columns=['pdbx_description'])
.merge(block2df(block, '_struct_asym.').rename(columns={'id': 'asym_id'}),on='entity_id')
.merge(block2df(block, '_entity_poly.').rename(columns={'type': 'poly_type'}).drop(columns=['pdbx_strand_id']),
on='entity_id', how='left')
.drop(columns=['entity_id'])
.set_index('asym_id')
.to_dict(orient='index')
)
return asym2entity
def plot_plddt(asym2entity:dict, filtering:str) -> tuple:
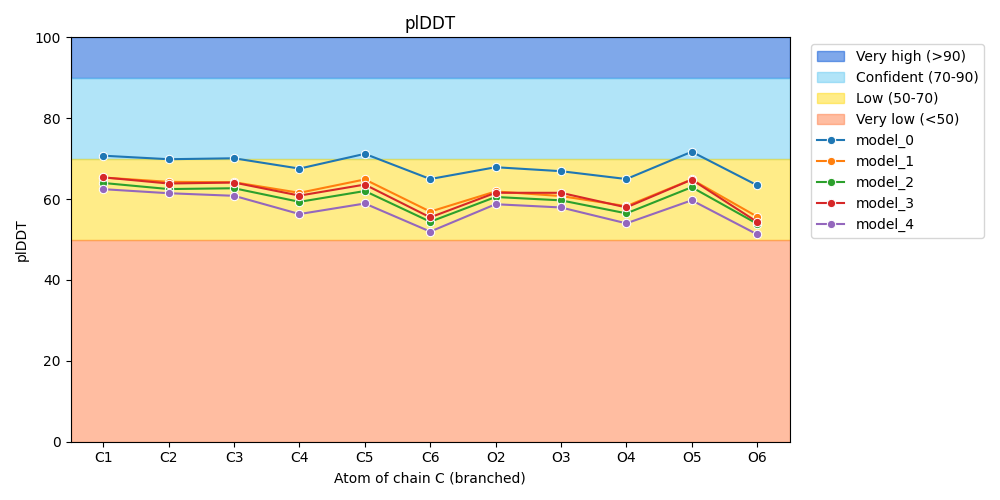
if asym2entity['entity_type'] in ['non-polymer', 'branched']:
xy = {'x': 'label_atom_id', 'y': 'B_iso_or_equiv'}
return lambda atoms,ax,label: sns.lineplot(**xy, data=atoms, label=label, ax=ax, marker='o'), xy
elif asym2entity['entity_type'] == 'polymer':
xy = {'x': 'label_seq_id', 'y': 'B_iso_or_equiv'}
if filtering == 'CA':
if asym2entity['poly_type'] == 'polypeptide(L)':
return lambda atoms,ax,label: sns.lineplot(**xy, data=atoms[atoms['label_atom_id'] == "CA"], label=label, ax=ax), xy
elif asym2entity['poly_type'] == 'polyribonucleotide':
return lambda atoms,ax,label: sns.lineplot(**xy, data=atoms[atoms['label_atom_id'] == "C4'"], label=label, ax=ax), xy
else:
raise ValueError('Invalid poly_type')
elif filtering == 'mean':
return lambda atoms,ax,label: sns.lineplot(**xy, data=atoms.groupby('label_seq_id').mean().reset_index(), label=label, ax=ax), xy
elif filtering == 'min':
return lambda atoms,ax,label: sns.lineplot(**xy, data=atoms.groupby('label_seq_id').min().reset_index(), label=label, ax=ax), xy
elif filtering == 'max':
return lambda atoms,ax,label: sns.lineplot(**xy, data=atoms.groupby('label_seq_id').max().reset_index(), label=label, ax=ax), xy
else:
raise ValueError('Invalid filtering')
else:
raise ValueError('Invalid entity_type')
def plot_plddt_per_models(cif_dir:str, chain_id:str, filtering=Literal['CA', 'mean', 'min', 'max']):
files = sorted(list(gemmi.CifWalk(cif_dir)))
fig, ax = plot_init()
for i,path in enumerate(files):
block = cif.read(path).sole_block()
if i == 0:
asym2entity = get_asym2entity(block)
if chain_id not in asym2entity:
raise ValueError(f'Invalid chain_id: {chain_id}')
ploter,xy = plot_plddt(asym2entity[chain_id], filtering)
atoms = block2df(block, '_atom_site').astype({'B_iso_or_equiv': float, 'label_seq_id': int})
atoms = atoms[atoms['label_asym_id'] == chain_id]
ax = ploter(atoms, ax, f'model_{i}')
plt.savefig(f'test_{i}.png')
return plot_customize(atoms, xy['x'], ax, filtering, chain_id, asym2entity[chain_id])
def main(cif_dir:str, chain_id:str, filtering:str='CA', save_path:str|None=None):
plot_plddt_per_models(cif_dir, chain_id, filtering)
if save_path:
plt.savefig(save_path)
plt.show()
if __name__ == '__main__':
typer.run(main)
いかがでしょうか、このスクリプトでは引数として入力されたchain_idがpolymerなのかnon-polymerなのか、polymerならpolypeptideなのかpolyribonucleotideなのかを、asym2entityという辞書を作って判定しています。dataframeを作ることでmerge(join)が楽にできますね。
また、gemmi.CifWalk(cif_dir)は引数のdir内のcifファイルを再帰的に取得してくれるクラスです。dir内のcifファイルをまとめて処理する時にとっても便利です!!
このスクリプトを実行すると
python gemmi_table.py fold_protein_rna_ion_pdb_8aw3 A
python gemmi_table.py fold_protein_glycan_ion_pdb_7bbv C #Cはglycan
このようにdataframeにすることでデータサイエンスをやっている人には慣れ親しんだ操作でcifファイルを解析することができます!!
CIFファイルの書き出し
最後にCIFファイルを編集して書き出すってことをやってみましょう。今回もAF ServerのCIFファイルを使ってみます。
複数のCIFファイルを結合して一つのCIFファイルにする
まずは簡単なところから 複数のCIFファイルを結合した一つのCIFファイルを作ってみましょう。
document(もしくはblock)クラスからCIFファイルを書き出すには.write_file メソッドを使います。
import os
import gemmi
from gemmi import cif
result_dir = 'fold_protein_glycan_ion_pdb_7bbv' #af3のresultディレクトリ
doc = cif.Document() # 空のCIFドキュメントを作成
for path in gemmi.CifWalk(result_dir): #resultディレクトリ内のCIFファイルを読み込む
block = cif.read(path).sole_block()
block.name = block.name + '_model_' + os.path.basename(path).split('.')[0].split('_')[-1] #モデル番号を追加
doc.add_copied_block(block) #CIFファイルをドキュメントに追加
doc.write_file(os.path.join('results.cif')) #結果をresults.cifとして保存
len(cif.read(os.path.join('results.cif')))
>>5
簡単ですね!
一応やっていることを解説すると
- 空のDocumentを作る
- CifWalkを使ってdir内の全てのcifファイルを再帰的に取得
- blockを取得
- 取得したblockのnameプロパティ(CIFファイル内の
data_***に該当)をmodel番号を含むように変更する(model間で同じnameになってしまっているため)。 - DocumentにAdd
- 最後にwrite_fileで書き出し
という感じです。
CIFファイルに情報を追加して書き出し
もちろんですが、CIFファイルに情報を追加/削除することもできます。
解析した日付や内容を_edit_logという新しいカテゴリを作って書いてみましょう。key-valueの追加にはset_pairsメソッドを使います。
import gemmi
from gemmi import cif
from datetime import datetime
path = 'fold_protein_glycan_ion_pdb_7bbv/fold_protein_glycan_ion_pdb_7bbv_model_0.cif'
block = cif.read(path).sole_block()
block.set_pairs('_edit_log.', { 'id': '1',
'date': datetime.now().strftime('%Y-%m-%d'),
'description': 'This is a test',
'software': 'gemmi',
'version': gemmi.__version__,})
block.write_file('test.cif')
書き出したファイルの末尾を見てみると…
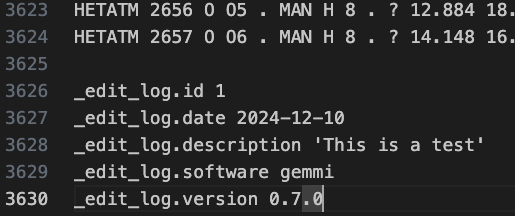
_edit_logが追加されていますね!
今度はloop型を追加してみましょう。loop型の場合はset_mmcif_categoryを使います。
from gemmi import cif
import gemmi
import datetime
path = 'fold_protein_glycan_ion_pdb_7bbv/fold_protein_glycan_ion_pdb_7bbv_model_0.cif'
block = cif.read(path).sole_block()
block.set_mmcif_category('_edit_log.', {
'id': ['1', '2', '3'],
'date': [datetime.datetime.now().strftime('%Y-%m-%d')]*3,
'description': [f'Test {i}' for i in range(3)],
'software': ['gemmi']*3,
'version': [gemmi.__version__]*3,})
block.write_file('test.cif
中身を見ると…

loop型が追加されていますね!!
これを応用することで、例えばSASAなどの他で計算した値を書き込んだり、足りない情報を追加しておいたりすることができます!
AF3のCIFファイルにentityの情報を足してみる
AF3で取得したCIFファイルにentityの情報を追加して書き出してみましょう。
とりあえず、各entityについて調べてみましょう。
from gemmi import cif
import pandas as pd
def block2df(block:cif.Block, category_name:str) -> pd.DataFrame:
return pd.DataFrame(block.get_mmcif_category(category_name))
def get_entity_info(block:cif.Block) -> pd.DataFrame:
entity = (block2df(block, '_entity.')
.rename(columns={'id': 'entity_id', 'type': 'entity_type'})
.merge(block2df(block, '_struct_asym.') #chainの情報を追加
.rename(columns={'id': 'asym_id'}),
on='entity_id')
.merge(block2df(block, '_entity_poly.') # polymerの情報を追加
.rename(columns={'type': 'poly_type'})
.drop(columns=['pdbx_strand_id']),
on='entity_id', how='left')
.merge(block2df(block, '_entity_poly_seq') # polmerのseqの長さを追加
.groupby('entity_id')
.size()
.reset_index(name='seq_length'),
on='entity_id', how='left')
)
return entity[['entity_id', 'pdbx_description','entity_type','asym_id','poly_type','seq_length']]
path = 'fold_protein_rna_ion_pdb_8aw3/fold_protein_rna_ion_pdb_8aw3_model_0.cif'
block = cif.read(path).sole_block()
entity = get_entity_info(block)
entity

するとこんな感じになりました。ローカル版のAF3ではchain IDが指定できるのでここまでやる必要はないですが、今回はServerなのでどれがどのchainになるのかよくわからないのでPDBのエントリーをカンニングしちゃいましょう。
元になってる8aw3.cifをダウンロードして可視化
path = '/Users/nagaet/qiita/8aw3.cif.gz'
entity_8aw3 = get_entity_info(cif.read(path).sole_block()
).drop(columns=['asym_id']
).drop_duplicates()
entity_8aw3
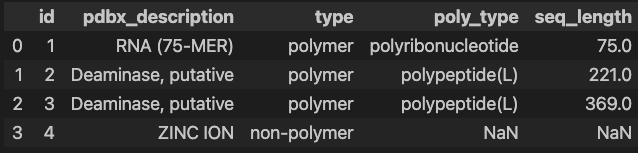
なんとなく対応関係がわかりました。少し面倒ですが、元のentity_idとAFのentity_idのmappingを作りましょう。
entity2base_entity = {
'1':'2',
'2':'3',
'3':'4',
'4':'4',
'5':'1',
}
これをentityに足して、pdbx_descriptionを追加しましょうか
entity = (entity
.merge(entity_8aw3[['id', 'pdbx_description']], left_on='base_entity_id', right_on='id')
.drop(columns=['id_y', 'pdbx_description_x', 'base_entity_id'])
.rename(columns={'id_x': 'id', 'pdbx_description_y': 'pdbx_description'})
)
entity
情報が補完できました!!block内のentityを書き換えてCIFファイルに書き出してみると…
entity = entity.to_dict(orient='list')
block.set_mmcif_category('_entity.', entity)
block.write_file('test.cif')

変更されていますね!!
この編集したCIFファイルをChimeraXで開くと…
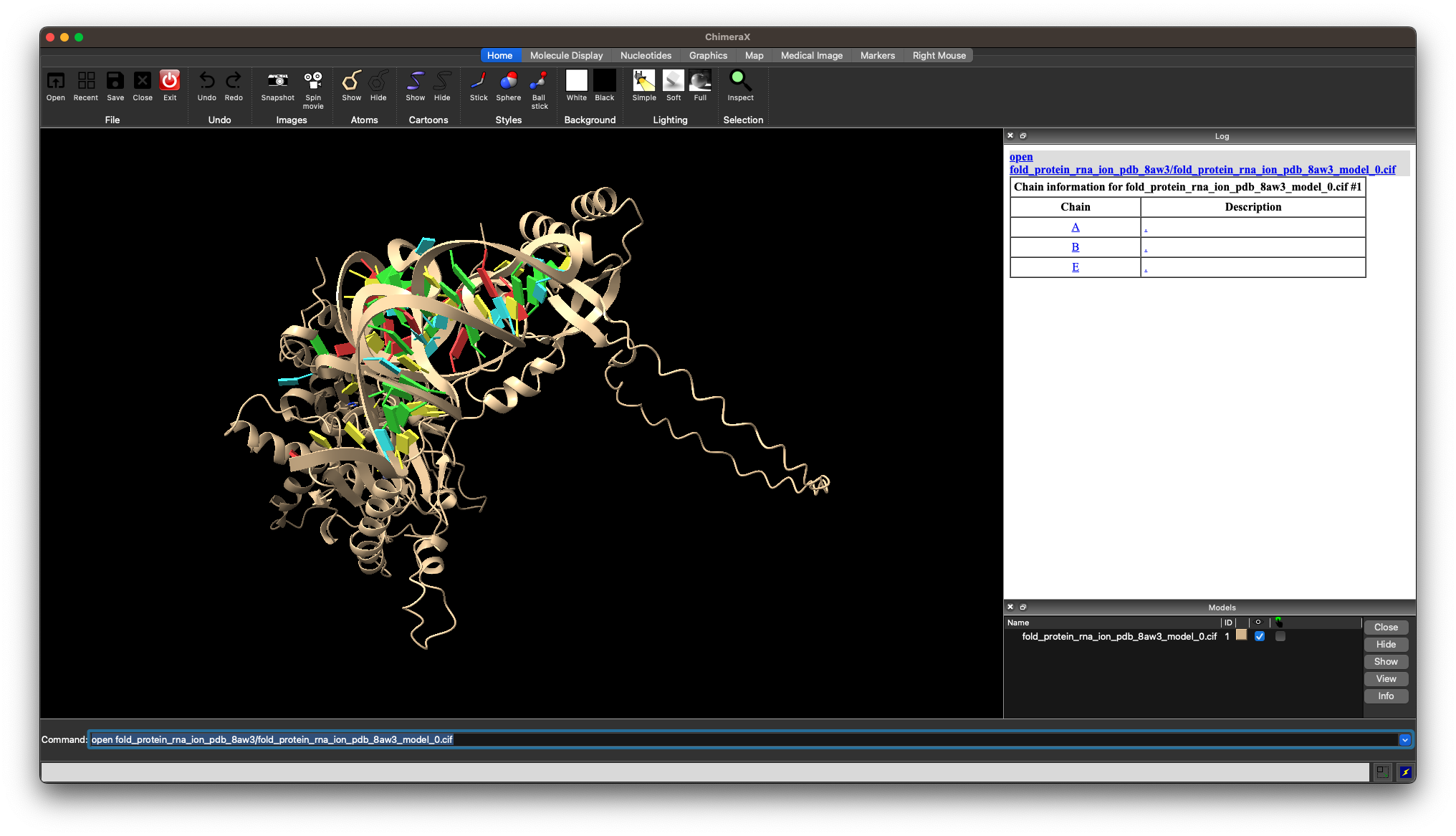
書き込む前のファイルのDescriptionは何もないけど…
座標情報をDataFrameでごり押し編集
もちろん、DataFrameを使って座標情報を編集することもできます。
auth_asym_idをlabel_asym_idに置き換える
labelのが便利なことがあるのは前述しましたので、実際に置き換えてみましょう。
from gemmi import cif
block = cif.read('1alk.cif.gz').sole_block()
atoms = block.get_mmcif_category('_atom_site.') # dataframe使う必要ないのでdictで
atoms['auth_asym_id'] = atoms['label_asym_id']
block.set_mmcif_category('_atom_site.', atoms)
block.write_file('1alk_label.cif')
これをviewerでchainごとに色付けして見てみましょう。

左がauthのまま、右がlabelに置き換えた場合です。こうすると一目瞭然ですね!!
次は座標情報をCA原子だけにしてみましょう。
from gemmi import cif
def block2df(block:cif.Block, category_name:str) -> pd.DataFrame:
return pd.DataFrame(block.get_mmcif_category(category_name))
block = cif.read('1alk.cif.gz').sole_block()
atoms = block2df(block, '_atom_site.')
atoms = atoms[atoms['label_atom_id'] == 'CA']
block.set_mmcif_category('_atom_site.', atoms.to_dict(orient='list'))
block.write_file('1alk_CA.cif')
座標をいじる際は他のカテゴリなどはそのままになることに注意してください!!!
おわりに
いかがでしたでしょうか。一見よくわからないCIFファイルの中身がDataFrameになるだけでとっつきやすくなったんじゃないでしょうか。
CIFファイルは座標だけでなく色々な情報が含まれていますので、是非活用してみてください!!
また、GEMMIにもたくさんの機能があります。ぜひ触ったり、公式ドキュメントを見てみてください!!構造解析のモードの方も余裕があったら書きたいと思ってます!!
コピーしてChimeraXで開いてみてね!
data_message
loop_
_atom_site.group_PDB
_atom_site.id
_atom_site.type_symbol
_atom_site.label_atom_id
_atom_site.label_alt_id
_atom_site.label_comp_id
_atom_site.label_asym_id
_atom_site.label_entity_id
_atom_site.label_seq_id
_atom_site.pdbx_PDB_ins_code
_atom_site.occupancy
_atom_site.B_iso_or_equiv
_atom_site.pdbx_formal_charge
_atom_site.auth_seq_id
_atom_site.auth_comp_id
_atom_site.auth_asym_id
_atom_site.auth_atom_id
_atom_site.pdbx_PDB_model_num
_atom_site.Cartn_x
_atom_site.Cartn_y
_atom_site.Cartn_z
ATOM 1 O O '.' UNL A 1 1 '.' 1.0 20.0 0 1 UNL A O 1 3.3 9.0 0
ATOM 2 O O '.' UNL A 1 2 '.' 1.0 20.0 0 2 UNL A O 1 5.1 9.0 0
ATOM 3 O O '.' UNL A 1 3 '.' 1.0 20.0 0 3 UNL A O 1 5.3999999999999995 9.0 0
ATOM 4 O O '.' UNL A 1 4 '.' 1.0 20.0 0 4 UNL A O 1 5.7 9.0 0
ATOM 5 O O '.' UNL A 1 5 '.' 1.0 20.0 0 5 UNL A O 1 6.0 9.0 0
ATOM 6 O O '.' UNL A 1 6 '.' 1.0 20.0 0 6 UNL A O 1 6.8999999999999995 9.0 0
ATOM 7 O O '.' UNL A 1 7 '.' 1.0 20.0 0 7 UNL A O 1 7.199999999999999 9.0 0
ATOM 8 O O '.' UNL A 1 8 '.' 1.0 20.0 0 8 UNL A O 1 7.5 9.0 0
ATOM 9 O O '.' UNL A 1 9 '.' 1.0 20.0 0 9 UNL A O 1 7.8 9.0 0
ATOM 10 O O '.' UNL A 1 10 '.' 1.0 20.0 0 10 UNL A O 1 8.1 9.0 0
ATOM 11 O O '.' UNL A 1 11 '.' 1.0 20.0 0 11 UNL A O 1 9.0 9.0 0
ATOM 12 O O '.' UNL A 1 12 '.' 1.0 20.0 0 12 UNL A O 1 9.9 9.0 0
ATOM 13 O O '.' UNL A 1 13 '.' 1.0 20.0 0 13 UNL A O 1 10.2 9.0 0
ATOM 14 O O '.' UNL A 1 14 '.' 1.0 20.0 0 14 UNL A O 1 10.5 9.0 0
ATOM 15 O O '.' UNL A 1 15 '.' 1.0 20.0 0 15 UNL A O 1 3.3 8.7 0
ATOM 16 O O '.' UNL A 1 16 '.' 1.0 20.0 0 16 UNL A O 1 5.1 8.7 0
ATOM 17 O O '.' UNL A 1 17 '.' 1.0 20.0 0 17 UNL A O 1 7.5 8.7 0
ATOM 18 O O '.' UNL A 1 18 '.' 1.0 20.0 0 18 UNL A O 1 9.0 8.7 0
ATOM 19 O O '.' UNL A 1 19 '.' 1.0 20.0 0 19 UNL A O 1 9.6 8.7 0
ATOM 20 O O '.' UNL A 1 20 '.' 1.0 20.0 0 20 UNL A O 1 10.799999999999999 8.7 0
ATOM 21 O O '.' UNL A 1 21 '.' 1.0 20.0 0 21 UNL A O 1 3.3 8.399999999999999 0
ATOM 22 O O '.' UNL A 1 22 '.' 1.0 20.0 0 22 UNL A O 1 5.1 8.399999999999999 0
ATOM 23 O O '.' UNL A 1 23 '.' 1.0 20.0 0 23 UNL A O 1 7.5 8.399999999999999 0
ATOM 24 O O '.' UNL A 1 24 '.' 1.0 20.0 0 24 UNL A O 1 9.0 8.399999999999999 0
ATOM 25 O O '.' UNL A 1 25 '.' 1.0 20.0 0 25 UNL A O 1 9.6 8.399999999999999 0
ATOM 26 O O '.' UNL A 1 26 '.' 1.0 20.0 0 26 UNL A O 1 3.3 8.1 0
ATOM 27 O O '.' UNL A 1 27 '.' 1.0 20.0 0 27 UNL A O 1 5.1 8.1 0
ATOM 28 O O '.' UNL A 1 28 '.' 1.0 20.0 0 28 UNL A O 1 7.5 8.1 0
ATOM 29 O O '.' UNL A 1 29 '.' 1.0 20.0 0 29 UNL A O 1 9.9 8.1 0
ATOM 30 O O '.' UNL A 1 30 '.' 1.0 20.0 0 30 UNL A O 1 10.2 8.1 0
ATOM 31 O O '.' UNL A 1 31 '.' 1.0 20.0 0 31 UNL A O 1 3.3 7.8 0
ATOM 32 O O '.' UNL A 1 32 '.' 1.0 20.0 0 32 UNL A O 1 5.1 7.8 0
ATOM 33 O O '.' UNL A 1 33 '.' 1.0 20.0 0 33 UNL A O 1 5.3999999999999995 7.8 0
ATOM 34 O O '.' UNL A 1 34 '.' 1.0 20.0 0 34 UNL A O 1 5.7 7.8 0
ATOM 35 O O '.' UNL A 1 35 '.' 1.0 20.0 0 35 UNL A O 1 6.0 7.8 0
ATOM 36 O O '.' UNL A 1 36 '.' 1.0 20.0 0 36 UNL A O 1 7.5 7.8 0
ATOM 37 O O '.' UNL A 1 37 '.' 1.0 20.0 0 37 UNL A O 1 10.5 7.8 0
ATOM 38 O O '.' UNL A 1 38 '.' 1.0 20.0 0 38 UNL A O 1 10.799999999999999 7.8 0
ATOM 39 O O '.' UNL A 1 39 '.' 1.0 20.0 0 39 UNL A O 1 3.3 7.5 0
ATOM 40 O O '.' UNL A 1 40 '.' 1.0 20.0 0 40 UNL A O 1 5.1 7.5 0
ATOM 41 O O '.' UNL A 1 41 '.' 1.0 20.0 0 41 UNL A O 1 7.5 7.5 0
ATOM 42 O O '.' UNL A 1 42 '.' 1.0 20.0 0 42 UNL A O 1 10.799999999999999 7.5 0
ATOM 43 O O '.' UNL A 1 43 '.' 1.0 20.0 0 43 UNL A O 1 3.3 7.2 0
ATOM 44 O O '.' UNL A 1 44 '.' 1.0 20.0 0 44 UNL A O 1 5.1 7.2 0
ATOM 45 O O '.' UNL A 1 45 '.' 1.0 20.0 0 45 UNL A O 1 7.5 7.2 0
ATOM 46 O O '.' UNL A 1 46 '.' 1.0 20.0 0 46 UNL A O 1 9.6 7.2 0
ATOM 47 O O '.' UNL A 1 47 '.' 1.0 20.0 0 47 UNL A O 1 10.799999999999999 7.2 0
ATOM 48 O O '.' UNL A 1 48 '.' 1.0 20.0 0 48 UNL A O 1 3.3 6.8999999999999995 0
ATOM 49 O O '.' UNL A 1 49 '.' 1.0 20.0 0 49 UNL A O 1 3.5999999999999996 6.8999999999999995 0
ATOM 50 O O '.' UNL A 1 50 '.' 1.0 20.0 0 50 UNL A O 1 3.9 6.8999999999999995 0
ATOM 51 O O '.' UNL A 1 51 '.' 1.0 20.0 0 51 UNL A O 1 4.2 6.8999999999999995 0
ATOM 52 O O '.' UNL A 1 52 '.' 1.0 20.0 0 52 UNL A O 1 4.5 6.8999999999999995 0
ATOM 53 O O '.' UNL A 1 53 '.' 1.0 20.0 0 53 UNL A O 1 5.1 6.8999999999999995 0
ATOM 54 O O '.' UNL A 1 54 '.' 1.0 20.0 0 54 UNL A O 1 5.3999999999999995 6.8999999999999995 0
ATOM 55 O O '.' UNL A 1 55 '.' 1.0 20.0 0 55 UNL A O 1 5.7 6.8999999999999995 0
ATOM 56 O O '.' UNL A 1 56 '.' 1.0 20.0 0 56 UNL A O 1 6.0 6.8999999999999995 0
ATOM 57 O O '.' UNL A 1 57 '.' 1.0 20.0 0 57 UNL A O 1 6.3 6.8999999999999995 0
ATOM 58 O O '.' UNL A 1 58 '.' 1.0 20.0 0 58 UNL A O 1 7.5 6.8999999999999995 0
ATOM 59 O O '.' UNL A 1 59 '.' 1.0 20.0 0 59 UNL A O 1 9.9 6.8999999999999995 0
ATOM 60 O O '.' UNL A 1 60 '.' 1.0 20.0 0 60 UNL A O 1 10.2 6.8999999999999995 0
ATOM 61 O O '.' UNL A 1 61 '.' 1.0 20.0 0 61 UNL A O 1 10.5 6.8999999999999995 0
ATOM 62 O O '.' UNL A 1 62 '.' 1.0 20.0 0 62 UNL A O 1 3.5999999999999996 4.8 0
ATOM 63 O O '.' UNL A 1 63 '.' 1.0 20.0 0 63 UNL A O 1 3.9 4.8 0
ATOM 64 O O '.' UNL A 1 64 '.' 1.0 20.0 0 64 UNL A O 1 4.2 4.8 0
ATOM 65 O O '.' UNL A 1 65 '.' 1.0 20.0 0 65 UNL A O 1 5.3999999999999995 4.8 0
ATOM 66 O O '.' UNL A 1 66 '.' 1.0 20.0 0 66 UNL A O 1 5.7 4.8 0
ATOM 67 O O '.' UNL A 1 67 '.' 1.0 20.0 0 67 UNL A O 1 6.0 4.8 0
ATOM 68 O O '.' UNL A 1 68 '.' 1.0 20.0 0 68 UNL A O 1 7.199999999999999 4.8 0
ATOM 69 O O '.' UNL A 1 69 '.' 1.0 20.0 0 69 UNL A O 1 8.4 4.8 0
ATOM 70 O O '.' UNL A 1 70 '.' 1.0 20.0 0 70 UNL A O 1 9.299999999999999 4.8 0
ATOM 71 O O '.' UNL A 1 71 '.' 1.0 20.0 0 71 UNL A O 1 10.5 4.8 0
ATOM 72 O O '.' UNL A 1 72 '.' 1.0 20.0 0 72 UNL A O 1 11.7 4.8 0
ATOM 73 O O '.' UNL A 1 73 '.' 1.0 20.0 0 73 UNL A O 1 12.9 4.8 0
ATOM 74 O O '.' UNL A 1 74 '.' 1.0 20.0 0 74 UNL A O 1 14.1 4.8 0
ATOM 75 O O '.' UNL A 1 75 '.' 1.0 20.0 0 75 UNL A O 1 14.7 4.8 0
ATOM 76 O O '.' UNL A 1 76 '.' 1.0 20.0 0 76 UNL A O 1 15.899999999999999 4.8 0
ATOM 77 O O '.' UNL A 1 77 '.' 1.0 20.0 0 77 UNL A O 1 17.4 4.8 0
ATOM 78 O O '.' UNL A 1 78 '.' 1.0 20.0 0 78 UNL A O 1 18.3 4.8 0
ATOM 79 O O '.' UNL A 1 79 '.' 1.0 20.0 0 79 UNL A O 1 19.2 4.8 0
ATOM 80 O O '.' UNL A 1 80 '.' 1.0 20.0 0 80 UNL A O 1 3.3 4.5 0
ATOM 81 O O '.' UNL A 1 81 '.' 1.0 20.0 0 81 UNL A O 1 4.5 4.5 0
ATOM 82 O O '.' UNL A 1 82 '.' 1.0 20.0 0 82 UNL A O 1 5.1 4.5 0
ATOM 83 O O '.' UNL A 1 83 '.' 1.0 20.0 0 83 UNL A O 1 6.3 4.5 0
ATOM 84 O O '.' UNL A 1 84 '.' 1.0 20.0 0 84 UNL A O 1 7.199999999999999 4.5 0
ATOM 85 O O '.' UNL A 1 85 '.' 1.0 20.0 0 85 UNL A O 1 8.4 4.5 0
ATOM 86 O O '.' UNL A 1 86 '.' 1.0 20.0 0 86 UNL A O 1 9.299999999999999 4.5 0
ATOM 87 O O '.' UNL A 1 87 '.' 1.0 20.0 0 87 UNL A O 1 10.5 4.5 0
ATOM 88 O O '.' UNL A 1 88 '.' 1.0 20.0 0 88 UNL A O 1 11.4 4.5 0
ATOM 89 O O '.' UNL A 1 89 '.' 1.0 20.0 0 89 UNL A O 1 11.7 4.5 0
ATOM 90 O O '.' UNL A 1 90 '.' 1.0 20.0 0 90 UNL A O 1 12.9 4.5 0
ATOM 91 O O '.' UNL A 1 91 '.' 1.0 20.0 0 91 UNL A O 1 13.799999999999999 4.5 0
ATOM 92 O O '.' UNL A 1 92 '.' 1.0 20.0 0 92 UNL A O 1 14.7 4.5 0
ATOM 93 O O '.' UNL A 1 93 '.' 1.0 20.0 0 93 UNL A O 1 15.899999999999999 4.5 0
ATOM 94 O O '.' UNL A 1 94 '.' 1.0 20.0 0 94 UNL A O 1 17.4 4.5 0
ATOM 95 O O '.' UNL A 1 95 '.' 1.0 20.0 0 95 UNL A O 1 18.3 4.5 0
ATOM 96 O O '.' UNL A 1 96 '.' 1.0 20.0 0 96 UNL A O 1 19.2 4.5 0
ATOM 97 O O '.' UNL A 1 97 '.' 1.0 20.0 0 97 UNL A O 1 3.3 4.199999999999999 0
ATOM 98 O O '.' UNL A 1 98 '.' 1.0 20.0 0 98 UNL A O 1 4.8 4.199999999999999 0
ATOM 99 O O '.' UNL A 1 99 '.' 1.0 20.0 0 99 UNL A O 1 6.6 4.199999999999999 0
ATOM 100 O O '.' UNL A 1 100 '.' 1.0 20.0 0 100 UNL A O 1 7.199999999999999 4.199999999999999 0
ATOM 101 O O '.' UNL A 1 101 '.' 1.0 20.0 0 101 UNL A O 1 8.4 4.199999999999999 0
ATOM 102 O O '.' UNL A 1 102 '.' 1.0 20.0 0 102 UNL A O 1 9.6 4.199999999999999 0
ATOM 103 O O '.' UNL A 1 103 '.' 1.0 20.0 0 103 UNL A O 1 10.2 4.199999999999999 0
ATOM 104 O O '.' UNL A 1 104 '.' 1.0 20.0 0 104 UNL A O 1 11.4 4.199999999999999 0
ATOM 105 O O '.' UNL A 1 105 '.' 1.0 20.0 0 105 UNL A O 1 12.0 4.199999999999999 0
ATOM 106 O O '.' UNL A 1 106 '.' 1.0 20.0 0 106 UNL A O 1 12.9 4.199999999999999 0
ATOM 107 O O '.' UNL A 1 107 '.' 1.0 20.0 0 107 UNL A O 1 13.5 4.199999999999999 0
ATOM 108 O O '.' UNL A 1 108 '.' 1.0 20.0 0 108 UNL A O 1 14.7 4.199999999999999 0
ATOM 109 O O '.' UNL A 1 109 '.' 1.0 20.0 0 109 UNL A O 1 15.899999999999999 4.199999999999999 0
ATOM 110 O O '.' UNL A 1 110 '.' 1.0 20.0 0 110 UNL A O 1 17.4 4.199999999999999 0
ATOM 111 O O '.' UNL A 1 111 '.' 1.0 20.0 0 111 UNL A O 1 18.3 4.199999999999999 0
ATOM 112 O O '.' UNL A 1 112 '.' 1.0 20.0 0 112 UNL A O 1 19.2 4.199999999999999 0
ATOM 113 O O '.' UNL A 1 113 '.' 1.0 20.0 0 113 UNL A O 1 3.5999999999999996 3.9000000000000004 0
ATOM 114 O O '.' UNL A 1 114 '.' 1.0 20.0 0 114 UNL A O 1 3.9 3.9000000000000004 0
ATOM 115 O O '.' UNL A 1 115 '.' 1.0 20.0 0 115 UNL A O 1 4.8 3.9000000000000004 0
ATOM 116 O O '.' UNL A 1 116 '.' 1.0 20.0 0 116 UNL A O 1 6.6 3.9000000000000004 0
ATOM 117 O O '.' UNL A 1 117 '.' 1.0 20.0 0 117 UNL A O 1 7.199999999999999 3.9000000000000004 0
ATOM 118 O O '.' UNL A 1 118 '.' 1.0 20.0 0 118 UNL A O 1 8.4 3.9000000000000004 0
ATOM 119 O O '.' UNL A 1 119 '.' 1.0 20.0 0 119 UNL A O 1 9.6 3.9000000000000004 0
ATOM 120 O O '.' UNL A 1 120 '.' 1.0 20.0 0 120 UNL A O 1 9.9 3.9000000000000004 0
ATOM 121 O O '.' UNL A 1 121 '.' 1.0 20.0 0 121 UNL A O 1 10.2 3.9000000000000004 0
ATOM 122 O O '.' UNL A 1 122 '.' 1.0 20.0 0 122 UNL A O 1 12.0 3.9000000000000004 0
ATOM 123 O O '.' UNL A 1 123 '.' 1.0 20.0 0 123 UNL A O 1 12.9 3.9000000000000004 0
ATOM 124 O O '.' UNL A 1 124 '.' 1.0 20.0 0 124 UNL A O 1 13.5 3.9000000000000004 0
ATOM 125 O O '.' UNL A 1 125 '.' 1.0 20.0 0 125 UNL A O 1 14.7 3.9000000000000004 0
ATOM 126 O O '.' UNL A 1 126 '.' 1.0 20.0 0 126 UNL A O 1 15.899999999999999 3.9000000000000004 0
ATOM 127 O O '.' UNL A 1 127 '.' 1.0 20.0 0 127 UNL A O 1 17.4 3.9000000000000004 0
ATOM 128 O O '.' UNL A 1 128 '.' 1.0 20.0 0 128 UNL A O 1 18.3 3.9000000000000004 0
ATOM 129 O O '.' UNL A 1 129 '.' 1.0 20.0 0 129 UNL A O 1 19.2 3.9000000000000004 0
ATOM 130 O O '.' UNL A 1 130 '.' 1.0 20.0 0 130 UNL A O 1 4.2 3.5999999999999996 0
ATOM 131 O O '.' UNL A 1 131 '.' 1.0 20.0 0 131 UNL A O 1 4.5 3.5999999999999996 0
ATOM 132 O O '.' UNL A 1 132 '.' 1.0 20.0 0 132 UNL A O 1 4.8 3.5999999999999996 0
ATOM 133 O O '.' UNL A 1 133 '.' 1.0 20.0 0 133 UNL A O 1 6.6 3.5999999999999996 0
ATOM 134 O O '.' UNL A 1 134 '.' 1.0 20.0 0 134 UNL A O 1 7.199999999999999 3.5999999999999996 0
ATOM 135 O O '.' UNL A 1 135 '.' 1.0 20.0 0 135 UNL A O 1 8.4 3.5999999999999996 0
ATOM 136 O O '.' UNL A 1 136 '.' 1.0 20.0 0 136 UNL A O 1 9.9 3.5999999999999996 0
ATOM 137 O O '.' UNL A 1 137 '.' 1.0 20.0 0 137 UNL A O 1 11.1 3.5999999999999996 0
ATOM 138 O O '.' UNL A 1 138 '.' 1.0 20.0 0 138 UNL A O 1 11.4 3.5999999999999996 0
ATOM 139 O O '.' UNL A 1 139 '.' 1.0 20.0 0 139 UNL A O 1 11.7 3.5999999999999996 0
ATOM 140 O O '.' UNL A 1 140 '.' 1.0 20.0 0 140 UNL A O 1 12.0 3.5999999999999996 0
ATOM 141 O O '.' UNL A 1 141 '.' 1.0 20.0 0 141 UNL A O 1 12.9 3.5999999999999996 0
ATOM 142 O O '.' UNL A 1 142 '.' 1.0 20.0 0 142 UNL A O 1 13.2 3.5999999999999996 0
ATOM 143 O O '.' UNL A 1 143 '.' 1.0 20.0 0 143 UNL A O 1 13.5 3.5999999999999996 0
ATOM 144 O O '.' UNL A 1 144 '.' 1.0 20.0 0 144 UNL A O 1 14.7 3.5999999999999996 0
ATOM 145 O O '.' UNL A 1 145 '.' 1.0 20.0 0 145 UNL A O 1 15.899999999999999 3.5999999999999996 0
ATOM 146 O O '.' UNL A 1 146 '.' 1.0 20.0 0 146 UNL A O 1 17.4 3.5999999999999996 0
ATOM 147 O O '.' UNL A 1 147 '.' 1.0 20.0 0 147 UNL A O 1 18.3 3.5999999999999996 0
ATOM 148 O O '.' UNL A 1 148 '.' 1.0 20.0 0 148 UNL A O 1 19.2 3.5999999999999996 0
ATOM 149 O O '.' UNL A 1 149 '.' 1.0 20.0 0 149 UNL A O 1 4.5 3.3000000000000007 0
ATOM 150 O O '.' UNL A 1 150 '.' 1.0 20.0 0 150 UNL A O 1 4.8 3.3000000000000007 0
ATOM 151 O O '.' UNL A 1 151 '.' 1.0 20.0 0 151 UNL A O 1 6.6 3.3000000000000007 0
ATOM 152 O O '.' UNL A 1 152 '.' 1.0 20.0 0 152 UNL A O 1 7.199999999999999 3.3000000000000007 0
ATOM 153 O O '.' UNL A 1 153 '.' 1.0 20.0 0 153 UNL A O 1 8.4 3.3000000000000007 0
ATOM 154 O O '.' UNL A 1 154 '.' 1.0 20.0 0 154 UNL A O 1 9.9 3.3000000000000007 0
ATOM 155 O O '.' UNL A 1 155 '.' 1.0 20.0 0 155 UNL A O 1 11.1 3.3000000000000007 0
ATOM 156 O O '.' UNL A 1 156 '.' 1.0 20.0 0 156 UNL A O 1 12.299999999999999 3.3000000000000007 0
ATOM 157 O O '.' UNL A 1 157 '.' 1.0 20.0 0 157 UNL A O 1 12.9 3.3000000000000007 0
ATOM 158 O O '.' UNL A 1 158 '.' 1.0 20.0 0 158 UNL A O 1 13.5 3.3000000000000007 0
ATOM 159 O O '.' UNL A 1 159 '.' 1.0 20.0 0 159 UNL A O 1 14.7 3.3000000000000007 0
ATOM 160 O O '.' UNL A 1 160 '.' 1.0 20.0 0 160 UNL A O 1 15.899999999999999 3.3000000000000007 0
ATOM 161 O O '.' UNL A 1 161 '.' 1.0 20.0 0 161 UNL A O 1 17.4 3.3000000000000007 0
ATOM 162 O O '.' UNL A 1 162 '.' 1.0 20.0 0 162 UNL A O 1 18.3 3.3000000000000007 0
ATOM 163 O O '.' UNL A 1 163 '.' 1.0 20.0 0 163 UNL A O 1 19.2 3.3000000000000007 0
ATOM 164 O O '.' UNL A 1 164 '.' 1.0 20.0 0 164 UNL A O 1 3.3 3.0 0
ATOM 165 O O '.' UNL A 1 165 '.' 1.0 20.0 0 165 UNL A O 1 4.5 3.0 0
ATOM 166 O O '.' UNL A 1 166 '.' 1.0 20.0 0 166 UNL A O 1 5.1 3.0 0
ATOM 167 O O '.' UNL A 1 167 '.' 1.0 20.0 0 167 UNL A O 1 6.3 3.0 0
ATOM 168 O O '.' UNL A 1 168 '.' 1.0 20.0 0 168 UNL A O 1 7.199999999999999 3.0 0
ATOM 169 O O '.' UNL A 1 169 '.' 1.0 20.0 0 169 UNL A O 1 8.4 3.0 0
ATOM 170 O O '.' UNL A 1 170 '.' 1.0 20.0 0 170 UNL A O 1 9.9 3.0 0
ATOM 171 O O '.' UNL A 1 171 '.' 1.0 20.0 0 171 UNL A O 1 11.1 3.0 0
ATOM 172 O O '.' UNL A 1 172 '.' 1.0 20.0 0 172 UNL A O 1 12.299999999999999 3.0 0
ATOM 173 O O '.' UNL A 1 173 '.' 1.0 20.0 0 173 UNL A O 1 12.9 3.0 0
ATOM 174 O O '.' UNL A 1 174 '.' 1.0 20.0 0 174 UNL A O 1 13.799999999999999 3.0 0
ATOM 175 O O '.' UNL A 1 175 '.' 1.0 20.0 0 175 UNL A O 1 14.7 3.0 0
ATOM 176 O O '.' UNL A 1 176 '.' 1.0 20.0 0 176 UNL A O 1 15.899999999999999 3.0 0
ATOM 177 O O '.' UNL A 1 177 '.' 1.0 20.0 0 177 UNL A O 1 3.5999999999999996 2.6999999999999993 0
ATOM 178 O O '.' UNL A 1 178 '.' 1.0 20.0 0 178 UNL A O 1 3.9 2.6999999999999993 0
ATOM 179 O O '.' UNL A 1 179 '.' 1.0 20.0 0 179 UNL A O 1 4.2 2.6999999999999993 0
ATOM 180 O O '.' UNL A 1 180 '.' 1.0 20.0 0 180 UNL A O 1 5.3999999999999995 2.6999999999999993 0
ATOM 181 O O '.' UNL A 1 181 '.' 1.0 20.0 0 181 UNL A O 1 5.7 2.6999999999999993 0
ATOM 182 O O '.' UNL A 1 182 '.' 1.0 20.0 0 182 UNL A O 1 6.0 2.6999999999999993 0
ATOM 183 O O '.' UNL A 1 183 '.' 1.0 20.0 0 183 UNL A O 1 7.5 2.6999999999999993 0
ATOM 184 O O '.' UNL A 1 184 '.' 1.0 20.0 0 184 UNL A O 1 7.8 2.6999999999999993 0
ATOM 185 O O '.' UNL A 1 185 '.' 1.0 20.0 0 185 UNL A O 1 8.1 2.6999999999999993 0
ATOM 186 O O '.' UNL A 1 186 '.' 1.0 20.0 0 186 UNL A O 1 9.9 2.6999999999999993 0
ATOM 187 O O '.' UNL A 1 187 '.' 1.0 20.0 0 187 UNL A O 1 10.799999999999999 2.6999999999999993 0
ATOM 188 O O '.' UNL A 1 188 '.' 1.0 20.0 0 188 UNL A O 1 12.299999999999999 2.6999999999999993 0
ATOM 189 O O '.' UNL A 1 189 '.' 1.0 20.0 0 189 UNL A O 1 12.9 2.6999999999999993 0
ATOM 190 O O '.' UNL A 1 190 '.' 1.0 20.0 0 190 UNL A O 1 14.1 2.6999999999999993 0
ATOM 191 O O '.' UNL A 1 191 '.' 1.0 20.0 0 191 UNL A O 1 15.0 2.6999999999999993 0
ATOM 192 O O '.' UNL A 1 192 '.' 1.0 20.0 0 192 UNL A O 1 15.299999999999999 2.6999999999999993 0
ATOM 193 O O '.' UNL A 1 193 '.' 1.0 20.0 0 193 UNL A O 1 15.6 2.6999999999999993 0
ATOM 194 O O '.' UNL A 1 194 '.' 1.0 20.0 0 194 UNL A O 1 17.4 2.6999999999999993 0
ATOM 195 O O '.' UNL A 1 195 '.' 1.0 20.0 0 195 UNL A O 1 18.3 2.6999999999999993 0
ATOM 196 O O '.' UNL A 1 196 '.' 1.0 20.0 0 196 UNL A O 1 19.2 2.6999999999999993 0