本記事では、HELM-GPTを実際に動かして,論文の内容を追試した内容を報告します。 特に、論文で提案されているHELM-GPTの学習手法を再現するために、公開されているGitHub上のコードを基に環境構築から実行まで一通り試しました。なお、学習済みのモデルは公開されていなかったため、全てのモデルを学習させました。また、単なる論文の再現に留まらず、独自の視点から追加の評価を行い、学習プロセスごとの効果や生成された分子の特性についても検証しました。これにより、HELM-GPTがどのように膜透過性やKRAS結合親和性に優れた環状ペプチドを生成できるのか、その性能をさらに詳細に分析していきます。
また、今回学習したモデルは以下のリンクで公開していますので、興味のある方はご参照ください。HELM-GPT学習済みモデル
はじめに:HELM-GPTとは
HELM-GPTは、環状ペプチドのde novoデザインを目的とした生成モデルです。本記事の基になった論文は「HELM-GPT: de novo macrocyclic peptide design using generative pre-trained transformer 」であり、そのコードはGitHubで公開されています。従来の創薬アプローチでは、小分子化合物の設計には成功していたものの、非天然アミノ酸を含む環状ペプチドの設計には限界がありました。これらの課題を克服するため、HELM-GPTは環状分子の階層的な編集言語であるHELM(Hierarchical Editing Language for Macromolecules)表記を使用し、生成型の事前学習済みトランスフォーマー(GPT)と組み合わせることで、環状ペプチドのデザインを行います。
HELM-GPTはまずChEMBLなどのデータセットからHELM表記のペプチドを用いて事前学習を行い、その後、膜透過性やKRAS結合親和性といった特定の特性を持つ分子を生成するために強化学習を行います。さらに、強化学習の性能向上のため、新たな学習手法である「Contrastive Preference Learning(CPL)」を導入し、ターゲット特性の最適化を効率化しました。このようにしてHELM-GPTは、膜透過性と標的タンパク質(ここではKRAS)への結合親和性の両特性が優れた「欲張りな」環状ペプチドの生成を可能としています。
環境構築と準備
Pythonバージョンと主要なライブラリ
Python: 3.7.12
Pandas: 1.3.5
Numpy: 1.21.6
Scipy: 1.7.3
Matplotlib: 3.5.3
Scikit-learn: 0.23.2
Torch: 1.13.1 (CUDA 11.7)
Torchaudio 0.13.1 (CUDA 11.7)
Torchvision 0.14.1 (CUDA 11.7)
RDKit: 2022.09.1
JupyterLab: 3.6.7
Biopython: 1.77
具体的な仮想環境の作成手順はオリジナルのGitHubに記載されているのでそちらを参照してください。
データセットの確認・準備
以下に論文中で使用されるデータセットを紹介します。
a. ChEMBLデータセット
- モデルの事前学習に用いられます。22,046のペプチドと2,851のモノマーから構成されています。
- 学習の際、HELM表記の長さが200を超えるものや、モノマーライブラリに含まれないものを除外し、最終的に20,783のHELMシーケンスを使用しています。
b. CycPeptMPDBデータセット
- ファインチューニングおよび強化学習に使用されたデータセットで、HELM表記で格納された7,451の環状ペプチドと312種類のモノマーが含まれています。
- 環状ペプチドの膜透過性最適化に利用されます。
c. KRAS Kdデータセット
- KRASに対する結合親和性(Kd値)の予測および最適化に使用されたデータセットで、2,757のペプチドから構成されています。
- 元は特許から取得したデータで、HELM表記に変換されています。KRASへの結合親和性の向上に焦点を当てたモデルのトレーニングに利用されます。
上記の3種のデータセットをまとめたものとChEMBLデータセットとCycPeptMPDBデータセットのそれぞれが著者らのGitHub上で個別に公開されていましたが、KRAS Kdデータセットは単独では公開されていませんでした。しかし、以下の実験を行うにあたって、KRAS KdデータセットとCycPeptMPDBデータセットを合わせたものが必要であったため、上記の公開されたデータセットを組み合わせて疑似的に作成しました。上記のデータセットに加え、この疑似的に作成したKRAS Kdデータセット+CycPeptMPDBデータセットを用いて以下の実験を行います。
HELM-GPTの学習 + 欲張り環状ペプチドの生成
本節では、HELM-GPTを学習させ、分子を生成させるまでの一連の流れを紹介します。基本的に、著者らのGitHub上で公開されているコードを使います。そのため、まず著者らのGitHubリポジトリをローカル環境にクローンし、実行環境をセットアップしてください。なお、本記事の内容に基づいて学習した全てのモデルを公開しておきます。これらのファイルをhelm-gpt/trained_models/においておくと、スムーズに動作します。
事前学習+ファインチューニング
HELM表記の基本的な文法を学習するため、まずChEMBLデータセット(helm-gpt/data/prior/chembl32/biotherapeutics_dict_prot_flt.csv)を用いて事前学習を行いました(Prior$ChEMBL$)。なお、この学習の際に、オリジナルのGitHubで公開されているコードを一部改変した、train_prior_dataset_split.pyを用います。このコードはこちらからダウンロードできます。
cd CLONE_PATH/helm-gpt/ # クローンした場所(CLONE_PATH)を指定
python train_prior_dataset_split.py --data data/prior/chembl32/biotherapeutics_dict_prot_flt.csv --output_dir result/prior/chembl_5.0 --n_epochs 200 --max_len 200 --batch_size 512
上記の結果得られた事前学習モデルはtrained_models/Prior_ChEMBL.ptとして保存しています。
続いて、先ほどの事前学習モデル(Prior$ChEMBL$)を用いて、CycPeptMPDBデータセット(data/prior/CycPeptMPDB/CycPeptMPDB_Peptide_All.csv)、疑似的に作成したCycPeptMPDB + KRAS Kdデータセット(data/CycPeptMPDB_KRAS_Kd_Dataset_Subset.csv)でそれぞれファインチューニングを行います。
# Prior_CycPeptMPDB の学習
mkdir result/prior/CycPeptMPDB # 学習結果を格納するディレクトリの作成
python train_prior_dataset_split.py --data data/prior/CycPeptMPDB/CycPeptMPDB_Peptide_All.csv --model_path trained_model/Prior_ChEMBL.pt --output_dir result/prior/CycPeptMPDB --n_epochs 50 --max_len 200 --batch_size 512
# Prior_KRAS+CycPeptMPDB の学習
mkdir result/prior/KRAS_CycPeptMPDB # 学習結果を格納するディレクトリの作成
python train_prior_dataset_split.py --data data/CycPeptMPDB_KRAS_Kd_Dataset_Subset.csv --model_path trained_model/PriorChEMBL.pt --output_dir result/prior/KRAS_CycPeptMPDB --n_epochs 50 --max_len 200 --batch_size 512
上記の結果得られた事前学習モデルは、それぞれの学習結果を格納するディレクトリに保存される。事前に学習した学習済みモデルはtrained_models/Prior_CycPeptMPDB.pt、trained_models/Prior_KRAS_CycPeptMPDB.ptに含まれています。
これらの事前学習モデルを用いて1,000個の分子を生成させ、
# Prior_ChEMBLによる分子の生成
python generate.py --model_path trained_model/Prior_ChEMBL.pt --out_file trained_model/generated_molecules/Prior_ChEMBL_1k_samples.csv --n_samples 1000 --max_len 200 --batch_size 128
# Prior_CycPeptMPDBによる分子の生成
python generate.py --model_path trained_model/Prior_CycPeptMPDB.pt --out_file trained_model/generated_molecules/Prior_CycPeptMPDB_1k_samples.csv --n_samples 1000 --max_len 200 --batch_size 128
# Prior_KRAS_CycPeptMPDBによる分子の生成
python generate.py --model_path trained_model/Prior_KRAS_CycPeptMPDB.pt --out_file trained_model/generated_molecules/Prior_KRAS_CycPeptMPDB_1k_samples.csv --n_samples 1000 --max_len 200 --batch_size 128
上記のコードを実行し生成された分子は trained_models/generated_molucules/にそれぞれ保存しています。これらの生成した分子を基に、モデルの性能評価を行います。ただし、Baseline$ChEMBL$については、ChEMBLデータセットからランダムに選んだ1,000個の分子で評価しました。以下の表では、論文で報告されている文献値と、今回独自に行った実験結果の評価値を比較しています。

*:SAscore = 0 の分子を除いて平均を取った
なお、上記の項目についての詳細は以下の通りです。
- Validity: 1,000個のHELM配列から生成された分子のうち、HELM表記として文法的に有効な分子の割合を測定します。
- Uniqueness: 有効な分子の中で、ユニークな分子の割合を測定します。
- Diversity: 生成された分子の任意のペア間のタニモト類似度(Tanimoto similarity)の平均値を用い、それを1から差し引いて多様性を評価します。
- SNN (Similarity to Nearest Neighbor): 生成された分子のうち、トレーニングセットに最も近い分子との類似度を測定します。
- Novelty: ユニークに生成された分子の中で、トレーニングセットに存在しない新規な分子の割合を測定します。
- SAscore (Synthetic Accessibility score): 生成された分子の合成容易性を評価します。
今後の学習で用いるPrior$KRAS+CycPeptMPDB$について、ValidityとUniquenessの評価値が文献値ではいずれも1.000であるのに対し、我々が学習させたモデルではどちらも0.9を下回る結果となりました。この差異は、学習データを疑似的に作成したことに起因している可能性があります。しかし、論文と同じ学習データを用いたPrior$CycPeptMPDB$においても、ValidityとUniquenessが文献値より低いという結果が得られたことを踏まえると、学習データ以外の要因、例えば学習パラメータの調整方法などが影響していると考えられます。
ただし、Prior$ChEMBL$からPrior$KRAS+CycPeptMPDB$へのファインチューニングにおいて、Uniquenessを除く他の評価指標の変化(Validityの増加など)は、文献値と今回得られたモデルの双方で同様の傾向を示しました。この結果を踏まえ、我々が学習させたPrior$KRAS+CycPeptMPDB$を、この次のステップである強化学習に用いることにしました。
強化学習
以降では、我々が学習させたモデルであるPrior$KRAS+CycPeptMPDB$を基に、強化学習を行うことで膜透過性とKRASへの結合親和性を最適化しました。学習の流れを以下に示します。
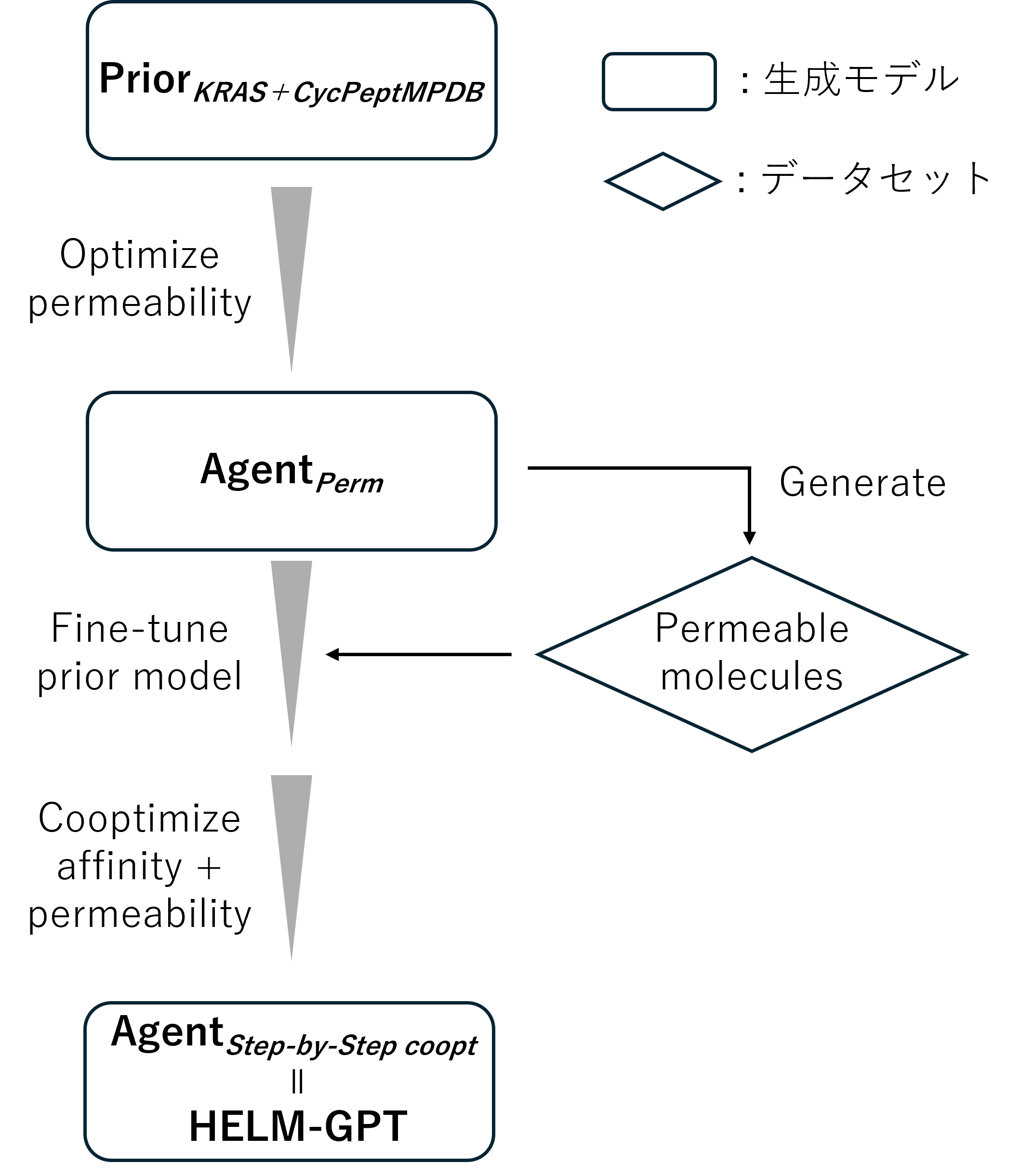
まず、Prior$KRAS+CycPeptMPDB$を基に膜透過性に関して強化学習を行います(Agent$Perm$)。
# Agent_permの強化学習
python train_agent.py --prior result/prior/trained_model/PriorKRAS_CycPeptMPDB.pt --output_dir result/agent/cpp/ --batch_size 32 --n_steps 500 --sigma 60 --task permeability --max_len 140
得られた学習モデルはresult/agent/cpp/内に保存され、予め学習したものはtrained_models/Agent_perm.ptに保存しています。
次に、論文中での操作と同様に、Agent$Perm$で1,000個の分子を生成し、そのデータセットを用いてAgent$Perm$をファインチューニングします。
# Agent_permによる分子の生成
python generate.py --model_path trained_model/Agent_perm.pt --out_file trained_model/generated_molecules/Agent_perm_1k_samples.csv --n_samples 1000 --max_len 200 --batch_size 128
# Agent_permが生成した分子によりAgent_permをfine-tuning
mkdir result/agent/Prior_after_1st_RL # 学習結果を格納するディレクトリの作成
python train_prior_dataset_split.py --data trained_model/generated_molecules/Agent_perm_1k_samples.csv --model_path trained_model/Agent_perm.pt --output_dir result/agent/finetuned_Agent_perm --n_epochs 200 --max_len 200 --batch_size 512
このようにしてファインチューニングしたモデル(trained_models/finetuned_Agent_perm.pt)について、膜透過性と結合親和性の両方に関して強化学習を行います。
# 膜透過性と結合親和性について共最適化
mkdir result/agent/co_affinity_optimization # 学習結果を格納するディレクトリの作成
python train_agent.py --prior trained_model/finetuned_Agent_perm.pt --output_dir result/agent/co_affinity_optimization --batch_size 32 --n_steps 500 --sigma 60 --task all --max_len 140
このようにして得られたモデルをHELM-GPT(=Agent$Step\ by\ step\ coopt$)としました。このHELM-GPTはtrained_models/HELM_GPT.ptとして保存しています。
HELM-GPTに環状ペプチドを作らせてみる
今回学習させたHELM-GPTを用いて30万個の分子を生成しました。生成した分子の膜透過能とKRASへの解離定数の予測値について、それぞれプロットした結果を以下に示します。この結果から、生成された分子の多くが、これら両方においてある程度優れていおり、同様の性能であることが分かります。
また、今回生成した分子の内、膜透過能と結合親和性の両方に優れた環状ペプチドの例を以下に示します。これらの環状ペプチドは非天然アミノ酸であるNメチルアミノ酸やD体のアミノ酸が含まれており、HELM-GPTでは非天然アミノ酸を含有する環状ペプチドであっても生成できることが分かります。

学習効果の検証
ここでは、論文中では行っていませんが、HELM-GPTのファインチューニング(強化学習により生成された分子による自己学習)に効果があるかを調査した結果を示します。具体的には、下図右側に示した本来のHELM-GPTに対して、下図左側に示すようにAgent$Perm$をファインチューニングせずに直接、膜透過性と結合親和性に関して強化学習のみを行ったモデル(Agent$direct\ coopt$)と比較しました。

学習の効果を検証するために、各モデルに30万個の分子を生成させ、その分子の性能を評価しました。

事前学習モデルであるPrior$KRAS+CycPeptMPDB$は、膜透過性と結合親和性の両方において幅広い分子を生成しています。このPrior$KRAS+CycPeptMPDB$を膜透過性に対して強化学習したAgent$Perm$では、膜透過性が向上していることが確認できます。一方で、結合親和性に関してはやや悪化(Kdが高くなる)しています。さらに、膜透過性と結合親和性の両方を最適化したAgent$direct\ coopt$およびAgent$Step\ by\ step\ coopt$では、膜透過性の向上に加えて結合親和性も改善されていることがわかります。ただし、Agent$direct\ coopt$とAgent$Step\ by\ step\ coopt$の間にはほとんど差が見られず、Agent$Perm$をベースにファインチューニングすることの効果には疑問が残る結果となりました。
これらのモデルから生成された分子のうち、Permeability > -5 かつ KRAS Kd < 20 nMとなる、膜透過能と結合親和性に優れた分子の数を出しました(下図)

膜透過性と親和性の上記の閾値によるフィルターを通過した分子数を比較すると、事前学習モデルであるPrior$KRAS+CycPeptMPDB$はフィルターを通過する分子が非常に少ないことがわかります。一方で、膜透過性に対して強化学習を行ったAgent$Perm$では、フィルターを通過する分子数がPrior$KRAS+CycPeptMPDB$に比べて大幅に増加しています。これは、膜透過性に関する特性が向上した結果であると考えられます。
さらに、Agent$Perm$を基に、膜透過性と結合親和性の両方で最適化したAgent$direct\ coopt$およびAgent$Step\ by\ step\ coopt$では、フィルターを通過する分子数がさらに増加しています。ただし、Agent$direct\ coopt$とAgent$Step\ by\ step\ coopt$ではフィルターを通過する分子数にはわずかな違いしかなく、どちらの手法もほぼ同等の性能を持つことが示唆されます。このため、膜透過性と結合親和性の同時最適化においては、手法間で大きな差は見られないと考えられます。
まとめ
本記事では、HELM-GPTを用いて環状ペプチドの生成と特性最適化を行う流れを紹介しました。従来のde novoデザインの課題を克服するため、事前学習と強化学習の組み合わせにより、膜透過性とKRAS結合親和性の両特性に優れた環状ペプチドを生成することが可能であることを示しました。
特に、膜透過性と結合親和性を効率的に最適化するために行った各ステップを検証し、Agent$Perm$やAgent$direct\ coopt$、Agent$Step\ by\ step\ coopt$といった強化学習モデルの性能を評価しました。その結果、膜透過性の最適化を行ったAgent$Perm$で大幅な膜透過性の向上が見られましたが、結合親和性はわずかに悪化していました。一方で、両特性を同時に最適化したAgent$direct\ coopt$およびAgent$Step\ by\ step\ coopt$では、膜透過性と結合親和性の両方が向上し、フィルターを通過する分子数も増加していることが確認されました。ただし、論文中で行っているファインチューニング手法による大きな性能差は見られず、さらなる改良の余地があると考えられます。
また、生成された分子の特性評価において、膜透過性と結合親和性の値はあくまで予測モデルに基づいたものであり、実験的な検証が必要であることが明らかです。特に、膜透過性の向上を目的とした結果として生成された分子は、いかにも高い疎水性を示す傾向がありました。高い疎水性は膜透過性の向上に寄与する一方で、分子の合成難易度が上昇するという課題もあります。さらに、疎水性が高い分子は溶解性に問題が生じる可能性もあり、実用面での最適化には慎重な検討が必要です。
このような問題を克服するため、膜透過性の最適化において疎水性を単に高めるだけでなく、カメレオン性(細胞膜の内外でコンフォメーションを変化させ、表面極性を制御する能力)に着目した分子の生成を目指すモデルの開発が有効であると考えられます。カメレオン性を備えた分子は細胞膜の透過性を維持しつつ、細胞内での機能性を高めることが期待されます。今後の研究では、カメレオン性を持つ分子のデザインに対応したHELM-GPTの拡張や強化学習手法の開発が重要な課題となるでしょう。
(この記事は研究室インターンで取り組みました:https://kojima-r.github.io/kojima/)
