0.Prologue
数か月前のことになるが、ONNX runtimeに Elixirを被せた OnnxInterpを書き上げ、そのサンプル・プログラムとして YOLOv7による物体検出デモをサクッと用意した。そこまでは良かったのだが…
ちょっとした好奇心から YOLOv7の原論文[*1]を読み始めたところ
... MCUNet and NanoDet focused on producing low-power single-chip and improving the inference speed on edge CPU.
という一文が目に留まり、「NanoDetって何だ?」「エッジCPU向けで超軽量・高速推論だと?」。これは是非とも OnnxInterpのサンプル・プログラムに加えねばと、試行錯誤の移植の旅が始まったのであった![]()
本稿では泥臭い話はカットして、Livebook + OnnxInterpでちゃっちゃと"NanoDet plus"を動作させる手順を紹介したいと思う。
尚、ごちゃごちゃした御託は飛ばして、さっさと NanoDet plusを試したい方は、次のRequirementを確認して4章に進もう![]()
1.Requirement
2022/8/1現在、NanoDet plusのサンプル・プログラムは下記の環境での動作を確認している。
- Windows WSL2/Ubuntu 20.04.2
- Elixir 1.13.4 (compiled with Erlang/OTP 25)
- Erlang/OTP 25 [erts-13.0.2] [source] [64-bit] [smp:8:8] [ds:8:8:10] [async-threads:1] [jit:ns]
- Livebook 0.6.3 / Phoenix 1.6.6
- ONNX Runtime 0.11.1
他にビルドならびに onnxモデルのダウンロードの際に下記のツールを使用している。
- CMake version 3.18 or later
- wget
- 残念ながら Windows, OSXでは -
Windowsでは:
OnnxInterpのコンパイル・ツールチェイン Visual C++ 2019 & MSBuildが吐き出す日本語メッセージ/ワーニング(CP932,ShiftJIS)と Livebook Desktop(Unicode)との相性が悪く、今のところ動作させることが出来ない。尤も、iexや Phoenixアプリとの組み合わせは支障なく動作する筈だ。
== 改定 2022.8.10 ==
下記の記事に書いた回避策を施すことで、Windows版 Livebook Desktopでも本稿の内容を実行できることを確認しました。お試しあれ![]()
===============
OSXでは:
OSXの環境を所有していないので、動作するかどうか分からない![]()
拙作OnnxInterpに関して言えば、本家 ONNX Runtimeの GitHubに OSX用のコンパイル済みライブラリが用意されているので、たぶんCMakeLists.txtをちょこちょこっと修正すれば対応できると思う。
……助っ人募集中
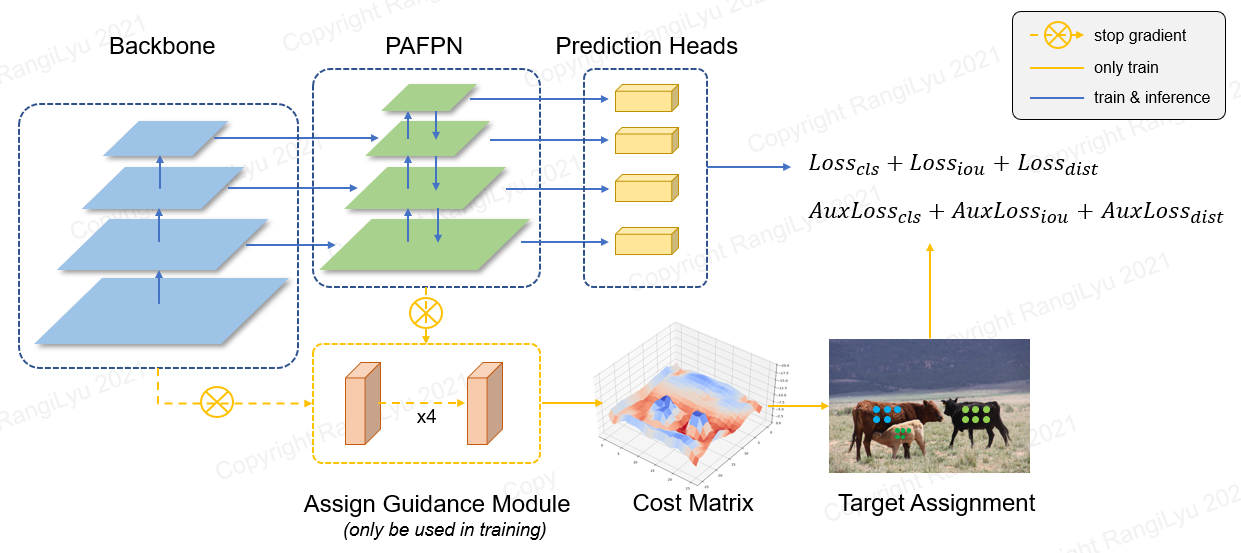
2.What is "NanoDet plus"?
RangiLyu 炼丹人さんが開発・メンテンナスを行っている物体検出DNNモデルで、
NanoDet plus:
Super fast and high accuracy lightweight anchor-free object detection model. Real-time on mobile devices.
だそうである。
最近流行りの FCOS-styleのアーキテクチャを採用しており、オブジェクト検出のBackboneに"ShuffleNetV2"を、マルチスケール特徴集約のFPNに"GhostPAN"を、物体位置等の計算Headsに"Generalized Focal Loss"を配しているようだ。
小生、中国語は読めないので詳しいことは解らないが、DeepLを頼りにRangiLyuさん文献の文字を追ってみると、物体検出に関するHotな技術ノウハウが書かれている様に思えた。参考文献を載せておくので各自精読のこと![]()
さて、技術的な興味は尽きないが、本稿のゴールはそこではない。移植作業に戻ろう。一般に DNNプログラムを移植するに当たって必須の設計情報は、
#「DNNモデルの inputs/outputsの形式仕様と意味仕様」
である。
形式仕様、すなわち inputs/outputsの tensorの形は、Netronを利用すれば簡単に判る。もちろん、拙作 OnnxInterpに付属の info/1関数でも調べることが出来る。
NanoDet plus:
inputs[0]: name: data, type: float32[1,3,416,416]
output[0]: name: output, type: float32[1,3598,112]
一方、意味仕様、すなわち tensorの中身のデータが何を意味しているのかは、残念ながら大概の場合はドキュメントとして公開されていない。本家のプログラムをリバース・エンジニアリングして調べることになろう。
極めて泥臭く地味な作業である……さらっとスキップして結論だけを確認しよう![]()
NanoDet plus:
inputs[0]:
name: data, type: float32[1,3,416,416]
- BGRカラー,NCWHレイアウト,416画素☓416画素の画像で、各カラーチャネルを
(μ,σ): B(103.53,57.375),G(116.28,57.12),R(123.675,58.395)
で正規化してfloat32化したもの
outputs[0]:
name: output, type: float32[1,3598,112]
- 416☓416の特徴マップを{8,16,32,64}の間隔で格子に分割し、各格子点(3598箇所)で対象物の推定値と
対象物を囲む bounding box(以下BBOX)の推定値を一つにまとめたレコード(112要素)が 3598個並んだテーブル
レコードの内訳
[ 0~ 79] - Cocoデータセットの80種の対象物それぞれに対する推定値(評価値)
[ 80~ 87] - 格子点からBBOX左端までの距離を、8か所の仮BBOX端における確からしさで表した分布表
[ 88~ 95] - BBOX上端までの距離(同上)
[ 96~103] - BBOX右端までの距離(同上)
[104~111] - BBOX下端までの距離(同上)
inputs[0]に関しては、画像認識系DNNではよく見かける入力仕様だ。本家プログラムでは、真面目にチャネル毎に(μ,σ)で正規化を行っているが、簡単に全チャネルまとめて{-2.2~2.7}の範囲に画素値変換しても支障はないだろう。
一方 output[0]に関しては、前半のマルチスケールな格子分割や、各格子点で対象物に対する推定値を80種類分返すところは Yolo等でもおなじみの内容なのだが、BBOXの大きさを確からしさ分布表(注:こういう呼び名で果たして良いのやら![]() …正しくはFocal Lossなのかなぁ)で返す設計には初めてお目にかかった。この部分を少し詳しく調べてみよう。
…正しくはFocal Lossなのかなぁ)で返す設計には初めてお目にかかった。この部分を少し詳しく調べてみよう。
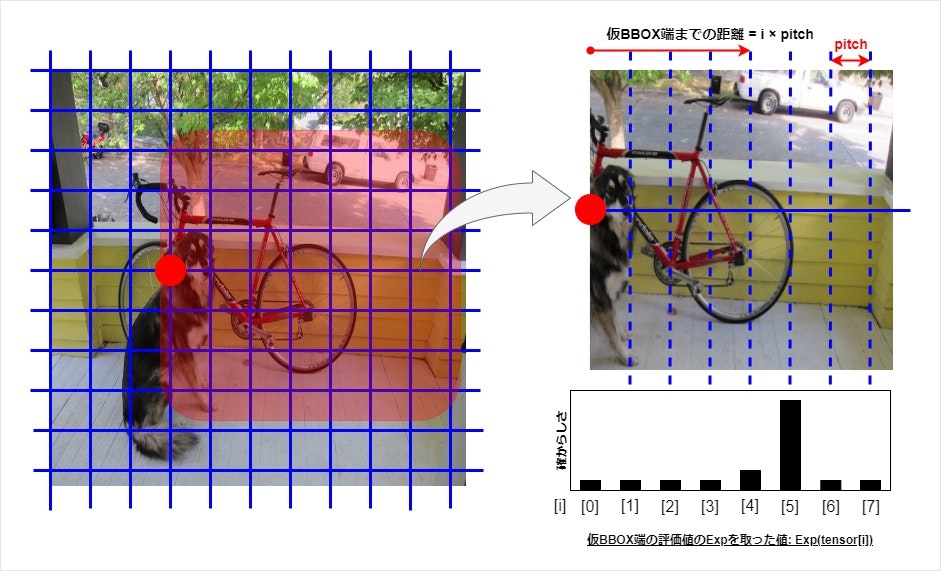
下図は、赤丸を付けた格子点における BBOX右端の確からしさ分布表dist を抜き出したものだ。NanoDet plusでは、分布表dist の要素数は8個固定で、それぞれの値は格子点から等間隔(pitch)に離れた仮BBOX端(格子点を含む)が対象物のBBOX右端である確からしさを表している。実際には、モデルは確からしさとして正負交じりの値を返してくるので、それらに softmaxを掛けて確率分布と同様の扱いが出来る様にしている。真のBBOX右端(自転車の車輪)に近接する仮BBOX端では確からしさが1.0に近づき、それ以外ではほぼ0.0になる。
なるほど、ここまで解れば確からしさ分布表distをBBOX端までの距離wing にデコードする方法は明白だ。分布表dist の平均(重心)を求めれば良いのだ。
格子点からBBOX端までの距離 wing = pitch*\frac{\sum_{i}(i*e^{dist[i]})}{\sum_{i} e^{dist[i]}}
さて、必要な情報は揃った。先に進もう![]()
3.Implement...You know Livebook, don't you?
NanoDet plusのElixirへの移植作業を始めよう。必要な作業は概ね次の一覧となる。
- サンプル・プログラム用のElixirプロジェクトを作成する
- 本家 NanoDet plusのトレーニング済み Pytorchモデルを変換して ONNXモデルを得る
- モデルの inputs仕様に基づいてプリプロセスを用意する
- モデルの outputs仕様に基づいてポストプロセスを用意する
- 人が見て分かるように、推論結果を表示するルーチンまたはアプリを用意する
作業2は、本家に export_onnx.pyなる Pythonスクリプトが用意されているので、Google Colaboratory辺りで実行すれば簡単に片付く。本稿では、学習済みモデル NanoDet-Plus-m-416を借用した。
作業3は、inputs仕様を満たす画像処理で、拙作CImgを使えば2行で事足りる。
作業4は、DNNモデルのoutputsから対象物の種類推定値scoresと、対象物のBBOX推定値boxesを取り出して、Non maximum suppressionに掛けてお仕舞だ。物体検出系ではよくあるパターンだ。まぁ、今回はBBOXデコードの件があるので、後ほどコードを見てみよう。
作業5は、本質的な部分ではないのだが……一番めんどくさい。UIにPhoenixを使おうが、素のPlugを使おうが、高々画像を一枚表示するだけであっても、あれこれとコードを書かねばならない。
ところで、Livebookはご存じですよね?
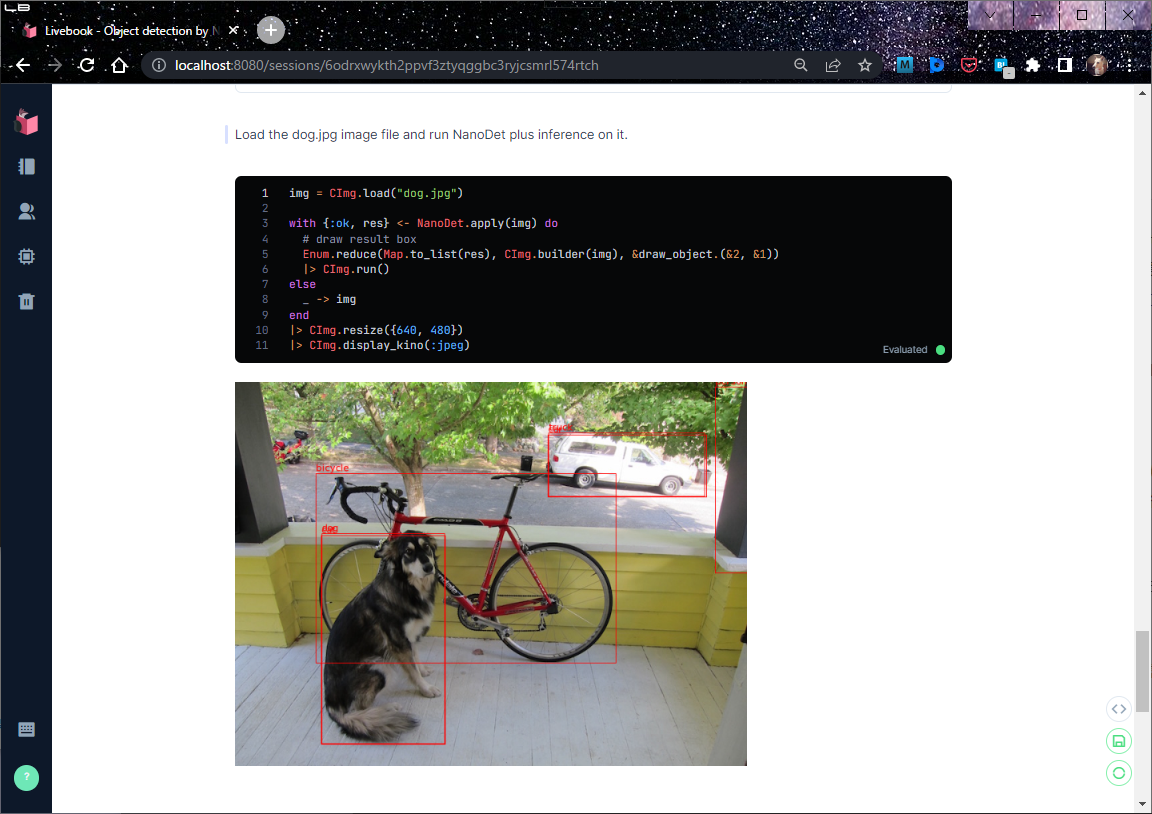
そう、Livebookと Kinoモジュールの助けを借りれば、ブラウザ上に NanoDet plusの推論結果を表示するなんてことはいとも簡単にできる。と言う訳で、NanoDet plusのサンプル・プログラムも Livebookで書きあげている。notebookの置き場所は4章を参照のこと![]()
さて話を戻して、モジュール NanoDetのコードを少し見てみよう![]()
下記のコードはNanoDet推論のメイン関数だ。
プリプロセスから DNNモデルによる推論までは、いつもと変わり映えしないコード。
ポストプロセスでは、最初にDNNモデルのoutputsに格子点座標とピッチのリストを付け加え(PostDNN.mesh_grid/3)、次に対象物の種類推定値が0.25以上になるレコードだけを残すように篩いかけをしている(PostDNN.sieve/2)。早い段階での篩いかけは、これが初めての試みだ。重~いexp()を含むBBOXデコードの無駄に行いたくない思う貧乏性である![]()
そのあと、篩に残ったレコードに2章で調べたBBOXデコードを掛け(decode_boxes/2)、最後にNMSを通す。
以上![]()
defmodule NanoDet do
use OnnxInterp, model: "./NanoDet-Plus-m-416.onnx", label: "./coco.label"
@nanodet_shape {416, 416}
def apply(img) do
# preprocess
bin = img
|> CImg.resize(@nanodet_shape)
|> CImg.to_binary([{:range, {-2.2, 2.7}}, :nchw, :bgr])
# prediction
outputs = __MODULE__
|> OnnxInterp.set_input_tensor(0, bin)
|> OnnxInterp.invoke()
|> OnnxInterp.get_output_tensor(0)
|> Nx.from_binary({:f, 32}) |> Nx.reshape({:auto, 112})
# postprocess
{scores, boxes} =
Nx.concatenate([outputs, PostDNN.mesh_grid(@nanodet_shape, [8, 16, 32, 64])], axis: 1)
|> PostDNN.sieve(fn tensor ->
Nx.slice_along_axis(tensor, 0, 80, axis: 1)
|> Nx.reduce_max(axes: [1])
|> Nx.greater_equal(0.25)
end)
|> (&{Nx.slice_along_axis(&1, 0, 80, axis: 1), Nx.slice_along_axis(&1, 80, 35, axis: 1)}).()
{width, height, _, _} = CImg.shape(img)
boxes = decode_boxes(boxes, {width, height})
OnnxInterp.non_max_suppression_multi_class(__MODULE__,
Nx.shape(scores), Nx.to_binary(boxes), Nx.to_binary(scores), boxrepr: :corner
)
end
:
BBOXデコードdecode_box/2は下の様に実装した。基本的に2章で説明した通りだ。
心臓部の「格子点からBBOX端までの距離」を求める関数wing/1は無名関数として実装している。
また、元画像のサイズに合うようにBBOXを拡大・縮小するしたり(scale/1)、BBOXが元画像からはみ出さないように制限したり(keep_within/2)するために、引数worldで元画像の幅と高さを貰っている。
def decode_boxes(tensor, world \\ {}) do
max_index = Nx.axis_size(tensor, 0) - 1
for(i <- 0..max_index, do: decode_box(tensor[i], world))
|> Nx.stack()
end
def decode_box(tensor, world \\ {}) do
grid_x = Nx.to_number(tensor[-3])
grid_y = Nx.to_number(tensor[-2])
arm = Nx.iota({8}) |> Nx.multiply(tensor[-1]) # [0, pitch, 2*pitch, ... 7*pitch]
# private func: decode probability table to wing.
wing = fn t ->
max = Nx.reduce_max(t)
{weight, sum} =
Nx.subtract(t, max) # prevent exp from becoming too big
|> Nx.exp()
|> (&{&1, Nx.sum(&1)}).()
# mean of probability list
Nx.dot(weight, arm) |> Nx.divide(sum) |> Nx.to_number()
end
{scale_w, scale_h} = scale(world)
[
scale_w * (grid_x - wing.(tensor[0..7])),
scale_h * (grid_y - wing.(tensor[8..15])),
scale_w * (grid_x + wing.(tensor[16..23])),
scale_h * (grid_y + wing.(tensor[24..31]))
]
|> keep_within(world) # keep coners of the box within the original photo.
|> Nx.stack()
end
4.Let's play!
3章で触れた通り、NanoDet plusのサンプル・プログラムは Livebookに書いており、下記で公開している。
GitHub: NanoDet_plus.livemd
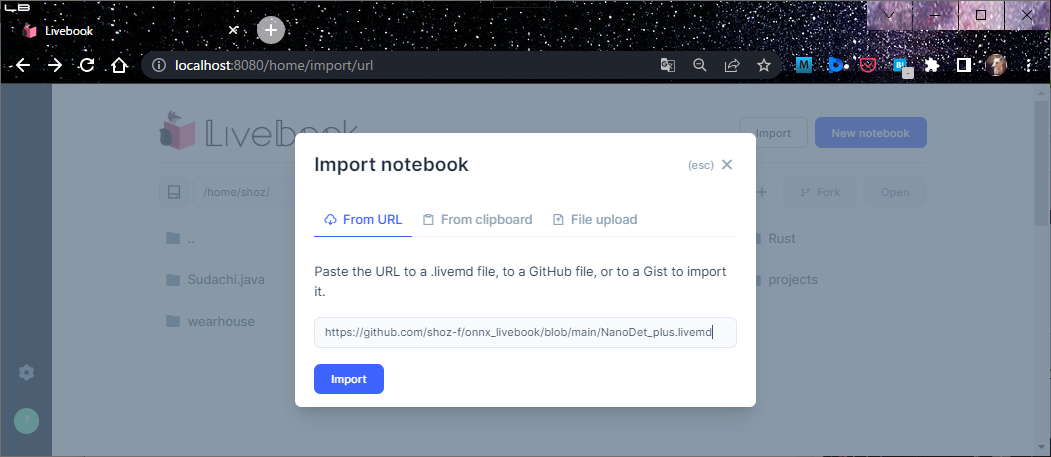
上記サイトから (A)notebookを適当なディレクトリにダウンロードして、Livebookのファイラ(?)でOpenするか、または(B)Livebookに直接Importすれば、すぐに試すことができる![]()
notobookの先頭部分のセルには、NanoDet plusサンプル・プログラムの実行に必要なモジュール群のインストールと、ONNXモデル・ファイルやテスト用の画像のダウンロード(Livebookのホームに格納)を記述している。手ぶらでどうぞ![]()
ご存じの通り、Livebookでは一番最後のコード・セルの実行ボタンを押せば、そこまでの全てのコード・セルが自動的に実行されるようになっている。まさしくボタン一発で NanoDet plusを動かしてみることができるのだ![]()
……簡単過ぎて他に書くことがない![]()
5.Epilog
終わったぁ。ふむ、あいかわらず中身のない記事だなぁ orz
とにかく DNN推論なら案外とお手軽に試してみることができるよってことが伝われば十分である。
本稿を書き始めた時点では、泥臭~い作業にフォーカスした【B面】も書こうかと考えていたが…要らないよな。
参考文献
- RangiLyu 炼丹人:
GitHub:"NanoDet-Plus"
"超简单辅助模块加速训练收敛,精度大幅提升!移动端实时的NanoDet升级版NanoDet-Plus来了!" - Xiang Li, Wenhai Wang, Lijun Wu, Shuo Chen, Xiaolin Hu, Jun Li, Jinhui Tang, and Jian Yang:
"Generalized Focal Loss: Learning Qualified and Distributed Bounding Boxes for Dense Object Detection"