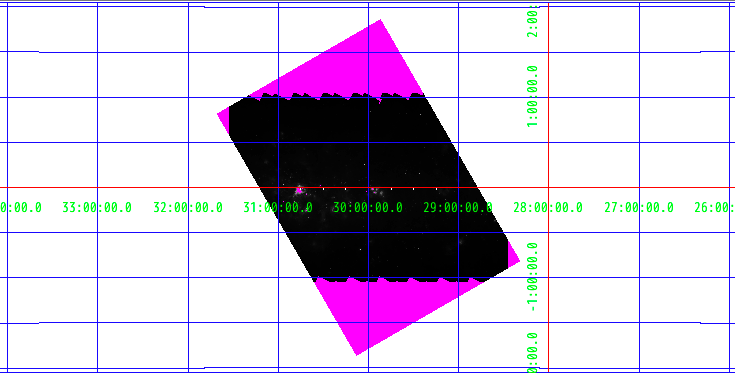
この記事では、fits の回転方法について記述します。使用するのは、 casa もしくは miriad です。
例によって、
https://irsa.ipac.caltech.edu/data/SPITZER/MIPSGAL/images/mosaics24/
から
MG0290n005_024.fits
MG0290p005_024.fits
MG0300n005_024.fits
MG0300p005_024.fits
MG0310n005_024.fits
MG0310p005_024.fits
の計6つの fits をダウンロードし、つなぎ合わせた W43_24um.fits を用います (以前の記事 を参照)。本 fits は 2D ですが、3D の場合でも方法は同様かと思います。
casa
casa の image-analysis-tools を用います。
ターミナルで casa を起動します。パスが通っている場合は、casa と打つだけです。通していない場合は、
your/casa/dir/casa-X.X.X-XXX/bin/casa
で起動します。
例として座標軸に対して60度回転させます。
importfits(fitsimage="W43_24um.fits", imagename="W43_24um.im", overwrite=True)
ia.open("W43_24um.im")
imr = ia.rotate(outfile="W43_24um.rot.im", pa="60.0deg")
imr.done()
ia.close()
exportfits(imagename="W43_24um.rot.im", fitsimage="W43_24um.rot.fits", overwrite=True)
使い方に少しくせがありますが、これだけです。
miriad
ターミナルから miriad を起動し、以下を実行します。
fits in=W43_24um.fits out=W43_24um.mir op=xyin
regrid in=W43_24um.mir out=W43_24um.rot2.mir rotate=60.0
fits in=W43_24um.rot2.mir out=W43_24um.rot2.fits op=xyout
上の画像と同じものが出てくると思います。 (※ header に RESTFREQ がないとエラーになります。ない場合は、miriad の puthd を使うか、astropy.io.fits で開いて header を編集しましょう。)
注意点
基本的には以上です。ただ、注意点として、第一軸と第二軸 (X と Y) の基準点 (つまり、CRPIX1 と CRPIX2) を中心に回転が行われるため、大抵データが見切れます。回転中心を元の fits のど真ん中にしたい場合は、以下の関数を使用してください。
def change_CRPIX12_CRVAL12_center(fits_dir, fits_name, save_dir): # CRPIX12 と CRVAL12 を中心にする関数
from astropy.io import fits
import os
from astropy.wcs import WCS
fits_path = os.path.join(fits_dir, fits_name)
hdu = fits.open(fits_path)[0]
header = hdu.header
w = WCS(header)
NAXIS1_ori = header["NAXIS1"]
NAXIS2_ori = header["NAXIS2"]
CRPIX1_ori = header["CRPIX1"]
CRPIX2_ori = header["CRPIX2"]
if hdu.data.ndim==2:
x_cen, y_cen = w.wcs_pix2world(NAXIS1_ori/2, NAXIS2_ori/2, 0)
elif hdu.data.ndim==3:
x_cen, y_cen, v_tempo = w.wcs_pix2world(NAXIS1_ori/2, NAXIS2_ori/2, 0, 0)
else:
"error: hdu.data.ndim must be 2 or 3."
return
header["CRVAL1"] = float(x_cen)
header["CRVAL2"] = float(y_cen)
header["CRPIX1"] = NAXIS1_ori/2+1
header["CRPIX2"] = NAXIS2_ori/2+1
newfits = fits.PrimaryHDU(hdu.data, header)
newfits.writeto(os.path.join(save_dir, fits_name[:-5]+".center.fits"), clobber=True)
change_CRPIX12_CRVAL12_center("./your/fits/dir/", "W43_24um.fits", "./your/fits/dir/")
それでもどうしても見切れてしまう場合は、astropy で fits を開き、numpy (concatenate 等) で元の fits の周り (食パンでいう耳) に NaN を追加しましょう。そして NAXIS1・2 と CRPIX1・2 を変更しましょう (projection には注意)。
座標とか関係なく、とにかく回転させたい人
astropy.io.fits で fits を開いて、hdu.data を scipy.ndimage.rotate に食べさせましょう。
以上です。
リンク
目次