【以下追記】
numpy のバージョンが 1.23 以上だと、astrodendro 内のパラメータの計算の中で
module 'numpy' has no attribute 'asscalar'
とエラーが出ます。
astrodendro はおそらくもうアップデートされないので、numpy のバージョンを下げる (もしくは自分でastrodendroの中身を修正する) 必要があります。例えば、一時的な解決策として、解析ディレクトリで
pip install 'numpy==1.22.4' -t ./
などとして対応してください。
また、Python3.10などでは、import 時に、
ImportError: cannot import name 'Iterable' from 'collections' (/usr/lib/python3.10/collections/__init__.py)
のエラーが出ることがあります。
import collections.abc
collections.Iterable = collections.abc.Iterable
import astrodendro
とすることで、一時的に対応できます。
【以上追記】
本記事では、astrodendro (Astronomical Dendrograms in Python) の基本的な使い方について記述します。
今回も、例として国立天文台の FUGIN プロジェクトで得られた野辺山45m電波望遠鏡の CO 輝線のアーカイブデータを用います。データは
http://jvo.nao.ac.jp/portal/nobeyama/
から
FGN_03100+0000_2x2_12CO_v1.00_cube.fits
をダウンロードします (重いです)。
2D fits の場合
まずは例によって必要なものを import し、fitsを読み込みます。
from astropy.io import fits
import numpy as np
from matplotlib import pyplot as plt
import aplpy
from astropy.wcs import WCS
hdu = fits.open("~/your/fits/dir/FGN_03100+0000_2x2_12CO_v1.00_cube.fits")[0] # 3D
w = WCS(hdu.header)
2D fitsにするため、以下のような関数を定義します。
def v2ch(v, w): # v(km/s)をchに変える
x_tempo, y_tempo, v_tempo = w.wcs_pix2world(0, 0, 0, 0)
x_ch, y_ch, v_ch = w.wcs_world2pix(x_tempo, y_tempo, v*1000.0, 0)
v_ch = int(round(float(v_ch), 0))
return v_ch
def del_header_key(header, keys): # headerのkeyを消す
import copy
h = copy.deepcopy(header)
for k in keys:
try:
del h[k]
except:
pass
return h
def make_new_hdu_integ(hdu, v_start_wcs, v_end_wcs, w): # 積分強度のhduを作る
data = hdu.data
header = hdu.header
start_ch, end_ch = v2ch(v_start_wcs, w), v2ch(v_end_wcs, w)
new_data = np.nansum(data[start_ch:end_ch+1], axis=0)*header["CDELT3"]/1000.0
header = del_header_key(header, ["CRVAL3", "CRPIX3", "CRVAL3", "CDELT3", "CUNIT3", "CTYPE3", "CROTA3", "NAXIS3"])
header["NAXIS"] = 2
new_hdu = fits.PrimaryHDU(new_data, header)
return new_hdu
以下のように 2D fits を作成します。
integ_hdu = make_new_hdu_integ(hdu, 25.0, 125.0, w)
Dendrogram と、統計量を出すためのものなどを import します。
from astrodendro import Dendrogram
from astrodendro.analysis import PPStatistic
import datetime
以下のように、3つのパラメータを設定します。詳しくはドキュメントを参照してください。
dendro_min_value = 10
dendro_min_delta = 50
dendro_min_npix = 100
以下でDendrogramを実行します。結果は全て d に格納されます。
(一応時間を計測しています。)
t1 = datetime.datetime.now()
print("[dendro] started......", t1)
d = Dendrogram.compute(integ_hdu.data, min_value=dendro_min_value, min_delta=dendro_min_delta, min_npix=dendro_min_npix, verbose=False)
t2 = datetime.datetime.now()
print("[dendro] finished.....", t2)
print("[dendro] total time... ", (t2 - t1).total_seconds(),"sec")
ここまでやっておいて今更言うのもあれですが、以上を ipython で実行していると
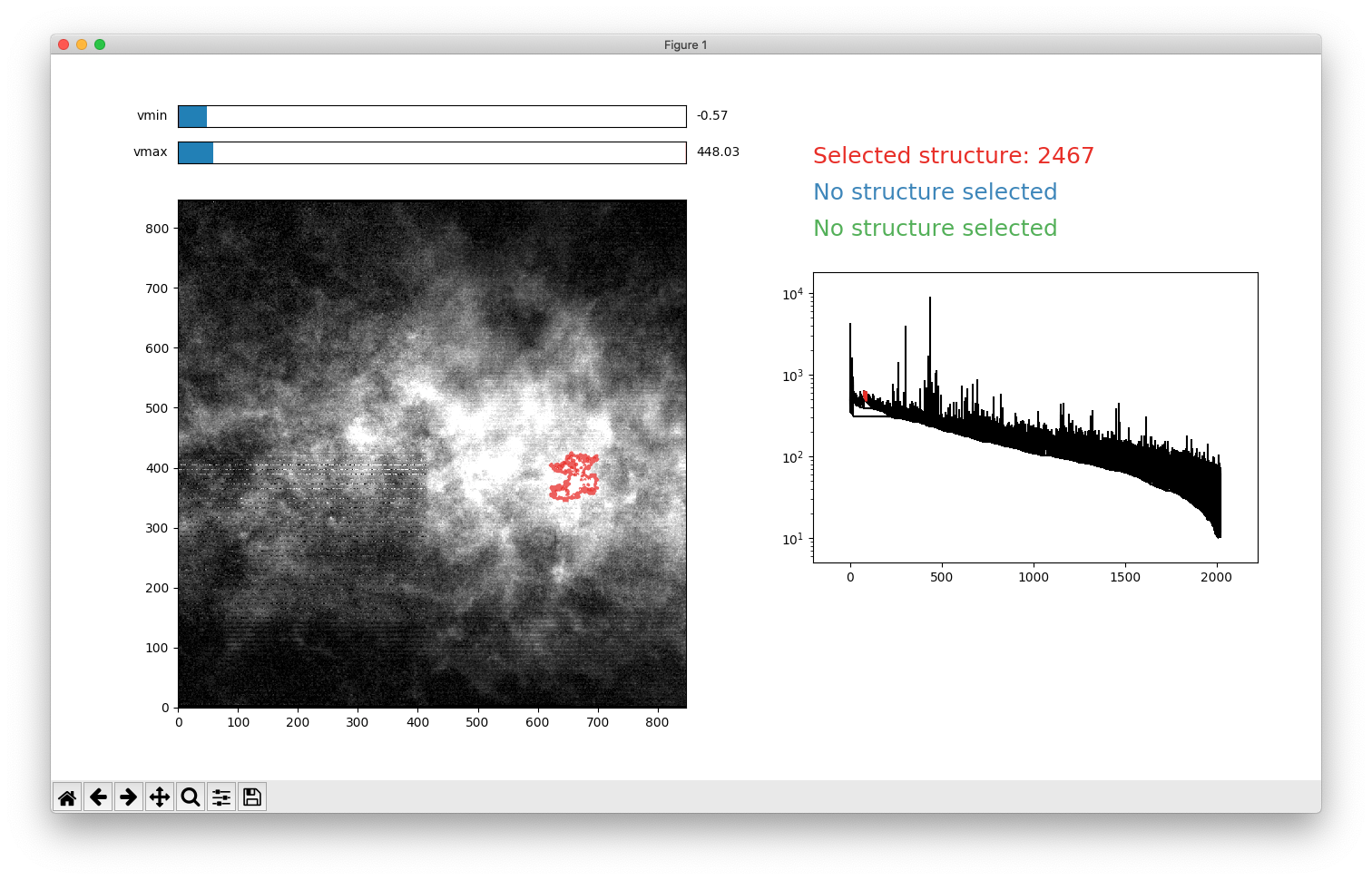
d.viewer()
plt.show()
を実行することで GUI で結果を確認できます。(Jupyter だとうまく動きません。)
カタログ化
同定した構造をカタログ化したいと思います。標準では
from astrodendro import pp_catalog
というものが備わっているのですが、少し扱いづらいので、今回は pandas を使って独自にカタログを作る方法をとることにします。
import pandas as pd
まず、どれだけの構造が同定されたか個数を確認します。
print(len(d))
# 1002
d.trunk や d.leaves と実行するとそれぞれ trunk と leaf の structure のリストが返ってきます (詳細はドキュメントを参照)。数を以下で確認します。
print(len(d.trunk))
# 6
print(len(d.leaves))
# 504
それでは、全 leaf に対してパラメータを出してカタログを作ろうと思います。
(追記)
trunk に対する vmax は、ドキュメントによれば、sub-structure を取り除いた pixel/voxel たちでの最大値だそうです。一方、 get_peak() はその structure 内での最大値の場所と値を返してくれます。
w_integ = WCS(integ_hdu.header)
x_peak, y_peak, x_cen, y_cen, val_min, val_max = [], [], [], [], [], []
index_list, ancestor = [], []
n_vox, area_ellipse, area_exact, minor_sigma, major_sigma, position_angle = [], [], [], [], [], []
radius, total_flux = [], []
for s in d.leaves:
stat = PPStatistic(s)
index_list.append(s.idx)
ancestor.append(str(s.ancestor.idx))
y_ch, x_ch = s.get_peak()[0]
x_peak_, y_peak_ = w_integ.wcs_pix2world(x_ch, y_ch, 0)
x_peak.append(round(float(x_peak_), 6))
y_peak.append(round(float(y_peak_), 6))
x_cen_ch, y_cen_ch = stat.x_cen.value, stat.y_cen.value
x_cen_, y_cen_ = w_integ.wcs_pix2world(x_cen_ch, y_cen_ch, 0)
x_cen.append(round(float(x_cen_), 6))
y_cen.append(round(float(y_cen_), 6))
val_min.append(round(s.vmin, 6))
#val_max.append(round(s.vmax, 6))
val_max.append(round(s.get_peak()[1], 6))
ind = s.indices()
n_vox.append(len(ind[0]))
area_ellipse.append(round(stat.area_ellipse.value, 6))
area_exact.append(round(stat.area_exact.value, 0))
minor_sigma.append(round(stat.minor_sigma.value, 6))
major_sigma.append(round(stat.major_sigma.value, 6))
position_angle.append(round(stat.position_angle.value, 6))
radius.append(round(stat.radius.value, 6))
total_flux.append(round(stat.stat.mom0(), 6))
results_leaves = pd.DataFrame({
'id':index_list, # ID
'ancestor':ancestor, # (最も)先祖のID
'x_peak':x_peak, # peak の位置のx座標
'y_peak':y_peak, # peak の位置のy座標
'x_cen':x_cen, # 重心位置のx座標
'y_cen':y_cen, # 重心位置のy座標
'val_min':val_min, # 構造の持つ最小値
'val_max':val_max, # 構造の持つ最大値
'n_vox':n_vox, # 構造が使っている pixel 数
'area_ellipse':area_ellipse, # 構造の重みつき 1 sigma の楕円面積 (pixel^2)
'area_exact':area_exact, # 構造が実際に使っている面積 (pixel^2)
'minor_sigma':minor_sigma, # 楕円の短軸 (pixel)
'major_sigma':major_sigma, # 楕円の長軸 (pixel)
'position_angle':position_angle, # 楕円の position angle (degree)
'radius':radius, # 楕円の長軸短軸の相乗平均 (pixel)
'total_flux':total_flux # 構造の持つ値の総和
})
results_leaves という名前の pandas DataFrame が出来上がりました。
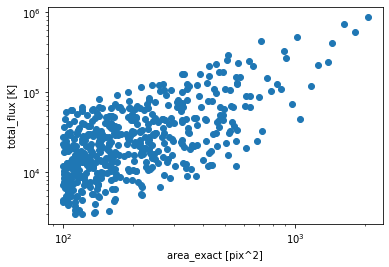
試しに何かplotしてみます。
plt.scatter(results_leaves["area_exact"], results_leaves["total_flux"])
plt.xscale("log")
plt.yscale("log")
plt.xlabel("area_exact [pix^2]")
plt.ylabel("total_flux [K]")
plt.show()
results_leaves.to_csv("leaves_test.csv")
で保存できます。読み込みは
results_leaves = pd.read_csv("leaves_test.csv")
です。
3D fits (cube) の場合
2D の場合と使用方法は変わりません。 (ただし、もちろん計算は重いです。)
カタログ化の際は
from astrodendro.analysis import PPVStatistic
を使って、v に関するパラメータも格納できるように上のコードを適宜書き換えましょう。
カタログの map 上への plot
aplpy と、aplpy の show_markers を使えば簡単にできます。
fig = plt.figure(figsize=(8, 8))
f = aplpy.FITSFigure(integ_hdu, slices=[0], convention='wells', figure=fig)
f.show_colorscale(vmin=1, vmax=500, stretch='log', cmap="Greys", aspect="equal")
f.show_markers(results_leaves["x_peak"], results_leaves["y_peak"], marker="x", c="r")
plt.show()
構造の輪郭のコントアを引きたい場合は、
mask = np.zeros(integ_hdu.data.shape, dtype=bool)
for s in d.leaves:
mask = mask | s.get_mask()
mask_hdu = fits.PrimaryHDU(mask.astype('short'), integ_hdu.header)
でコントアの hdu を作った後に、
fig = plt.figure(figsize=(8, 8))
f = aplpy.FITSFigure(integ_hdu, slices=[0], convention='wells', figure=fig)
f.show_colorscale(vmin=1, vmax=800, stretch='log', cmap="Greys", aspect="equal")
f.show_contour(mask_hdu, colors='r', linewidths=0.5)
plt.show()
以上です。
リンク
目次