半年ほど機械学習に触れてなかったため、忘れたことを復習しました。
欠損値を扱う方法について
提供してもらったデータセットには、時折NaN(Not a Number)という欠損値データが含まれていることがある。
これを扱う方法について記載する。
サンプルデータの作成、NaNの扱い
# サンプルデータを作成
import numpy as np
import pandas as pd
# ハードディスク上にcsvファイルを作成せずに実行したいのでStringIO関数をインポート
from io import StringIO
# csvファイルを作成する
csv_sample_data = '''
A,B,C,D
1.0,,3.5,5.0
4.0,,6.0,9.0
big,small, ,normal
red,blue,green,white
20,30,,40
'''
# DataFrameオブジェクトに格納
df = pd.read_csv(StringIO(csv_sample_data))
df
# print(df) # jupyterを使っている際、テーブル形式で表示させたくない場合(そんな人あまりいないと思うが)
# Kaggleのチュートリアルなどではprint文を使用していたが、ターミナルやjupyterでは無くても実行できる
-------------------------------------------------
A B C D
0 1.0 NaN 3.5 5.0
1 4.0 NaN 6.0 9.0
2 big small normal
3 red blue green white
4 20 30 NaN 40
pandasのread_csv関数は、CSVフォーマットファイルを読み込んだ時、欠損値を上記のようにNaNに変換する。
ただし、**[Big, small, ,normal]**のような半角スペースを空けるとNaNに変換してくれない(半角スペースも1特徴量として扱われるため)。
# 各列の欠損値(NaN)をカウントする
df.isnull().sum()
-------------------------------------------------
A 0
B 2
C 1
D 0
dtype: int64
DataFrameオブジェクトのisnull().sum()関数を使うことで各列に含まれるNaNの数を数えることができる。
では、NaN(欠損値)はどう処理すれば良いのだろうか。
解決策1) データセットから欠測値を取り除く
邪魔なのでデータセットから捨てる。
この際、行列が崩れないように1行単位で取り除く。
# 元のデータセット
df
-------------------------------------------------
A B C D
0 1.0 NaN 3.5 5.0
1 4.0 NaN 6.0 9.0
2 big small normal
3 red blue green white
4 20 30 NaN 40
# NaNを含む行を削除する
df.dropna()
-------------------------------------------------
A B C D
2 big small normal
3 red blue green white
DataFrameオブジェクトのdropna関数を使用することで、1行目と4行目が取り除かれて表示されている。
他にも引数を指定することで削除する内容を変更できる。
詳しい内容については公式ドキュメントを参照すること。
pandas.DataFrame.dropna
# NaNを含む"列"を削除する
df.dropna(axis='columns')
# df.dropna(axis=1) # こちらの指定方法でも可
# 行を削除する場合、axis=0 または axis="index"でも可
-------------------------------------------------
A D
0 1.0 5.0
1 4.0 9.0
2 big normal
3 red white
4 20 40
# B列の中でNaNが含まれる行だけを削除する
# 余談ですが、僕はこの模擬データセットを作ったときにカンマの後に半角スペースを入れたせいで
# 列名が異なってしまったためKeyErrorが発生しました。
# 見やすさのためにカンマの後に半角スペースを入れないようにしましょう。
df.dropna(subset=['B'])
-------------------------------------------------
A B C D
2 big small normal
3 red blue green white
4 20 30 NaN 40
しかし、これでは他の特徴量も一緒に捨てられてしまう。
そのため、有益な情報を失ってしまう可能性がある。
ある行に含まれるNaNの比率が90%を超える場合などに使用に限定しよう。
トレーニングデータセットとテストデータセットから同じ列を削除する
データサイエンティストが扱うデータセットは多くの場合、トレーニングデータセットと
テストデータセットを持っている。
そのため、2つのデータセットに差異が発生しないよう、両方から同じ列を削除するようにする。
やり方は簡単、削除する列をリストで持っておき、df.drop()関数で削除する。
delete_col_list = ['B', 'D']
df.drop(delete_col_list, axis=1)
-------------------------------------------------
A C
0 1.0 3.5
1 4.0 6.0
2 big
3 red green
4 20 NaN
今回は1つしかサンプルデータセットを作成していないが、
df.drop(delete_col_list, axis=1)を別のDataFrameオブジェクトで実行すれば良い。
解決策2) 欠測値の補完
有益なデータを削除しないように、NaNを同じ列の値の平均値などで補完する。
これはscikit-learnのImputerクラスを使用すると楽。
sklearn.preprocessing.Imputer
なお、平均(mean)の他に以下もstrategy引数として用意されている。
- 中央値(median)
- 最頻値(most_frequent)
from sklearn.preprocessing import Imputer
imr = Imputer(missing_values='NaN', strategy='mean', axis=0)
# この後の説明のため、新規にDataFrameオブジェクトを作成
df2 = pd.DataFrame({"A":[1.0, 3.0, np.nan,np.nan],
"B":[1.0, np.nan,5.0,6.0],
"C":[9.0,np.nan,13.0, 2.0],
"D":[9.0,np.nan,5.0, np.nan]
})
df2
-------------------------------------------------
A B C D
0 1.0 1.0 9.0 9.0
1 3.0 NaN NaN NaN
2 NaN 5.0 13.0 5.0
3 NaN 6.0 2.0 NaN
# 平均値で補完する
imputed_data = imr.fit_transform(df2) # fit()とtransform()を1行で実行する関数
imputed_data
-------------------------------------------------
array([[ 1., 1., 9., 9.],
[ 3., 4., 8., 7.],
[ 2., 5., 13., 5.],
[ 2., 6., 2., 7.]])
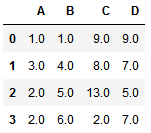
jupyter-notebookでグラフィカルに表示する
df2 = pd.DataFrame(data=imputed_data[0:,0:],
index=range(4),
columns=['A','B','C','D'])
df2
各列間でNaNを除いた値で平均値を算出し、NaNに補完してくれている。
では、文字列を含むDataFrameオブジェクトではどうだろうか。
imputed_data2 = imr.fit_transform(df)
imputed_data2
---------------------------------------------------------------------------
ValueError Traceback (most recent call last)
<ipython-input-21-4a8a719ead77> in <module>()
----> 1 imputed_data2 = imr.fit_transform(df)
2 imputed_data2
/.../site-packages/sklearn/base.py in fit_transform(self, X, y, **fit_params)
492 if y is None:
493 # fit method of arity 1 (unsupervised transformation)
--> 494 return self.fit(X, **fit_params).transform(X)
495 else:
496 # fit method of arity 2 (supervised transformation)
/.../site-packages/sklearn/preprocessing/imputation.py in fit(self, X, y)
154 if self.axis == 0:
155 X = check_array(X, accept_sparse='csc', dtype=np.float64,
--> 156 force_all_finite=False)
157
158 if sparse.issparse(X):
/.../site-packages/sklearn/utils/validation.py in check_array(array, accept_sparse, dtype, order, copy, force_all_finite, ensure_2d, allow_nd, ensure_min_samples, ensure_min_features, warn_on_dtype, estimator)
380 force_all_finite)
381 else:
--> 382 array = np.array(array, dtype=dtype, order=order, copy=copy)
383
384 if ensure_2d:
ValueError: could not convert string to float: 'white'
NaNを同じ列の数値の平均値で補完するため、文字列である'white'をfloat型に変換できないと言われてしまった。
そのため、この方法ではできない。
このようなカテゴリの値を含むデータセットを補完するにはどうするべきだろうか。
僕が考える対処法は以下の通り(素人の回答です)。
名義的特徴量(red, blue, green, whiteのような大小関係のない特徴量)の場合
- 「その他」というカテゴリを作成し、欠損値をそれに置き換える
- one-hotエンコーディングで数値に置き換える
順序特徴量(big, normal, small)の場合
- 一番小さなカテゴリ値としてマッピングする
他にもっと良い回答があると思います。