PLINKの---pcaコマンドで主成分分析を実施すると、
1.output.eigenvec
2.output.eigenval
という2つのファイルが出力されます。
今回はこれらの2つのファイルを使用して、主成分分析の結果を可視化します。
①Pythonで必要なライブラリをインポート
import matplotlib.pyplot as plt
import seaborn as sns
import pandas as pd
import japanize_matplotlib
②データの読み込み
pandasのread_csvで、2ファイルを読み込みます。
.eigenvalの値から、PC1とPC2がどれだけの割合を説明できるのかを計算します。
ついでに、今回の主成分分析で使用したSNP数も.mapファイルから計算しておきます。
# データの読み込み
df = pd.read_csv('output.eigenvec', sep=' ', header=None)
# PC1とPC2の説明量を計算する
PC = pd.read_csv('output.eigenval', sep=' ', header=None)
su = PC[0].sum()
p1 = PC.iloc[0, 0]
PC1 = p1 / su * 100
p2 = PC.iloc[1, 0]
PC2 = p2 / su * 100
# SNP数を計算する
ma = pd.read_csv('output.map', sep=' ', header=None)
snp = len(ma)
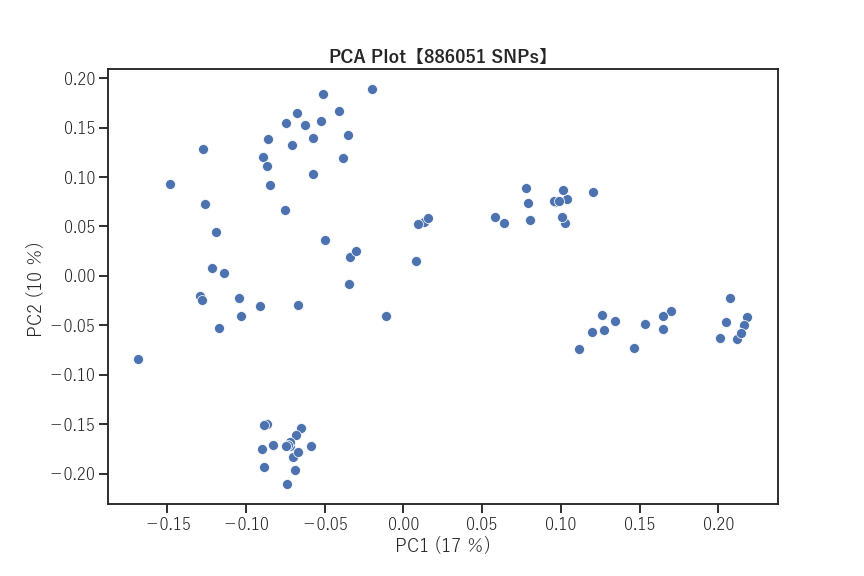
③PCAプロットの作成
先ほど計算した、SNP数や説明量をグラフに反映させました。
sns.set(font='Yu Gothic', context="talk", style='ticks')
plt.figure(figsize=(12,8))
sns.scatterplot(x=2, y=3, data=df, s=100)
plt.title("PCA Plot【%i SNPs】" %snp, fontweight="bold")
plt.xlabel('PC1 (%i %)' %PC1)
plt.ylabel('PC2 (%i %)' %PC2)
plt.savefig('Output.png') #PNGで画像を保存
集団が複数ある場合などは、sns.scatterplotのhueで色分けも行えます。
全体
import matplotlib.pyplot as plt
import seaborn as sns
import pandas as pd
import japanize_matplotlib
# データの読み込み
df = pd.read_csv('output.eigenvec', sep=' ', header=None)
# PC1とPC2の説明量を計算する
PC = pd.read_csv('output.eigenval', sep=' ', header=None)
su = PC[0].sum()
p1 = PC.iloc[0, 0]
PC1 = p1 / su * 100
p2 = PC.iloc[1, 0]
PC2 = p2 / su * 100
# SNP数を計算する
ma = pd.read_csv('output.map', sep=' ', header=None)
snp = len(ma)
sns.set(font='Yu Gothic', context="talk", style='ticks')
plt.figure(figsize=(12,8))
sns.scatterplot(x=2, y=3, data=df, s=100)
plt.title("PCA Plot【%i SNPs】" %snp, fontweight="bold")
plt.xlabel('PC1 (%i %)' %PC1)
plt.ylabel('PC2 (%i %)' %PC2)
plt.savefig('Output.png') #PNGで画像を保存
以上、PLINKの主成分分析の結果からPCAプロットを作成する方法でした。