2020年9月4日から約3ヶ月にわたってMechanisms of Action (MoA) PredictionというコンペティションがKaggleで開催されました。この記事では、このコンペの上位解法と私の解法の要点を紹介します。
なお、この記事は創薬 (dry) Advent Calendar 2020の16日目の記事です。
コンペの概要
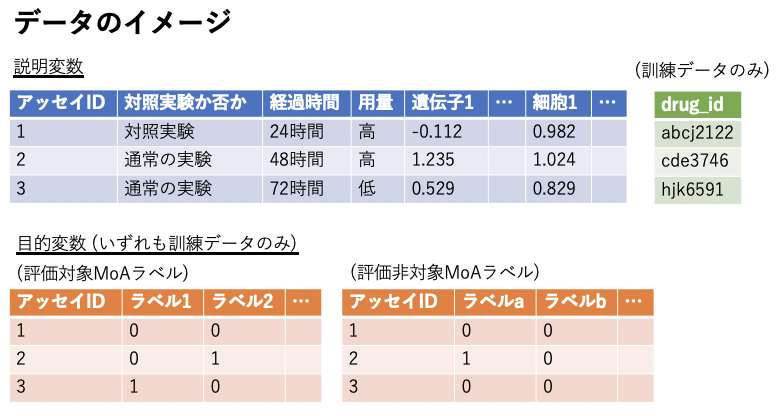
一言でいうと、薬剤で処理した (1)ある細胞の772種類の遺伝子発現データと (2)100種類の細胞の生存率のデータを主な特徴量として、作用機序(MoA)を予測するという問題設定でした。このコンペを通じて開発された予測モデルを適用することで、作用機序が不明な薬剤の作用機序を推定することができますし、場合によっては既知の薬剤の未知の作用を発見すること (drug repositioning/re-purposing) への応用も期待できると思います。
この手のデータに興味がある人を想定して、データの詳細について、もう少し説明しましょう。まず遺伝子発現のデータは各カラムが何らかの遺伝子に対応する形式のデータではありますが、遺伝子名は匿名化されているため、外部データと照合するような手法は封じられていました。細胞の生存率のデータも同様で、細胞名(cell-lineの名称)は匿名化されています。また、これらのアッセイデータとは別に、(アッセイ時の実験条件として(a) アッセイ時点での薬剤処理からの経過時間(time)(24/48/72時間の3種類)と(b)薬剤の用量(dose)(低/高用量の2種類)と(c)実際の薬剤を使った実験かそれとも溶媒などのvehicleだけを使った対照実験かの種別の情報が付与されていました。
目的変数としては1事例について複数のMoAのラベル1が付与された、マルチラベル分類問題でした。また、評価対象になる206のラベルとは別に、評価対象外の402のラベルが提供されていました。後者に関しては、ただこれを予測しても意味はないですし、訓練データのみでテストデータには与えられていないために特徴量として利用することもできないので、これをどう活用するかが一つのポイントだったと思います。
コンペ開催中に追加で開示された情報として、各事例がどの薬剤についてのアッセイなのかを区別するためのdrug_idがありました。これも具体的な薬剤名が分からないように匿名化されていて訓練データについてのみ開示されました。背景として薬剤によっては複数回測定されていて、どの薬剤がどの事例なのか不明だったのが区別できるようになったということです。これは適切なクロス・バリデーションを行う上では有用な情報だったと思います。
1位の解法
1位は、Mark Peng氏、poteman氏、kibuna氏、Nischay Dhankhar氏の4名からなるチームでした。1位の解法の全容は下記で公開されています。:
https://www.kaggle.com/c/lish-moa/discussion/201510
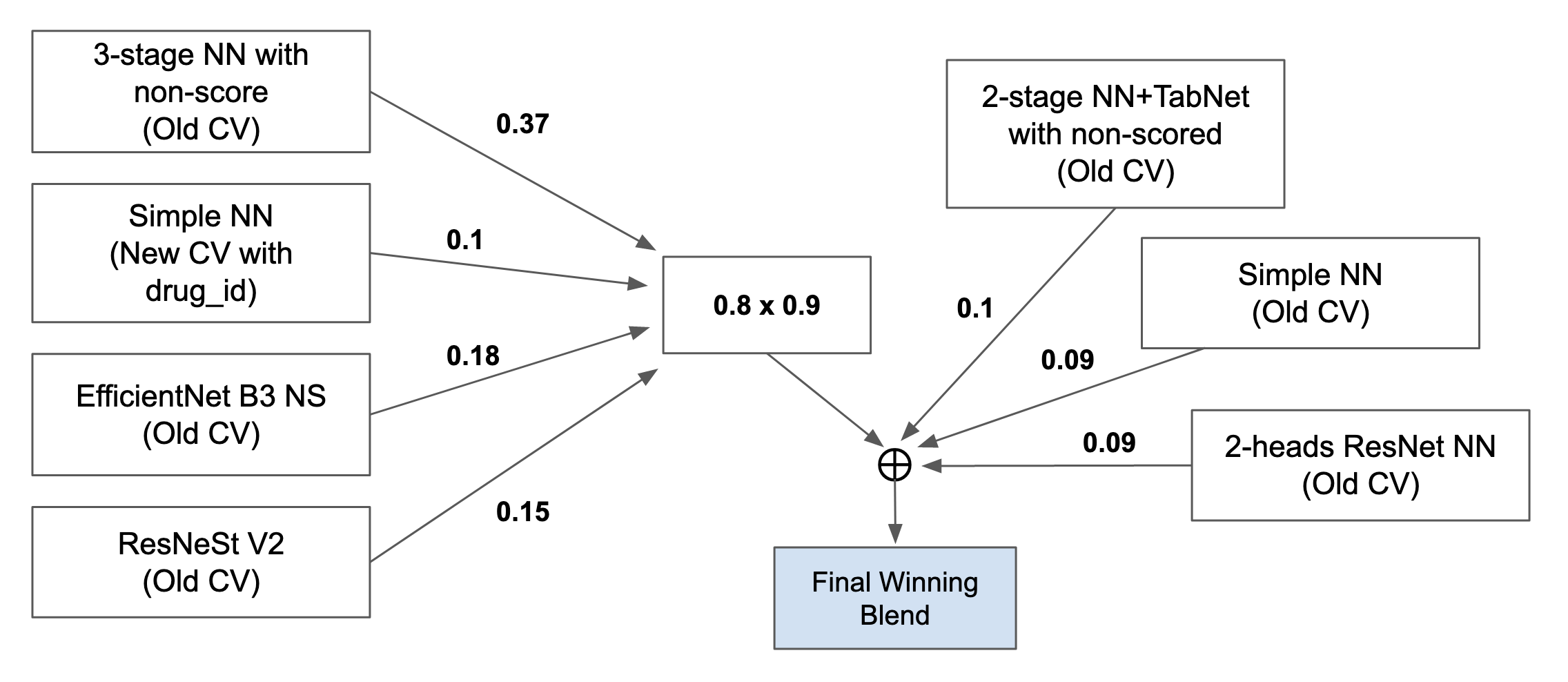
引用元: https://www.kaggle.com/c/lish-moa/discussion/201510
こちらのチームは、上図のように7種類のニューラルネットのモデルをblendingしており、効果的にアンサンブル学習を利用していたのが特徴でした。細かく見ていくと、一部のモデルはその内部でstackingを行っているので、多層的なアンサンブルになっています。このとき評価対象外のラベルについてもstackingのメタ特徴量として活用しています。モデル間の出力の相関も考慮してモデルを選択していたそうで、そういった実験管理も含めたチームワークが勝利の背景にはありそうです。
このチームのもう一つの特徴はDeepInsightというモデルを組み込んでいた点です(上図の「EfficientNet B3 NS」と「ResNeSt V2」に相当)。DeepInsightは過去に理化学研究所の研究グループが2019年に論文で発表していた手法2で、簡単に言うと、遺伝子発現のデータをPCAやt-SNEで画像化し、ある回転処理を加えた上で、これをCNNをベースのニューラルネットの入力とするという手法です。
この方法は、このコンペのdiscussionでも報告されていたのですが、実を言うと私自身はこの手法については懐疑的であり、全く試していませんでした。画像化を正当化する理由が想像できなかったということと、画像化によって個々の遺伝子が区別できなくなるなど大きな情報の損失があるように思われたことが理由でした。しかしながら、このチームはコンペの過程での分析と最終的な結果を通じて、この手法こそが優勝の鍵となったことを示しており、私にとってはちょっとした驚きでした。
2位の解法
2位はtmp氏のsoloでの受賞となりました。しかもkaggleは初参加のようで、快挙と言えると思います。
この解法は、3種類のニューラルネットのblendingの結果でしたが、決め手になったのはユニークで強力な1D CNNのシングル・モデルでした。2位の解法の全容は下記で公開されています。:
https://www.kaggle.com/c/lish-moa/discussion/202256
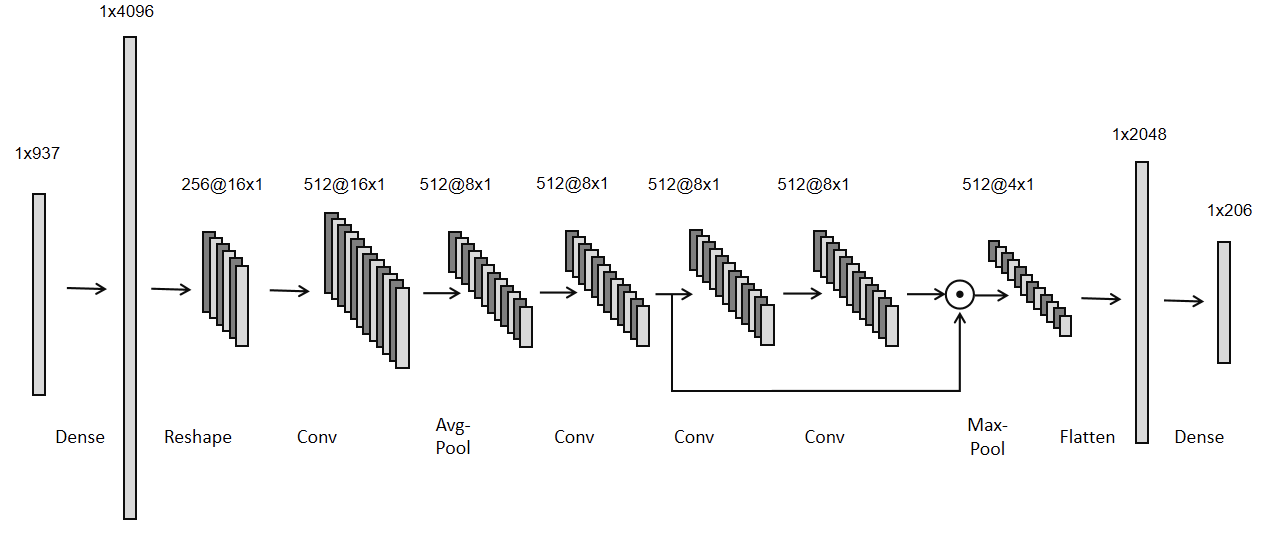
(引用元:https://www.kaggle.com/c/lish-moa/discussion/202256)
入力データを全結合層でより高次元にしたあと、これをreshapeして、多チャンネルの1次元の系列データとしてから、オーソドックスなCNNで処理していく構造となっています。入力の直後の全結合層が、入力データを系列データとして適切な順序をもったテンソルに変換する機能をもっていると解釈できると思います。
さらっと説明されていますが、この系列とは何を意味するのかは真剣に考える価値があると思っています。どのような表形式のデータであっても、このようなアーキテクチャのニューラルネットに入力すれば、形式上は系列データに変換できるわけですが、常にこの方法が奏功するとは考えにくいのではないでしょうか。言い換えれば、このニューラルネットが高い予測性能を示したということは、このコンペのアッセイ・データには系列として取り扱うべき性質が内在していたということだと思います。
なお、2位の解法では、評価対象外のラベルのうち、評価対象のラベルと相関が高いものに限って、これをモデルの事前学習(pre-training) に利用していました。
私の解法
私の解法は成績が振るわず、4373チーム中722位という結果にとどまりました。アンサンブルは未実施で、シングルモデルに終始してしまったことが敗因の一つだと思っています。最終的なスコアは0.01625で、1位の解法のシングルモデルと遜色はないものの、2位の解法の1D CNNの水準には及ばず、中途半端な結果に終わってしまった感は否めません。とはいえ、コンペ参加を通じていくつかの知見は得られたので、手前味噌ながら紹介したいと思います。
ドメインシフト対策
まず、データの前処理としては、経過時間(time)の影響を軽減することを目的として、時間毎の3通りのサブセットにデータを分割し、サブセットごとにデータを標準化するということを行いました。一般に平均を0、分散を1とする標準化では全データの平均で引き算して分散で割り算する処理を行いますが、ここではサブセット毎に対照実験だけを利用して求めた平均と分散をこの処理に用いました。
この特殊な標準化には時間の経過に伴う特徴量の変動をキャンセルし、経過時間に関わらず同じMoAを呈する薬剤のアッセイであれば、特徴空間上の近傍の座標に位置するように調整する狙いがありました。画像の問題に例えれば、暗いところで撮った写真と明るいところで作った写真が混ざっているデータセットで、画像の明度を統一する操作を行ったことに相当し、いわゆるドメイン・シフトに対する簡易的な対策になったものと期待されます。この処理によって、一定程度のスコア改善が認められました。なお、同様の操作を時間のみならず用量についても試行しましたが、こちらについては効果はありませんでした。対照実験については用量の差に意味は見出しづらくドメインシフトを引き起こすとは考えづらいので、これもまた合理的な結果だと思います。
DenseNet-likeなニューラルネット
モデルのアーキテクチャとしては、同様の遺伝子発現データを取り扱った論文を参考に、DenseNetに着想を得たskip-connectionを多用するネットワークを採用しました。ただし、これは参考にした論文もそうなのですが、オリジナルのDenseNetのように畳み込み層をもつネットワークではなくて、全結合層を基本にしたアーキテクチャとなっていた点が1位の解法に含まれていたdeepInsightや2位の1D CNNとは異なるところです。このアーキテクチャを採用したことで、単純なMLPよりは良いスコアが出せました。
同一薬剤の事例の混合によるデータ拡張
あるdrug_idが付与された複数のアッセイデータを混合して、架空のアッセイ事例を作るというデータ拡張 (data augmentation)を行いました。drug_idが同一であれば、対応するラベルも同一であるため、この架空のアッセイにも同一のラベルを付与し、実際のアッセイのデータとともに訓練に利用しました。この工夫でCVもLBともにスコアの改善に繋がりました。
なお、ここでは混合の方法として、一様分布に従う乱数を重みにして、同一drug_idのデータを重み付き算術平均しています。この方法は、クラス不均衡の対策のためのテクニックであるSMOTEに近いため、少数クラスの水増しによってクラス不均衡が緩和されたことによる効果も生じていたのかもしれません。
最後に
1位の解法は論文で提案されていた手法に客観的な裏付けを与え、2位の解法はデータが内包する潜在的な性質について示唆を含むものでした。総じて、論文という伝統的な形式での研究開発とコンペティションという比較的新しい技術的潮流の相互作用に期待感をもたせる内容だったと思います。上位手法の凄さや面白さは自分自身が同じ問題に取り組んだからこそ実感をもって理解できるところがありますし、最新技術のキャッチアップや問題そのものに対する考察を深める機会としても、コンペティション参加は有意義だと感じました。
-
具体的には、xxx_inhibitorやxxx_agonistのように、細胞内のどの標的にどのような作用を及ぼすかを数単語で記述したラベルが多かったです。一方で、本来のMoAの意味である作用機序=「薬剤が作用する仕組み」の概念からは逸脱するものが含まれていました。例えば、laxative (下剤)やantimalarial (抗マラリア活性)というラベルは、MoAというよりは効能に近い概念です。しかも、ちょっと考えれば分かることですが、細胞に下剤を振りかけても細胞のお腹がゆるくなるということはなく、細胞レベルのアッセイでは本質的に検証不可能なものだと思います。想像するに、これらのラベルは、何らかの既存薬に関するデータベースに依拠するものであって、アッセイデータの提供元が測定に基づいて独自にラベル付けしたものではないのだと思います。 ↩
-
日本語のプレス・リリース: https://www.riken.jp/press/2019/20190806_3/index.html ↩