missing value
欠損値を扱う場合、補完をする場合とそのままNAとして扱う場合があります。
bayesean networkの場合はそれ自身が補完に利用することもできるため、NAとしてそのまま渡したい場合があります。
Bayes Serverでは以下のように、欠損値をそのまま扱うことができます。
基本的にはEvidenceがすべてそろわなくても、学習、推論ができるという事なので、他のモデルよりも欠損値の扱いは、straightだと思います。
GUI では
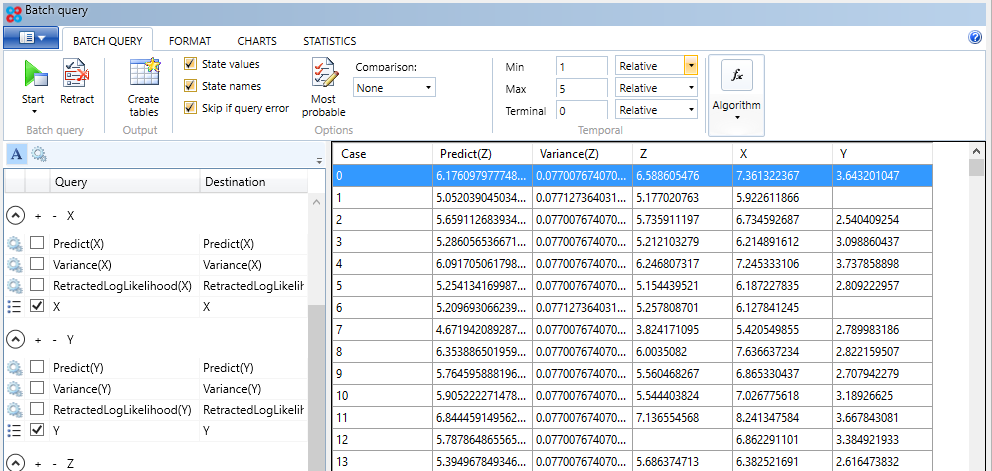
例えば、Tutorialにあるような、空白セルのあるExcelファイルを読み込んでも大丈夫です。
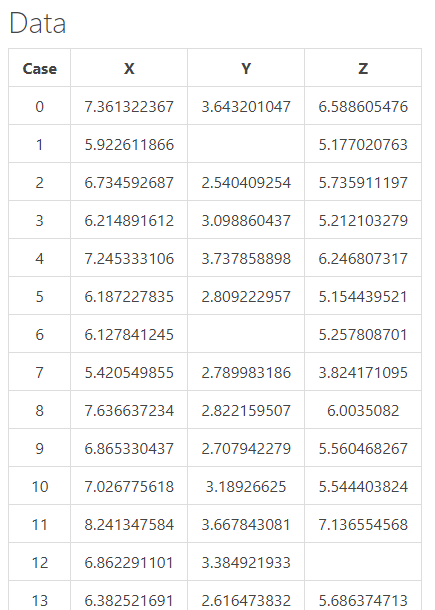
Batchクエリーなどの画面で見られますデータはこのように空白になったまま読み込まれています。
プログラム(C#)では
ネイティブのC#のコードを見ると、以下のように まずdataReaderCommandというものでevidenceReaderCommandに渡すDataTableを作成しています。
var dataReaderCommand = CreateDataReaderCommand();
var variableReferences = network.Variables.Select(v => new VariableReference(v, ColumnValueType.Name, v.Name)).ToArray();
var evidenceReaderCommand = new EvidenceReaderCommand(
dataReaderCommand,
variableReferences,
new ReaderOptions());
CreateDataReaderCommandという関数は以下のようにDataTableを単純に作成しているだけのように見えます。
var data = new DataTable();
data.Columns.Add("A", typeof(string));
data.Columns.Add("B", typeof(string));
data.Columns.Add("C", typeof(string));
data.Rows.Add("True", "False", "True");
data.Rows.Add("True", "True", "True");
DataTableは、DataRowをコレクションの要素として持っていますが、DataRow自体はコレクションではなくてIItemArrayというインターフェイスを持っているだけのようです。
Columnが先に指定されているわけですから、列方向には、NULLを要素に入れてもいけそうな雰囲気があります。
単純なList<>などのコレクションだと普通はNULLは入らないのですが、どうやって実装するべきかは良くわかりません。(C#のdoubleにはNaNがあるんですが、これを使うとはちょっと思えない)
実はC#は使っていないので、ここは宿題にします。たぶんNULLいけそうな雰囲気ってことだけ。
プログラム(R)では
Rから呼び出しているのですが、まずそのラップ先であるJavaはどうか?
同じコードのJava版を見ますと、以下のように、同じようなインターフェイスを持っているようです。
DataTable data = new DataTable();
DataColumnCollection columns = data.getColumns();
columns.add("A", String.class);
columns.add("B", String.class);
columns.add("C", String.class);
DataRowCollection rows = data.getRows();
rows.add("True", "False", "True");
rows.add("True", "True", "True");
Rから同じことをするには、ここにあるように、まず同じdata.frameを作ります。
注意点はRのdata.frameにはNULLは入れられず、NAが欠損値として扱われることです。
RからJavaラッパーを呼ぶヘルパーコードが公開されていますが、以下のRのdata.frameからJavaラッパー経由でdataTableを作る関数が、ややおおざっぱすぎます。
toDataTable <- function(df) {
dt <- new(DataTable)
dfTypes <- sapply(df, typeof)
dfClasses <- sapply(df, class)
dfNames <- names(df)
columnCount <- length(dfNames)
for (i in 1:columnCount)
{
dt$getColumns()$add(dfNames[i], toJavaClass(dfTypes[i], dfClasses[i]))
}
for (r in 1:nrow(df))
{
values <- lapply(df[r,], function(x) {
return(toJavaObject(x))
})
values <- .jarray(values, contents.class = "java.lang.Object")
dt$getRows()$add(values)
}
return(dt)
}
このままNA入りのdata.frameを渡すと、なぜか”NaN入りのDataTableは受け付けないぞ!"というエラーで怒られます。
これだと、toJavaObject()内では、NAを無理やりJavaのNAに変換しようとするようなので、うまくNULLとして伝わっていないようです。
差し当たり、以下のように欠損値ぽいものが来たら、全部NULLに変換してしまうことにしたら、うまくいきました。
values <- lapply(df[r,], function(x) {
if (!is.null(x) & !is.nan(x) & !is.na(x)) {
return(toJavaObject(x))
} else {
return(NULL)
}
})
本当にNULLでいいのかどうか、C#で叩いてみて検証も後日行いたいと思います。