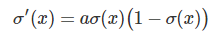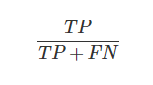1. 線形回帰モデル
線形とは何か
比例:比例関係であれば
y = Ax + B(2次元)
z = Ax + B y + C(3次元)
※n次元空間における超平面の方程式
y = a0 + a1x1 + a2x2 + ・・・+an-ixn-i
回帰問題
ある入力(離散あるいは連続値)から出力(連続値)を予測する問題
・直線で予測▶線形回帰
・曲線で予測▶非線形回帰
回帰で扱うデータ
入力(各要素を説明変数または特徴量と呼ぶ)
・m次元のベクトル(m=1の場合はスカラ)
x=(x1,x2,x3,⋯,xm)T∈Rm
出力(目的変数)
・スカラー値(目的変数)
y∈R
・線形回帰モデル
回帰問題を解くための機械学習モデルの一つ
教師あり学習(教師データから学習)
入力とm次元パラメータの線形結合を出力するモデル
・線形結合(入力とパラメータの内積)
入力ベクトルと道のパラメータの各要素を掛け足し合わせたもの
入力ベクトルとの線形結合に加え、切片も足し合わせる
(入力のベクトルが多次元でも)出力は1次元(スカラ)となる
・モデルのパラメータ
モデルに含まれる推定すべき未知のパラメータ
特徴量が予測値に対してどのように影響を与えるかを決定する重みの集合
◆正の重みをつける場合、その特徴量の値を増加させると、予測の値が増加
◆重みが大きければ(0であれば)、その特徴量は予測に大きな影響力を持つ(全く影響しない)
切片
◆y軸との交点を表す
データの分割
学習用データ:機械学習モデルの学習に利用するデータ
検証用データ:学習済みモデルの精度を検証するためのデータ
なぜ分割するか
モデルの汎化性能(Generalization)を測定するため
データへの当てはまりの良さではなく、未知のデータに対してどれくらい精度が高いかを測りたい
線形回帰モデルのパラメータは最小二乗法で推定
◆平均二乗誤差(残差平方和)
・データとモデル出力の二乗誤差の和
・パラメータのみに依存する関数(データは既知の値でパラメータのみ未知)
MSE=\frac{1}{n}\sum_{i=1}^{n}(yi−\hat{yi})^2
※二乗損失は外れ値に弱い為事前に外れ値がないか確認する必要がある
◆最小二乗法
・学習データの平均二乗誤差を最小とするパラメータを探索
・学習データの平均二乗誤差の最小化は、その勾配が0になる点を求めれば良い
wwに対して微分したものが0となるwwの点を求める。つまり勾配が0になる点を求める。
∂∂wMSEtrain=0
∂∂wMSEtrain=0
線形回帰ハンズオン
from sklearn.datasets import load_boston
from pandas import DataFrame
from sklearn.linear_model import LinearRegression
import numpy as np
boston = load_boston()
print(boston.feature_names)
df = DataFrame(data=boston.data, columns = boston.feature_names)
df['PRICE'] = np.array(boston.target)
train_X = df.loc[:, ['RM', 'CRIM']].values
train_y = df.loc[:, 'PRICE'].values
model = LinearRegression()
model.fit(train_X, train_y)
# 回帰係数と切片を出力
print(model.coef_)
print(model.intercept_)
print(model.predict([[6, 0.3]]))
2. 非線形回帰モデル
◆複雑な非線形構造を内在する現象に対して、非線形回帰モデリングを実施
・構造を線形で捉えられる場合は限られる
・非線形な構造を捉えられる仕組みが必要
◆基底展開法
・回帰関数として、基底関数と呼ばれる既知の非線形関数とパラメータベクトルの線型結合を使用
・未知パラメータは線形回帰モデルと同様に最小2乗法や最尤法により推定
◆基底関数の例
多項式関数
ϕj(x)=xj
ガウス型基底関数
ϕj(x)=exp((x−μj)^2/2hj)
◆未学習(underfitting)と過学習(overfitting)
・学習データに対して、十分小さな誤差が得られないモデルは未学習
→表現力の高いモデルを利用する
・小さな誤差は得られたけど、テスト集合誤差との差が大きいモデルは過学習
→学習データの数を増やす
→不要な基底関数(変数)を削除して表現力を抑止
→正則化法を利用して表現力を抑止
◆正則化法(罰則化法)
「モデルの複雑さに伴って、その値が大きくなる正則化項(罰則項)を課した関数」を最小化
正則化項(罰則項):形状によっていくつもの種類があり、それぞれ推定量の性質が異なる
正則化(平滑化)パラメータ:モデルの曲線のなめらかさを調節(適切に決める)
Sγ=(y−Φw)^T(y−Φw)+γR(w)
◆正則化項(罰則項)
無い:最小2乗推定量
正則化パラータの役割
小さくする▶制約面が大きく
大きくする▶制約面が小さく
◆ホールドアウト法
有限のデータを学習用とテスト用の2つに分割し、「予測精度」や「誤り率」を推定する為に使用。
手元にデータが大量にある場合を除いて、良い性能評価を与えないという欠点がある。
◆交差検証(クロスバリデーション)
データをいくつかのブロックに分割し、1つのブロックを検証データ、他を学習データとする。
学習データでモデルを学習させ、検証データで各モデルの精度を計測する、ということを分割数繰り返す。
最もCV値が小さいものを採用する。
◆グリッドサーチ
全てのチューニングパラメータの組み合わせで評価値を算出する。
非線形回帰ハンズオン
import numpy as np
import matplotlib.pyplot as plt
import seaborn as sns
%matplotlib inline
sns.set()
sns.set_style("darkgrid", {'grid.linestyle': '--'})
sns.set_context("paper")
n=100
def true_func(x):
z = 1-48*x+218*x**2-315*x**3+145*x**4
return z
def linear_func(x):
z = x
return z
# 真の関数からノイズを伴うデータを生成
# 真の関数からデータ生成
data = np.random.rand(n).astype(np.float32)
data = np.sort(data)
target = true_func(data)
# ノイズを加える
noise = 0.5 * np.random.randn(n)
target = target + noise
# ノイズ付きデータを描画
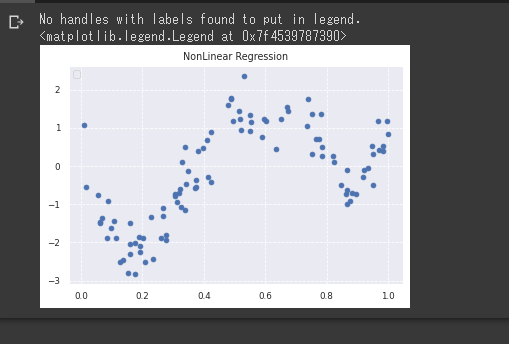
plt.scatter(data, target)
plt.title('NonLinear Regression')
plt.legend(loc=2)
from sklearn.linear_model import LinearRegression
clf = LinearRegression()
data = data.reshape(-1,1)
target = target.reshape(-1,1)
clf.fit(data, target)
p_lin = clf.predict(data)
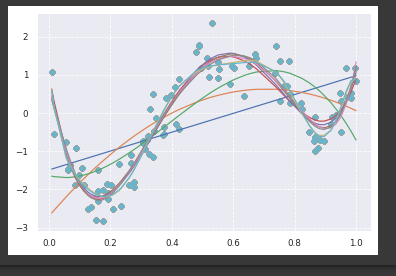
plt.scatter(data, target, label='data')
plt.plot(data, p_lin, color='darkorange', marker='', linestyle='-', linewidth=1, markersize=6, label='linear regression')
plt.legend()
print(clf.score(data, target))
from sklearn.kernel_ridge import KernelRidge
clf = KernelRidge(alpha=0.0002, kernel='rbf')
clf.fit(data, target)
p_kridge = clf.predict(data)
print(clf.score(data, target))
plt.scatter(data, target, color='blue', label='data')
plt.plot(data, p_kridge, color='orange', linestyle='-', linewidth=3, markersize=6, label='kernel ridge')
plt.legend()
# . 多項式を基底関数とした非線形回帰モデル
from sklearn.preprocessing import PolynomialFeatures
from sklearn.pipeline import Pipeline
deg = [1,2,3,4,5,6,7,8,9,10]
for d in deg:
regr = Pipeline([
('poly', PolynomialFeatures(degree=d)),
('linear', LinearRegression())
])
regr.fit(data, target)
# make predictions
p_poly = regr.predict(data)
# plot regression result
plt.scatter(data, target, label='data')
plt.plot(data, p_poly, label='polynomial of degree %d' % (d))
# . サポートベクター回帰(SVR)モデル
from sklearn import model_selection, preprocessing, linear_model, svm
# SVR-rbf
clf_svr = svm.SVR(kernel='rbf', C=1e3, gamma=0.1, epsilon=0.1)
clf_svr.fit(data, target)
y_rbf = clf_svr.fit(data, target).predict(data)
# plot
plt.scatter(data, target, color='darkorange', label='data')
plt.plot(data, y_rbf, color='red', label='Support Vector Regression (RBF)')
plt.legend()
plt.show()
# . Kerasによる深層学習実装
from sklearn.model_selection import train_test_split
x_train, x_test, y_train, y_test = train_test_split(data, target, test_size=0.1, random_state=0)
from keras.callbacks import EarlyStopping, TensorBoard, ModelCheckpoint
cb_cp = ModelCheckpoint('out/checkpoints/weights.{epoch:02d}-{val_loss:.2f}.hdf5', verbose=1, save_weights_only=True)
cb_tf = TensorBoard(log_dir='out/tensorBoard', histogram_freq=0)
def relu_reg_model():
model = Sequential()
model.add(Dense(10, input_dim=1, activation='relu'))
model.add(Dense(1000, activation='relu'))
model.add(Dense(1000, activation='relu'))
model.add(Dense(1000, activation='relu'))
model.add(Dense(1000, activation='relu'))
model.add(Dense(1000, activation='relu'))
model.add(Dense(1000, activation='relu'))
model.add(Dense(1000, activation='relu'))
model.add(Dense(1000, activation='linear'))
# model.add(Dense(100, activation='relu'))
# model.add(Dense(100, activation='relu'))
# model.add(Dense(100, activation='relu'))
# model.add(Dense(100, activation='relu'))
model.add(Dense(1))
model.compile(loss='mean_squared_error', optimizer='adam')
return model
from keras.models import Sequential
from keras.layers import Input, Dense, Dropout, BatchNormalization
from keras.wrappers.scikit_learn import KerasRegressor
# use data split and fit to run the model
estimator = KerasRegressor(build_fn=relu_reg_model, epochs=100, batch_size=5, verbose=1)
history = estimator.fit(x_train, y_train, callbacks=[cb_cp, cb_tf], validation_data=(x_test, y_test))
y_pred = estimator.predict(x_train)
3. ロジスティック回帰モデル
分類問題を解くための教師あり機械学習モデル
・入力とm次元パラメータの線形結合をシグモイド関数に入力
・出力はy=1になる確率の値になる
◆シグモイド関数
・入力は実数・出力は必ず0~1の値
・(クラス1に分類される)確率を表現
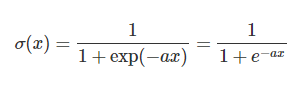
◆最尤推定
・元のデータであるx、yを生成する尤もらしいパラメータを探す推定
・尤度関数を最大化させるパラメータを探す
・ロジスティック回帰モデルではベルヌーイ分布を利用
◆尤度関数
データを固定し、パラメータを変化させる
1回の試行

◆勾配降下法(Gradient descent)
反復学習によりパラメータを逐次的に更新するアプローチの一つ
対数尤度関数をパラメータで微分して0になる値を求める必要があるのだが、解析的にこの値を求めることは困難であるため、勾配降下法を用いる
・F値(F score)
再現率(Recall)と適合率(Precision)の調和平均

ロジスティック回帰ハンズオン
import pandas as pd
from pandas import DataFrame
import numpy as np
import matplotlib.pyplot as plt
import seaborn as sns
%matplotlib inline
titanic_df = pd.read_csv('../data/titanic_train.csv')
titanic_df.head(5)
# 予測に不要と考えるカラムをドロップ (本当はここの情報もしっかり使うべきだと思っています)
titanic_df.drop(['PassengerId', 'Name', 'Ticket', 'Cabin'], axis=1, inplace=True)
# nullを含んでいる行を表示
titanic_df[titanic_df.isnull().any(1)].head(10)
# Ageカラムのnullを中央値で補完
titanic_df['AgeFill'] = titanic_df['Age'].fillna(titanic_df['Age'].mean())
# 再度nullを含んでいる行を表示 (Ageのnullは補完されている)
titanic_df[titanic_df.isnull().any(1)]
# 運賃だけのリストを作成
data1 = titanic_df.loc[:, ["Fare"]].values
# 生死フラグのみのリストを作成
label1 = titanic_df.loc[:,["Survived"]].values
from sklearn.linear_model import LogisticRegression
model=LogisticRegression()
model.fit(data1, label1)
print (model.predict([[61]]))
print (model.predict_proba([[62]]))
print (model.intercept_)
print (model.coef_)
# . ロジスティック回帰の実装(2変数から生死を判別)
# . 性別をparameterとして取り込むため、女性︓female=0、男性︓male=1とした変数を新たに作成する
titanic_df['Gender'] = titanic_df['Sex'].map({'female': 0, 'male': 1}).astype(int)
# . Pclass(乗客の社会階級)+Gender(乗客の性別)を新たにPclass_Genderと定義する。
titanic_df['Pclass_Gender'] = titanic_df['Pclass'] + titanic_df['Gender']
titanic_df = titanic_df.drop(['Pclass', 'Sex', 'Gender','Age'], axis=1)
np.random.seed = 0
xmin, xmax = -5, 85
ymin, ymax = 0.5, 4.5
index_survived = titanic_df[titanic_df["Survived"]==0].index
index_notsurvived = titanic_df[titanic_df["Survived"]==1].index
from matplotlib.colors import ListedColormap
fig, ax = plt.subplots()
cm = plt.cm.RdBu
cm_bright = ListedColormap(['#FF0000', '#0000FF'])
sc = ax.scatter(titanic_df.loc[index_survived, 'AgeFill'],
titanic_df.loc[index_survived, 'Pclass_Gender']+(np.random.rand(len(index_survived))-0.5)*0.1,
color='r', label='Not Survived', alpha=0.3)
sc = ax.scatter(titanic_df.loc[index_notsurvived, 'AgeFill'],
titanic_df.loc[index_notsurvived, 'Pclass_Gender']+(np.random.rand(len(index_notsurvived))-0.5)*0.1,
color='b', label='Survived', alpha=0.3)
ax.set_xlabel('AgeFill')
ax.set_ylabel('Pclass_Gender')
ax.set_xlim(xmin, xmax)
ax.set_ylim(ymin, ymax)
ax.legend(bbox_to_anchor=(1.4, 1.03))
# 運賃だけのリストを作成
data2 = titanic_df.loc[:, ["AgeFill", "Pclass_Gender"]].values
# 生死フラグのみのリストを作成
label2 = titanic_df.loc[:,["Survived"]].values
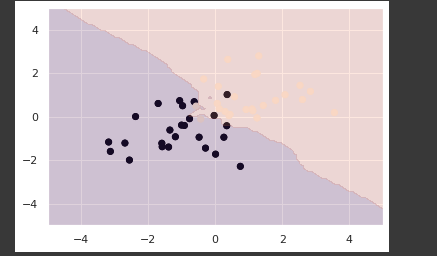
model2 = LogisticRegression()
print(model2.fit(data2, label2))
print(model2.predict([[10,1]]))
print(model2.predict_proba([[10,1]])
h = 0.02
xmin, xmax = -5, 85
ymin, ymax = 0.5, 4.5
xx, yy = np.meshgrid(np.arange(xmin, xmax, h), np.arange(ymin, ymax, h))
Z = model2.predict_proba(np.c_[xx.ravel(), yy.ravel()])[:, 1]
Z = Z.reshape(xx.shape)
fig, ax = plt.subplots()
levels = np.linspace(0, 1.0)
cm = plt.cm.RdBu
cm_bright = ListedColormap(['#FF0000', '#0000FF'])
# contour = ax.contourf(xx, yy, Z, cmap=cm, levels=levels, alpha=0.5)
sc = ax.scatter(titanic_df.loc[index_survived, 'AgeFill'],
titanic_df.loc[index_survived, 'Pclass_Gender']+(np.random.rand(len(index_survived))-0.5)*0.1,
color='r', label='Not Survived', alpha=0.3)
sc = ax.scatter(titanic_df.loc[index_notsurvived, 'AgeFill'],
titanic_df.loc[index_notsurvived, 'Pclass_Gender']+(np.random.rand(len(index_notsurvived))-0.5)*0.1,
color='b', label='Survived', alpha=0.3)
ax.set_xlabel('AgeFill')
ax.set_ylabel('Pclass_Gender')
ax.set_xlim(xmin, xmax)
ax.set_ylim(ymin, ymax)
# fig.colorbar(contour)
x1 = xmin
x2 = xmax
y1 = -1*(model2.intercept_[0]+model2.coef_[0][0]*xmin)/model2.coef_[0][1]
y2 = -1*(model2.intercept_[0]+model2.coef_[0][0]*xmax)/model2.coef_[0][1]
ax.plot([x1, x2] ,[y1, y2], 'k--')
print('------------------------------------------')
print(eval_model1.score(traindata1, trainlabel1))
print(eval_model1.score(testdata1,testlabel1))
print(eval_model2.score(traindata2, trainlabel2))
print(eval_model2.score(testdata2,testlabel2))
print('------------------------------------------')
import seaborn as sns
sns.set(style="whitegrid")
# Load the example Titanic dataset
titanic = sns.load_dataset("titanic")
# Set up a grid to plot survival probability against several variables
g = sns.PairGrid(titanic, y_vars="survived",
x_vars=["class", "sex", "who", "alone"],
size=5, aspect=.5)
# Draw a seaborn pointplot onto each Axes
g.map(sns.pointplot, color=sns.xkcd_rgb["plum"])
g.set(ylim=(0, 1))
sns.despine(fig=g.fig, left=True)
plt.show()
import seaborn as sns
sns.set(style="darkgrid")
# Load the example titanic dataset
df = sns.load_dataset("titanic")
# Make a custom palette with gendered colors
pal = dict(male="#6495ED", female="#F08080")
# Show the survival proability as a function of age and sex
g = sns.lmplot(x="age", y="survived", col="sex", hue="sex", data=df,
palette=pal, y_jitter=.02, logistic=True)
g.set(xlim=(0, 80), ylim=(-.05, 1.05))
plt.show()
4. 主成分分析
多変量データの持つ構造をより少数個の指標に圧縮
・変量の個数を減らすことに伴う情報の損失はなるべく小さく
・少数変数を利用した分析や可視化(2・3次元の場合)が実現可能
第1~元次元分の主成分の分散は、元のデータの分散と一致
2次元のデータを2次元の主成分で表示した時、固有値の和と元のデータの分散が一致
第k主成分の分散は主成分に対応する固有値
寄与率:第k主成分の分散の全分散に対する割合(第k主成分が持つ情報量の割合)
累積寄与率:第1-k主成分まで圧縮した際の情報損失量の割合
主成分分析ハンズオン
import pandas as pd
from sklearn.model_selection import train_test_split
from sklearn.preprocessing import StandardScaler
from sklearn.linear_model import LogisticRegressionCV
from sklearn.metrics import confusion_matrix
from sklearn.decomposition import PCA
import matplotlib.pyplot as plt
%matplotlib inline
cancer_df = pd.read_csv('../data/cancer.csv')
cancer_df.drop('Unnamed: 32', axis=1, inplace=True)
cancer_df
# 目的変数の抽出
y = cancer_df.diagnosis.apply(lambda d: 1 if d == 'M' else 0)
# 説明変数の抽出
X = cancer_df.loc[:, 'radius_mean':]
# 学習用とテスト用でデータを分離
X_train, X_test, y_train, y_test = train_test_split(X, y, random_state=0)
# 標準化
scaler = StandardScaler()
X_train_scaled = scaler.fit_transform(X_train)
X_test_scaled = scaler.transform(X_test)
# ロジスティック回帰で学習
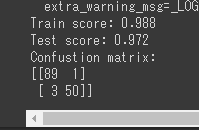
logistic = LogisticRegressionCV(cv=10, random_state=0)
logistic.fit(X_train_scaled, y_train)
# 検証
print('Train score: {:.3f}'.format(logistic.score(X_train_scaled, y_train)))
print('Test score: {:.3f}'.format(logistic.score(X_test_scaled, y_test)))
print('Confustion matrix:\n{}'.format(confusion_matrix(y_true=y_test, y_pred=logistic.predict(X_test_scaled))))
pca = PCA(n_components=30)
pca.fit(X_train_scaled)
plt.bar([n for n in range(1, len(pca.explained_variance_ratio_)+1)], pca.explained_variance_ratio_)
# PCA
# 次元数2まで圧縮
pca = PCA(n_components=2)
X_train_pca = pca.fit_transform(X_train_scaled)
print('X_train_pca shape: {}'.format(X_train_pca.shape))
# X_train_pca shape: (426, 2)
# 寄与率
print('explained variance ratio: {}'.format(pca.explained_variance_ratio_))
# explained variance ratio: [ 0.43315126 0.19586506]
# 散布図にプロット
temp = pd.DataFrame(X_train_pca)
temp['Outcome'] = y_train.values
b = temp[temp['Outcome'] == 0]
m = temp[temp['Outcome'] == 1]
plt.scatter(x=b[0], y=b[1], marker='o') # 良性は○でマーク
plt.scatter(x=m[0], y=m[1], marker='^') # 悪性は△でマーク
plt.xlabel('PC 1') # 第1主成分をx軸
plt.ylabel('PC 2') # 第2主成分をy軸
第1成分の寄与率が0.433、第2成分の寄与率が0.195
5. k近傍法
分類問題のための機械学習手法
新しいデータ点を分類するときに、そのデータ点の最近傍のデータをk個取り、それらが最も多く属するクラスに識別する。
kがハイパーパラメータであり、kを大きくすると決定境界は滑らかになる
k近傍法ハンズオン
%matplotlib inline
import numpy as np
import matplotlib.pyplot as plt
from scipy import stats
def gen_data():
x0 = np.random.normal(size=50).reshape(-1, 2) - 1
x1 = np.random.normal(size=50).reshape(-1, 2) + 1.
x_train = np.concatenate([x0, x1])
y_train = np.concatenate([np.zeros(25), np.ones(25)]).astype(np.int)
return x_train, y_train
X_train, ys_train = gen_data()
plt.scatter(X_train[:, 0], X_train[:, 1], c=ys_train)
※色が見づらくなったが分けられている
# 予測
def distance(x1, x2):
return np.sum((x1 - x2)**2, axis=1)
def knc_predict(n_neighbors, x_train, y_train, X_test):
y_pred = np.empty(len(X_test), dtype=y_train.dtype)
for i, x in enumerate(X_test):
distances = distance(x, X_train)
nearest_index = distances.argsort()[:n_neighbors]
mode, _ = stats.mode(y_train[nearest_index])
y_pred[i] = mode
return y_pred
def plt_resut(x_train, y_train, y_pred):
xx0, xx1 = np.meshgrid(np.linspace(-5, 5, 100), np.linspace(-5, 5, 100))
xx = np.array([xx0, xx1]).reshape(2, -1).T
plt.scatter(x_train[:, 0], x_train[:, 1], c=y_train)
plt.contourf(xx0, xx1, y_pred.reshape(100, 100).astype(dtype=np.float), alpha=0.2, levels=np.linspace(0, 1, 3))
n_neighbors = 3
xx0, xx1 = np.meshgrid(np.linspace(-5, 5, 100), np.linspace(-5, 5, 100))
X_test = np.array([xx0, xx1]).reshape(2, -1).T
y_pred = knc_predict(n_neighbors, X_train, ys_train, X_test)
plt_resut(X_train, ys_train, y_pred)
# numpyによる実装
xx0, xx1 = np.meshgrid(np.linspace(-5, 5, 100), np.linspace(-5, 5, 100))
xx = np.array([xx0, xx1]).reshape(2, -1).T
from sklearn.neighbors import KNeighborsClassifier
knc = KNeighborsClassifier(n_neighbors=n_neighbors).fit(X_train, ys_train)
plt_resut(X_train, ys_train, knc.predict(xx))
6. k-平均法(k-means)
・教師なし学習
・クラスタリング手法
・与えられたデータをk個のクラスタに分類を行う
k平均法(k-means)のアルゴリズム
- 各クラスタ中心の初期値を設定する
- 各クラスタ中心との距離を計算し、最も距離が近いクラスタを割り当てる
- 各クラスタの平均ベクトル(中心)を計算する
- 収束するまで2, 3の処理を繰り返す
中心の初期値を変えるとクラスタリング結果も変わりうる
kの値を変えるとクラスタリング結果も変わる
%matplotlib inline
import numpy as np
import matplotlib.pyplot as plt
# データ生成
def gen_data():
x1 = np.random.normal(size=(100, 2)) + np.array([-5, -5])
x2 = np.random.normal(size=(100, 2)) + np.array([5, -5])
x3 = np.random.normal(size=(100, 2)) + np.array([0, 5])
return np.vstack((x1, x2, x3))
# データ作成
X_train = gen_data()
# データ描画
plt.scatter(X_train[:, 0], X_train[:, 1])
def distance(x1, x2):
return np.sum((x1 - x2)**2, axis=1)
n_clusters = 3
iter_max = 100
# 各クラスタ中心をランダムに初期化
centers = X_train[np.random.choice(len(X_train), n_clusters, replace=False)]
for _ in range(iter_max):
prev_centers = np.copy(centers)
D = np.zeros((len(X_train), n_clusters))
# 各データ点に対して、各クラスタ中心との距離を計算
for i, x in enumerate(X_train):
D[i] = distance(x, centers)
# 各データ点に、最も距離が近いクラスタを割り当
cluster_index = np.argmin(D, axis=1)
# 各クラスタの中心を計算
for k in range(n_clusters):
index_k = cluster_index == k
centers[k] = np.mean(X_train[index_k], axis=0)
# 収束判定
if np.allclose(prev_centers, centers):
break
def plt_result(X_train, centers, xx):
# データを可視化
plt.scatter(X_train[:, 0], X_train[:, 1], c=y_pred, cmap='spring')
# 中心を可視化
plt.scatter(centers[:, 0], centers[:, 1], s=200, marker='X', lw=2, c='black', edgecolor="white")
# 領域の可視化
pred = np.empty(len(xx), dtype=int)
for i, x in enumerate(xx):
d = distance(x, centers)
pred[i] = np.argmin(d)
plt.contourf(xx0, xx1, pred.reshape(100, 100), alpha=0.2, cmap='spring')
y_pred = np.empty(len(X_train), dtype=int)
for i, x in enumerate(X_train):
d = distance(x, centers)
y_pred[i] = np.argmin(d)
xx0, xx1 = np.meshgrid(np.linspace(-10, 10, 100), np.linspace(-10, 10, 100))
xx = np.array([xx0, xx1]).reshape(2, -1).T
plt_result(X_train, centers, xx)
# numpy実装
from sklearn.cluster import KMeans
kmeans = KMeans(n_clusters=3, random_state=0).fit(X_train)
print("labels: {}".format(kmeans.labels_))
print("cluster_centers: {}".format(kmeans.cluster_centers_))
kmeans.cluster_centers_
plt_result(X_train, kmeans.cluster_centers_, xx)
7. サポートベクターマシーン(SVM)
サポートベクターマシン(SVM)とは、教師あり学習において回帰・分類。外れ値の検出を行う方法の一つである。SVMでは例えば2つの分類に対して、データ点をもっとも引き離すような境界線を引くことが目的である。特徴量(説明変数)が2つなら直線、3つなら平面が境界となるが、4つ以上になると超平面で表されるため図示はできない。
サポートベクター(サポートベクトル)
サポートベクトルとは境界線にもっとも近いデータ点のことである。新しいデータが入力された時の誤判定を防ぐため、境界に近いデータであっても境界からできるだけ離すことが重要になる。サポートベクトルと境界との距離をマージンといい、SVMではこのマージンを最大化する。
◆手法
・ソフトマージンSVM
ソフトマージンSVMは、誤差を認めるとより良い線形分離ができるようなデータに用いる。通常のSVM(ハードマージンSVMという)とは異なり、マージンにデータが入ることを許すのが特徴である。ただし、データがマージンに入った場合はペナルティを与え、マージンの最大化とペナルティの最小化を同時に行うことで、できるだけうまく分離するような境界を見つける。データには通常ノイズが含まれるため、実際の機械学習では主にソフトマージンSVMが用いられる。
・カーネル法
カーネル法は、クラスの境界が線形では表せない(曲線を使うしかない)場合に用いる。クラスの境界が曲面場合は、データから新しい特徴量を作って、うまく線形分離可能になるようにプロットする。この作業を、「線形分離可能な高次元特徴空間に写像する」という。データに存在する特徴量から、うまく線形分離可能になるような特徴量を新たに作り出す手法として、カーネル関数を用いる。カーネル関数としてはガウシアン(RBF)カーネルや、多項式カーネルなどがある。カーネル関数を使いつつ計算量を減らす工夫は、カーネルトリックと呼ばれる。
サポートベクターマシーン(SVM)のハンズオン
%matplotlib inline
import numpy as np
import matplotlib.pyplot as plt
# 訓練データ生成
def gen_data():
x0 = np.random.normal(size=50).reshape(-1, 2) - 2.
x1 = np.random.normal(size=50).reshape(-1, 2) + 2.
X_train = np.concatenate([x0, x1])
ys_train = np.concatenate([np.zeros(25), np.ones(25)]).astype(np.int)
return X_train, ys_train
X_train, ys_train = gen_data()
plt.scatter(X_train[:, 0], X_train[:, 1], c=ys_train)
t = np.where(ys_train == 1.0, 1.0, -1.0)
n_samples = len(X_train)
# 線形カーネル
K = X_train.dot(X_train.T)
eta1 = 0.01
eta2 = 0.001
n_iter = 500
H = np.outer(t, t) * K
a = np.ones(n_samples)
for _ in range(n_iter):
grad = 1 - H.dot(a)
a += eta1 * grad
a -= eta2 * a.dot(t) * t
a = np.where(a > 0, a, 0)
index = a > 1e-6
support_vectors = X_train[index]
support_vector_t = t[index]
support_vector_a = a[index]
term2 = K[index][:, index].dot(support_vector_a * support_vector_t)
b = (support_vector_t - term2).mean()
xx0, xx1 = np.meshgrid(np.linspace(-5, 5, 100), np.linspace(-5, 5, 100))
xx = np.array([xx0, xx1]).reshape(2, -1).T
X_test = xx
y_project = np.ones(len(X_test)) * b
for i in range(len(X_test)):
for a, sv_t, sv in zip(support_vector_a, support_vector_t, support_vectors):
y_project[i] += a * sv_t * sv.dot(X_test[i])
y_pred = np.sign(y_project)
# 訓練データを可視化
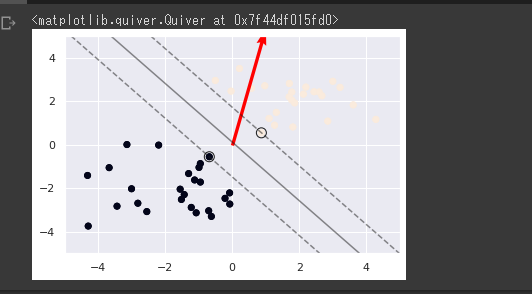
plt.scatter(X_train[:, 0], X_train[:, 1], c=ys_train)
# サポートベクトルを可視化
plt.scatter(support_vectors[:, 0], support_vectors[:, 1],
s=100, facecolors='none', edgecolors='k')
# 領域を可視化
# plt.contourf(xx0, xx1, y_pred.reshape(100, 100), alpha=0.2, levels=np.linspace(0, 1, 3))
# マージンと決定境界を可視化
plt.contour(xx0, xx1, y_project.reshape(100, 100), colors='k',
levels=[-1, 0, 1], alpha=0.5, linestyles=['--', '-', '--'])
# マージンと決定境界を可視化
plt.quiver(0, 0, 0.1, 0.35, width=0.01, scale=1, color='red')
# 訓練データ生成(線形分離不可能)
factor = .2
n_samples = 50
linspace = np.linspace(0, 2 * np.pi, n_samples // 2 + 1)[:-1]
outer_circ_x = np.cos(linspace)
outer_circ_y = np.sin(linspace)
inner_circ_x = outer_circ_x * factor
inner_circ_y = outer_circ_y * factor
X = np.vstack((np.append(outer_circ_x, inner_circ_x),
np.append(outer_circ_y, inner_circ_y))).T
y = np.hstack([np.zeros(n_samples // 2, dtype=np.intp),
np.ones(n_samples // 2, dtype=np.intp)])
X += np.random.normal(scale=0.15, size=X.shape)
x_train = X
y_train = y
plt.scatter(x_train[:,0], x_train[:,1], c=y_train)
def rbf(u, v):
sigma = 0.8
return np.exp(-0.5 * ((u - v)**2).sum() / sigma**2)
X_train = x_train
t = np.where(y_train == 1.0, 1.0, -1.0)
n_samples = len(X_train)
# RBFカーネル
K = np.zeros((n_samples, n_samples))
for i in range(n_samples):
for j in range(n_samples):
K[i, j] = rbf(X_train[i], X_train[j])
eta1 = 0.01
eta2 = 0.001
n_iter = 5000
H = np.outer(t, t) * K
a = np.ones(n_samples)
for _ in range(n_iter):
grad = 1 - H.dot(a)
a += eta1 * grad
a -= eta2 * a.dot(t) * t
a = np.where(a > 0, a, 0)
index = a > 1e-6
support_vectors = X_train[index]
support_vector_t = t[index]
support_vector_a = a[index]
term2 = K[index][:, index].dot(support_vector_a * support_vector_t)
b = (support_vector_t - term2).mean()
xx0, xx1 = np.meshgrid(np.linspace(-1.5, 1.5, 100), np.linspace(-1.5, 1.5, 100))
xx = np.array([xx0, xx1]).reshape(2, -1).T
X_test = xx
y_project = np.ones(len(X_test)) * b
for i in range(len(X_test)):
for a, sv_t, sv in zip(support_vector_a, support_vector_t, support_vectors):
y_project[i] += a * sv_t * rbf(X_test[i], sv)
y_pred = np.sign(y_project)
# 訓練データを可視化
plt.scatter(x_train[:, 0], x_train[:, 1], c=y_train)
# サポートベクトルを可視化
plt.scatter(support_vectors[:, 0], support_vectors[:, 1],
s=100, facecolors='none', edgecolors='k')
# 領域を可視化
plt.contourf(xx0, xx1, y_pred.reshape(100, 100), alpha=0.2, levels=np.linspace(0, 1, 3))
# マージンと決定境界を可視化
plt.contour(xx0, xx1, y_project.reshape(100, 100), colors='k',
levels=[-1, 0, 1], alpha=0.5, linestyles=['--', '-', '--'])
# 訓練データ生成(重なりあり)
x0 = np.random.normal(size=50).reshape(-1, 2) - 1.
x1 = np.random.normal(size=50).reshape(-1, 2) + 1.
x_train = np.concatenate([x0, x1])
y_train = np.concatenate([np.zeros(25), np.ones(25)]).astype(np.int)
plt.scatter(x_train[:, 0], x_train[:, 1], c=y_train)
X_train = x_train
t = np.where(y_train == 1.0, 1.0, -1.0)
n_samples = len(X_train)
# 線形カーネル
K = X_train.dot(X_train.T)
C = 1
eta1 = 0.01
eta2 = 0.001
n_iter = 1000
H = np.outer(t, t) * K
a = np.ones(n_samples)
for _ in range(n_iter):
grad = 1 - H.dot(a)
a += eta1 * grad
a -= eta2 * a.dot(t) * t
a = np.clip(a, 0, C)
index = a > 1e-8
support_vectors = X_train[index]
support_vector_t = t[index]
support_vector_a = a[index]
term2 = K[index][:, index].dot(support_vector_a * support_vector_t)
b = (support_vector_t - term2).mean()
xx0, xx1 = np.meshgrid(np.linspace(-4, 4, 100), np.linspace(-4, 4, 100))
xx = np.array([xx0, xx1]).reshape(2, -1).T
X_test = xx
y_project = np.ones(len(X_test)) * b
for i in range(len(X_test)):
for a, sv_t, sv in zip(support_vector_a, support_vector_t, support_vectors):
y_project[i] += a * sv_t * sv.dot(X_test[i])
y_pred = np.sign(y_project)
# 訓練データを可視化
plt.scatter(x_train[:, 0], x_train[:, 1], c=y_train)
# サポートベクトルを可視化
plt.scatter(support_vectors[:, 0], support_vectors[:, 1],
s=100, facecolors='none', edgecolors='k')
# 領域を可視化
plt.contourf(xx0, xx1, y_pred.reshape(100, 100), alpha=0.2, levels=np.linspace(0, 1, 3))
# マージンと決定境界を可視化
plt.contour(xx0, xx1, y_project.reshape(100, 100), colors='k',
levels=[-1, 0, 1], alpha=0.5, linestyles=['--', '-', '--'])