次世代シーケンサーのデータ解析について理解を深めるために、下記のワークショップに参加中です。
第213回農林交流センターワークショップ 〜次世代シーケンサーのデータ解析技術〜 (2018/9/25-28)
9/25の公開セミナーでナノポアMinIONについての話があったので、雑感を備忘録として。
次世代シーケンサーには第2、第3、第4世代とありますが、ナノポアMinIONは第3世代シーケンサーと呼ばれるものです。
最近,USBメモリほどの大きさのコンパクトで持ち運び可能な一分子シーケンサーとしてOxford Nanopore社からナノポアDNAシーケンサー「MinION」が販売された.非常にコンパクトで場所を問わないことや長いリード長を得られることから,環境調査からベッドサイドまで広い分野での展開が考えられる.
普通のパソコンにUSBでつなげてゲノム解析ができちゃう、使い捨ての次世代シーケンサーです。
以下のリンクを見てもらえるとわかるのですが、めちゃくちゃコンパクトで、ご自宅でも扱えちゃうサイズという、画期的な商品です。
MinION Nanopore DNA Sequencing Technology (IMAGE)
ナノポアMinIONはポテンシャルが半端ない
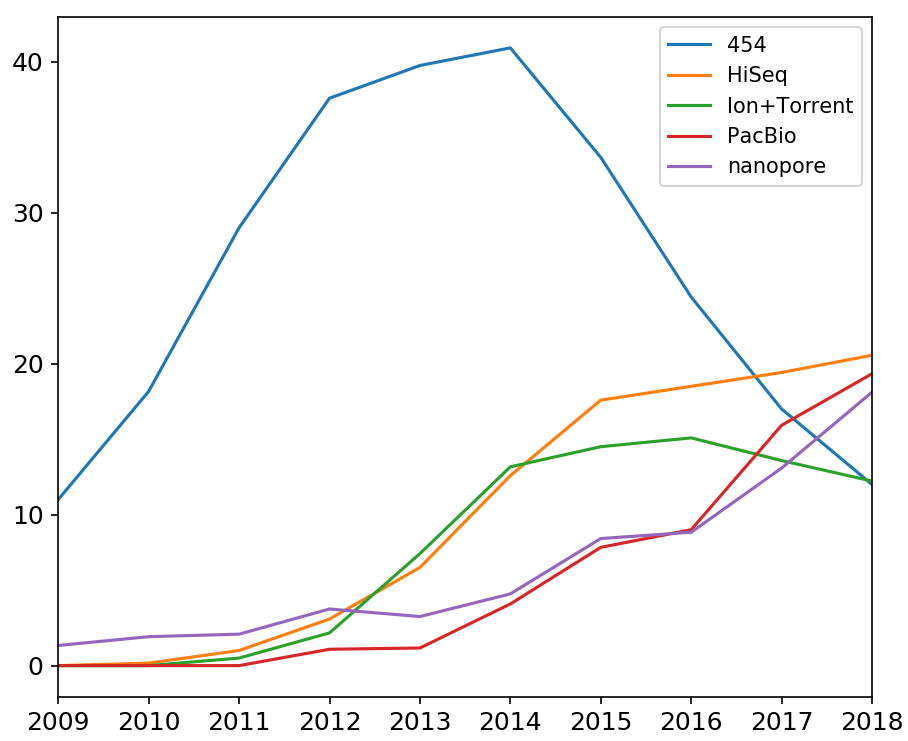
PubMedで調べてみたところ、ナノポアの関連論文数はここ数年増加傾向です。
各次世代シーケンサーをクエリにした際のPubMed検索ヒット数の月平均。454、HiSeq、Ion Torrentは第2世代シーケンサー。PacBioとナノポア(グラフでは"nanopore")は第3世代シーケンサー。詳細は末尾に記載。
ナノポアMinIONは、他の次世代シーケンサーとは異なり、でっかくてお高い機械を買う必要がないです。
なので、途上国の研究機関でも十分に入手可能で、簡易な設備でも使うことができます。
鈴木教授の発表によると、多国籍の研究者同士でそれぞれのデータを片手にガヤガヤ議論するということが普通に行われており、「民主的な側面もある」とのことでした。
また、エボラやマラリア等検体の持ち帰りが難しく、現地で解析までする必要がある場合などに積極的に活用されており、Natureなどのメジャー誌にも掲載されるなど勢いもあります。
-
エボラの調査に使われたNatureの論文
Real-time, portable genome sequencing for Ebola surveillance -
インドネシアでのマラリアの調査に関する論文
Nanopore sequencing of drug-resistance-associated genes in malaria parasites, Plasmodium falciparum
このようなことから、今後も利用は増えていくことが予想されます。
ナノポアMinIONは手軽に使えるようになってきた
この商品、前から知ってはいたのですが、発売当初は届いたうちのほとんどが不良品で役に立たなかったとか、結果が安定しない、などの不満の声が多かったように思います。

しかし、鈴木教授の話によると、最近はこれらの点は改善され、とても安定して解析できるようになっているそうです。

一方で、まだイマイチな点もあります。HiSeqなどの第2世代シーケンサーと比べて、シーケンス時の変異が1割程度入るそうで、これは無視できないくらい高い割合です。
NanoPack: visualizing and processing long-read sequencing data
In contrast, long-read sequencing methods from Oxford Nanopore Technologies (ONT) and Pacific Biosciences routinely achieve read lengths of 10 kb, with a long tail of up to 1.2 Megabases for ONT (unpublished results). These long reads come with a tradeoff of lower accuracy of about 85–95% (Giordano et al., 2017; Jain et al., 2017, 2018).
対照的に、Oxford Nanopore Technologies(ONT)とPacific Biosciencesのロングリードシーケンシング法は、ルーティーンに10kbの読み取り長を達成しており、ONTでは最大1.2 Mbの長いテールを伴っています(未発表データ)。 これらの長い読み取りは、約85〜95%という低い精度のトレードオフを伴います(Giordanoら、2017; Jainら、2017、2018)。
また、ナノポアやPacBioといった第3世代シーケンサーのデータ解析手法は、専門家以外には分かりづらい部分がまだまだ多いです。
これらの点が改善されれば、専門家以外にもとっつきやすくなってさらに爆発的に広まっていきそうですね。
備考
次世代シーケンサーをクエリにした際のPubMed検索ヒット数の月平均を調べたPythonコードは以下の通り。
# Jupyter Labで実行
%matplotlib inline
import matplotlib.pyplot as plt
import requests
import time
from bs4 import BeautifulSoup
seq_sys_list = ["nanopore","PacBio","HiSeq","454","Ion+Torrent"]
url_0 = "https://www.ncbi.nlm.nih.gov/pubmed/"
kwd2 = "sequencing"
result_no_dic_all = {}
for seq_sys in seq_sys_list:
result_no_dic = {}
#2009年から2018年までの論文ヒット数をPubmedより取得
for year in range(2009,2019):
url = url_0 + f"?term=%22{year}%22%5BDate+-+Publication%5D+AND+%22{seq_sys}%22+AND+%22{kwd2}%22"
#PubMedの結果を取得
response = requests.get(url)
soup = BeautifulSoup(response.text, "lxml")
#検索ヒット総数をHTMLより抽出
result_no = int(soup.body.find_all("h3")[4].text.split(" ")[-1])
result_no_dic[year] = result_no
time.sleep(3)
result_no_dic_all[seq_sys] = result_no_dic
print(f"Finish:{seq_sys}")
# 2009年から2017年までは12で割り、2018年のみ9で割ってPandasデータフレーム化
df = pd.DataFrame(result_no_dic_all).T/np.array([12 for i in range(9)]+[9])
df.T.plot(figsize=[7,6])
plt.xticks(fontsize=12)
plt.yticks(fontsize=12)
plt.savefig("20180927_graph.png",dpi=150)
plt.show()