はじめに
この記事ではイチゴピューレ(ピュアなものとイチゴ以外の何かが混合されたものがある)を赤外分光法によって測定したデータを用いてグラフをSeabornで描きます(今回は機械学習とかの話はしません)。
データは983 Mid-infrared (MIR) spectra of fresh fruit pureesからダウンロードできます。なお、このデータの収集とその分析については元論文"Use of Fourier transform infrared spectroscopy and partial least squares regression for the detection of adulteration of strawberry purees" Holland JK, Kemsley EK, Wilson RH. (1998). Journal of the Science of Food and Agriculture, 76, 263-269.に詳しい説明があるようです(読めていません)。
元論文の研究に関連する話
元論文の研究は「バイオインフォマティクス」とか「ケモインフォマティクス」とか「メタボローム解析」と呼ばれている分野に関係するものであり、有名なものとして、ワインの測定データからそのワインの品質を予測する問題・尿の測定データから病変の有無を判別する問題があります。
あまり厳密ではない話なのですが気持ちとしては、「ワインの味は中に入っている物質の種類・量によって決まるので、中に入っている物質を知ることができればワインの味がわかる。」とか「がん患者の体の中での物質の代謝は健康な人のそれとは異なるので、体から出てきた尿に含まれる物質も違ってくる。だから、逆に尿の物質からがん患者なのか健康な人なのかわかるはずだ。」みたいな話です。
少しだけ化学の話
あまり興味のある人はいないかなと思うのですが、測定値の意味を理解するためにざっくり説明します。
今回利用するデータを取得するためにMid-IR Spectroscopyという手法を用いています。これは物質ごとに光の吸収特性の違うことを利用した測定方法です。イチゴピューレの場合、中に入っている物質がピュアなものとピュアでないもので異なるため、結果として光の吸収のされ方が違ってきて測定値に差が出てきます。今回のデータではWavenumbersという列があるのですが、これは「波数」と呼ばれているもので、波長の逆数です。
何をするのか
多くのサンプルについての測定結果があるため、ピュアなものとピュアでないものの平均を取り、その差があるのか直感的にわかるようにグラフ化することを目指します。今回はデータの読み込み~加工・集計、グラフの描画までを行いますが、やっていることはだいたいデータの加工です。以下の手順で進めます。
- データの読み込み~データの加工(元のCSVが処理しづらい形なので良い感じに)
- データの集計(ピュアなものと混合されたものについて各波長の平均値を算出)
- 集計結果をグラフ化
利用するライブラリなど
利用するものは以下の通りです。Windows10 Pro上のAnacondaでJupyterLabを利用して行っていきます。
- Jupyter Lab(Jupyter NotebookでもOK)
- os(ファイルを保管しているところに移動するために利用)
- re(データを加工するために利用)
- Pandas(CSVの読み込み・加工に利用)
- Seaborn(グラフを書くために利用)
実際のデータ処理
今回の処理はJupyterLabで行っていきます。
用意
Anaconda Promptを開いて、
jupyter lab
でJupyterLabをブラウザで開けます。Jupyter Notebookでも良いかなと思うのですが、私はJupyterLabの方が使いやすい気がするので最近はJupyterLabを使っています。以下のようにして新しいNotebookを作ります。
Notebookを作成したら以下のように入力してCtrl+Enterで実行します。
import os
import re
import pandas as pd
import seaborn as sns
まず、必要なライブラリをimportしました。
データの読み込み~データの加工
CSVの読み込み
ダウンロードして適当なフォルダに保存したCSVをPandasで読み込みます。
os.chdir('C:\\Users\\MKX\\Desktop\\MIRFruitPurees')
CSVを保存したフォルダに移動します(今回はデスクトップにフォルダを用意してその中にCSVを保存しています)。
mir_data = pd.read_csv('MIR_Fruit_purees.csv')
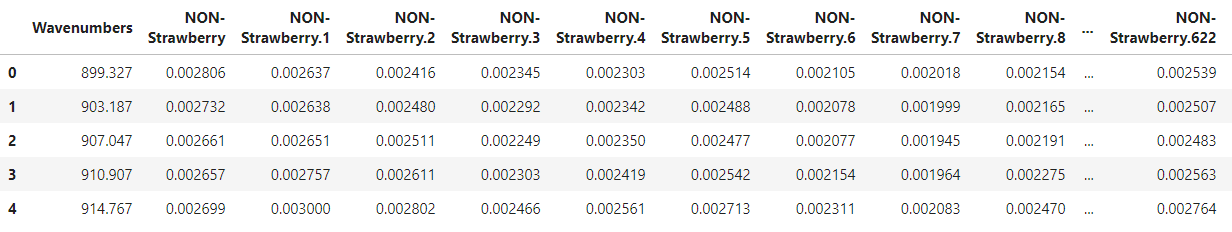
mir_data.head()
CSVを読み込んで先頭5行を表示してみます。
思わず「あ~~~やだなあ」と言ってしまう感じのデータの持ち方をしているので、pd.meltで処理しやすい形に整形しています。
データの整形
以下のようにすることで処理しやすい形に整形することができます。
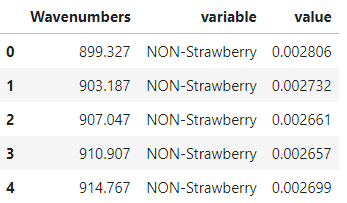
mir_data = pd.melt(mir_data, id_vars=['Wavenumbers'], value_vars = mir_data.columns.values[1:]) # 縦持ちに整形
mir_data.head()
良い感じに変形できました。これで後の処理が楽になります。
一度グラフを描いてみる
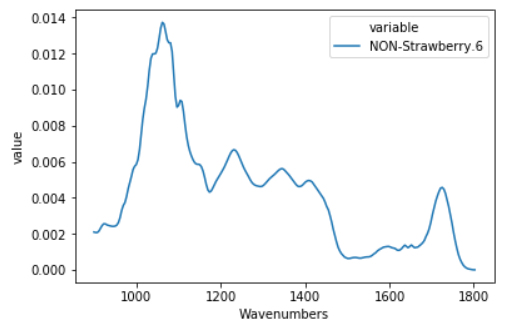
variableの列がNON-Strawberry.6のものについてSeabornでグラフを描いてみます。
sns.lineplot(x='Wavenumbers',y='value', hue='variable', data = mir_data[mir_data['variable']=='NON-Strawberry.6'])
それっぽいグラフができました。
データの集計
ここではピュアなものとピュアでないものについて平均的にどのように違いがあるのかをグラフで表現することを目指します。
集計用の列を追加する
このままでは処理しづらいので集計時に便利なように一列追加します。
mir_data['Strawberry?'] = mir_data['variable'] # 処理しやすくするためにStrawberryとNON-Strawberryを区別するための列を用意
mir_data.tail()
このStrawberry?列のNON-Strawberry.xxx(とStrawberry.xxx)の.xxxを取り除く処理を次に行います。まず関数を定義して、それを列全体に適用するという方法でこれを行います。
まずは関数を定義します。
# NON-Strawberry.10の.10はいらないので処理するための関数
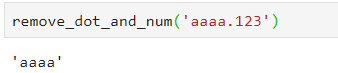
def remove_dot_and_num(x):
return re.sub('.\d+', '', x)
うまくいくのか試してみます。
remove_dot_and_num('aaaa.123')
確かにうまく取り除けました。この関数をStrawberry?の列に対してpd.Series.mapを使って適用します。
mir_data['Strawberry?'] = mir_data['Strawberry?'].map(remove_dot_and_num) # 新しく作ったStrawberry?列全体に関数を適用
mir_data.tail()
平均値の計算
Wavenumbers・Strawberry?ごとに平均値を計算します。
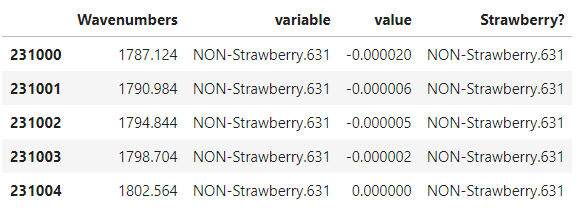
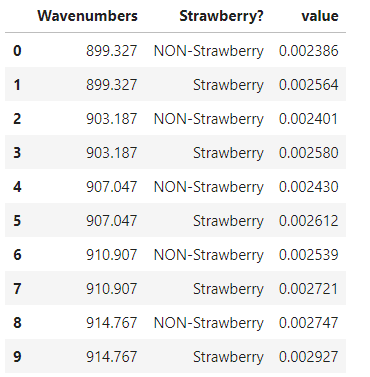
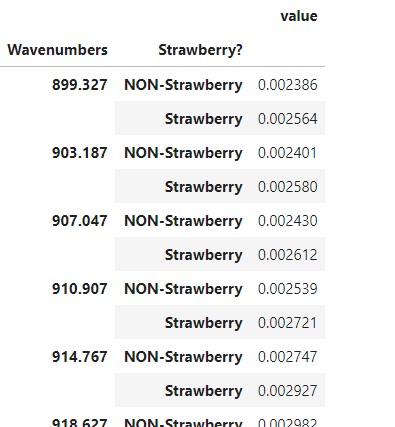
mean_by_wn_straw = mir_data.groupby(['Wavenumbers', 'Strawberry?'],as_index=False).mean() # Wavenumbers(波数)・Strawberry?ごとに平均値を計算
mean_by_wn_straw.head(10)
ここでas_index=Falseにしておかないと以下のようになり、Seabornでうまくグラフ化できないようになってしまうので注意が必要です。
集計結果をグラフ化
グラフを作ります。
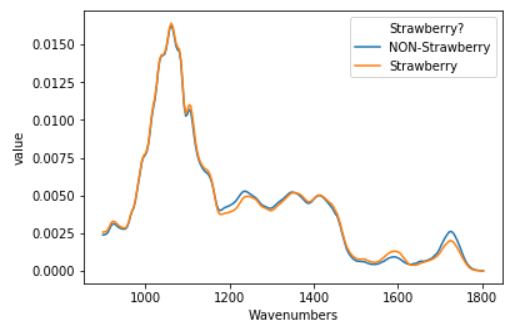
sns.lineplot(data = mean_by_wn_straw, x = 'Wavenumbers', y = 'value', hue = 'Strawberry?')
1200 cm-1から1250 cm-1あたりと1600 cm-1周辺・1750 cm-1周辺に差のある気がします。この波数に注目すればピュアなものとピュアでないものを分けられそうです。この波数に関係する物質がピュアなものとピュアでないもので含まれる量に違いがあるのでしょうか?
感想・今後の展望
データを分析しやすい形に変えるところで疲れてしまったので今回はここまでです。元論文ではPLS回帰でピュアなものとピュアでないイチゴピューレを判別しています。データをガチャガチャして良い感じに判別できないかなど遊んでみようと思います。
参考文献など
参考文献・役に立ちそうな情報をまとめておきます。
データの出典元の論文
"Use of Fourier transform infrared spectroscopy and partial least squares regression for the detection of adulteration of strawberry purees" Holland JK, Kemsley EK, Wilson RH. (1998). Journal of the Science of Food and Agriculture, 76, 263-269.
データ処理・グラフ化の操作について
Python, pandas, melt-ねこゆきのメモ
pd.meltの使い方について参考にしました。
Pandas の groupby の使い方-Qiita
as_index=Falseについて参考にしました。
役に立ちそうな情報
Spectral Features Characterizing Rice Wine “Sake” Variety Using Mid-Infrared Spectroscopy
日本酒を赤外分光法で分析しようという話についてです。
Core Science Resources at QI
いくつかの食品関連のデータセットがあります。今回のデータもここからダウンロードしています。