こんにちは。
前回の記事では、機械学習をテーマに材料研究を紹介しました。
機械学習でなんとなく材料研究者の気分を味わおう
今回は、自分の勉強も兼ねて、オープンソースのpythonライブラリである、pymatgen(Python Materials Genomics)を紹介しながら、手軽に材料研究に触れていただきたいと思っています。
想定読者
- プログラマだけど。物理とか材料とかちょっと興味ある人
- 材料やってるけど、もっと研究を効率化したい人
- 材料やってるけど。プログラムとかかじってみたい人
- materials informatics興味ある人
余談ですが、マテリアルズインフォマティクス若手の会というFacebookグループを作りました。まだ数人しかいませんが、興味がある方は僕(Qiitaアカウントに載せてるfb or メール)までお気軽にご連絡ください〜。materials informaticsに関する情報共有とか、勉強会とかもやりたいと思ってます。
では始めます。
目次
いますぐ材料研究者の気分を味わいたい人は、いきなりインストールに飛んでもらって構いません。
基本は、pymatgenの公式ドキュメントに則ってるので、英語読むのが苦でない方はこっちを読んだようがちゃんと理解できると思います。
では始めます。
材料研究ってそもそも何なの?
前記事からの引用です。読んだ方は飛ばしてください。
→pymatgenとは
まず、材料研究って何やねん、何の材料やねんって感じだと思います。
おっしゃる通り、材料と言ってもセラミックス、高分子、金属など様々なものがあります。
この中には、こんな小さなセラミックコンデンサが数百個入っています。
そして、Appleに選ばれるほどの高性能なコンデンサを作るためには、
・どんな元素を組み合わせよう?
・どんなプロセスで作ろう?
という難しい問題を解くことになります。解き方の一例を示します。
- とりあえず過去の偉人が作ってたやつちょっと変えて作ってみるか
- 意外とできない・・・もっと混ぜ時間増やしたほうがいいのかな(プロセス最適化)
- できた!色々測定したり観察したりしよ(測定)
- なるほど、こんな物理現象で高性能でてるんや(理論、解析)
- 理論と解析で傾向がわかったし、これなら次はこんな元素で作ってみるか・・・(探索)
という感じです。これはあくまで一例ですが、材料研究とは
プロセス最適化、測定、理論、解析、探索などをフル活用してすごい材料を作ること
めちゃくちゃ雑ですが、こんな感じです。
pymatgenとは
簡単に言うと、材料を解析する便利なツールです。
まずは、材料の解析についてざっくり理解するために下の図を見てみてください。
Wikipediaより
高校でもやったと思いますが、物質は原子で構成されていて、それぞれ多種多様な構造を持っています。材料解析の目的は、
どんな種類の原子がどんな割合でどんな構造を持っていたら、どんな性質を持つんだろう?
これを、物理を使って解決することです。
そこで本題のpymatgenですが、
①材料の可視化や解析データの利用に便利なツールがたくさん
②現在材料の解析に使われているツールとの連携が簡単
という強みを持ったオープンソースのpythonライブラリです。GUIで動かす解析ソフトではないのでご注意を。
具体的な機能として、
- 材料の中に含まれる元素の種類や位置、構造を、pythonのクラスで柔軟に表現
- 材料研究でよく使われる様々なデータフォーマットに対応
- 相図や電位-pH図を出力したり、化学反応や拡散の解析などに有用な分析ツールが豊富
- バンド構造や、状態密度などの電子構造解析が可能
- Materials Project REST APIとの連携が可能
が公式ドキュメントで挙げられています。
ここから、実際にpymatgenを使うことで材料研究者の気分を味わってみましょう。
基本機能
classによる原子や構造の表現
参考:http://pymatgen.org/pymatgen.core.html#module-pymatgen.core
まずはpymatgenで提供されている原子や構造の表現に用いるmoduleを見ていきます。
pymatgen.core.periodic_table module
periodic tableとは、みなさんご存知の周期表です。
このモジュールのElement class, Specie classを紹介します。
Element classでは、Enumクラスを継承して、周期表に対応して原子を定義できます。
class Element(Enum):
def __init__(self, symbol):
self.symbol = "%s" % symbol
d = _pt_data[symbol]
...
moduleのインポート時にpymatgenライブラリにあるperiodic_table.jsonを_pt_dataに読み込んであるので、
>>> fe = Element("Fe")
>>> fe.data
{'Superconduction temperature': 'no data K', 'Molar volume': '7.09 cm<sup>3</sup>', 'Ionic radii hs': {'2': 0.92, '3': 0.785}, 'Melting point': '1811 K', 'Atomic radius': 1.4, 'Mineral hardness': '4.0', 'Electrical resistivity': '10 10<sup>-8</sup> Ω m', 'Vickers hardness': '608 MN m<sup>-2</sup>', 'Brinell hardness': '490 MN m<sup>-2</sup>', 'Youngs modulus': '211 GPa', 'Ionic radii': {'2': 0.92, '3': 0.785}, 'Atomic no': 26, 'Mendeleev no': 61, 'Thermal conductivity': '80 W m<sup>-1</sup> K<sup>-1</sup>', 'Reflectivity': '65 %', 'Liquid range': '1323 K', 'Ionic radii ls': {'2': 0.75, '6': 0.39, '3': 0.69, '4': 0.725}, 'Rigidity modulus': '82 GPa', 'X': 1.83, 'Critical temperature': 'no data K', 'Poissons ratio': '0.29', 'Oxidation states': [-2, -1, 1, 2, 3, 4, 5, 6], 'Van der waals radius': 'no data', 'Velocity of sound': '4910 m s<sup>-1</sup>', 'Coefficient of linear thermal expansion': '11.8 x10<sup>-6</sup>K<sup>-1</sup>', 'Bulk modulus': '170 GPa', 'Common oxidation states': [2, 3], 'Name': 'Iron', 'Atomic mass': 55.845, 'Electronic structure': '[Ar].3d<sup>6</sup>.4s<sup>2</sup>', 'Density of solid': '7874 kg m<sup>-3</sup>', 'Refractive index': 'no data', 'Atomic radius calculated': 1.56, 'Boiling point': '3134 K'}
こんなに簡単にその原子のイオン半径、融点、抵抗率、質量、電子構造などなど、いろんな情報を持つオブジェクトが作れます。
それぞれアクセスしたい時は、以下のように属性を指定して取得してください。属性一覧もソースとかドキュメントとかみればわかります。
ionic_radii_fe = fe.ionic_radii
次に、 Specie classを見ていきます。
Specie classでは、酸化数を考慮して原子を表現できます。
supported_properties = ("spin",)
class Specie(symbol, oxidation_state, properties=None):
def __init__(self, symbol, oxidation_state, properties=None):
self._el = Element(symbol)
self._oxi_state = oxidation_state
self._properties = properties if properties else {}
for k in self._properties.keys():
if k not in Specie.supported_properties:
raise ValueError("{} is not a supported property".format(k))
元素に酸化数と特性を持たせることができます。単純に、Elementの拡張版だと考えてもらって良いと思います。Specieオブジェクトには、理想的な酸化数や特性を持たせることを推奨されており、後述するSiteオブジェクトでは、結晶構造中の元素の酸化状態やスピンの状態を表現できるので、シミュレーション結果を保存するためにはSiteオブジェクトを使います。
pymatgen.core.composition
http://pymatgen.org/pymatgen.core.composition.html#module-pymatgen.core.composition
このモジュールでは、H2OやNaClといった物質の組成(composition)を表現するモジュールです。もっともよく使われているComposition classクラスを紹介します。
class Composition(collections.Hashable, collections.Mapping, MSONable):
def __init__(self, *args, **kwargs):
self.allow_negative = kwargs.pop('allow_negative', False)
# it's much faster to recognize a composition and use the elmap than
# to pass the composition to dict()
if len(args) == 1 and isinstance(args[0], Composition):
elmap = args[0]
elif len(args) == 1 and isinstance(args[0], six.string_types):
elmap = self._parse_formula(args[0])
else:
elmap = dict(*args, **kwargs)
elamt = {}
self._natoms = 0
for k, v in elmap.items():
...
パッと見てもわかりにくいし説明大変なので、使い方だけ見ましょう。。。
>>> # NaClやH2Oのような文字列で簡単に定義できます
>>> comp = Composition("LiFePO4")
>>> # 原子数のカウント
>>> comp.num_atoms
7.0
>>> # それぞれの原子の数
>>> comp.formula
'Li1 Fe1 P1 O4'
>>> # 組成比(原子数/全原子数)
>>> comp.get_atomic_fraction(Element("Li"))
0.14285714285714285
などなど、定義も簡単で、便利な組成オブジェクトが作れます。他にもたくさん機能があるので、是非ドキュメントを参考にしてみてください。
pymatgen.core.lattice
http://pymatgen.org/pymatgen.core.lattice.html#module-pymatgen.core.lattice
laticeとは、格子という意味で、高校でも習う単位格子など覚えている人も少なくないのではないでしょうか。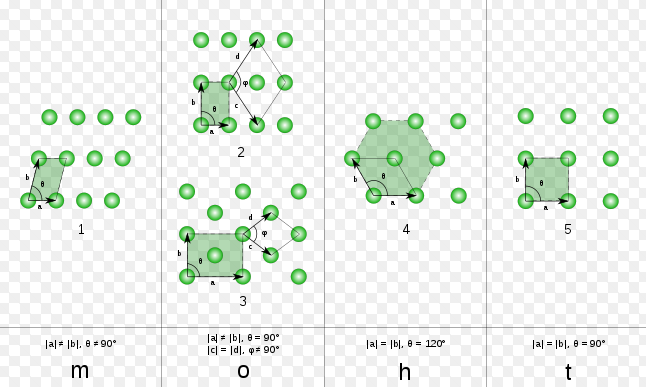
wikipediaより
ここで定義されているベクトル
R = {n}_1{a}_1+{n}_2{a}_2+{n}_3{a}_3
で、格子ベクトルが表現されます。この3次元のベクトルを、次のような多次元配列で定義します。
R = [[10,0,0], [20,10,0], [0,0,30]]
pymatgenでは、lattice classを用いるともっと便利になります。
class Lattice(MSONable):
def __init__(self, matrix):
m = np.array(matrix, dtype=np.float64).reshape((3, 3))
lengths = np.sqrt(np.sum(m ** 2, axis=1))
angles = np.zeros(3)
for i in range(3):
j = (i + 1) % 3
k = (i + 2) % 3
angles[i] = abs_cap(dot(m[j], m[k]) / (lengths[j] * lengths[k]))
self._angles = np.arccos(angles) * 180. / pi
self._lengths = lengths
self._matrix = m
self._inv_matrix = None
self._metric_tensor = None
self._diags = None
self._lll_matrix_mappings = {}
self._lll_inverse = None
self.is_orthogonal = all([abs(a - 90) < 1e-5 for a in self._angles])
...
引数のmatrixは、以下の形式もしくはnumpy arrayに対応しています。
# 多次元リスト
[[1, 0, 0], [0, 1, 0], [0, 0, 1]]
# リスト
[1, 0, 0 , 0, 1, 0, 0, 0, 1]
# タプル
(1, 0, 0, 0, 1, 0, 0, 0, 1)
上は、単純な立方晶(立方体)です。
>>> l = Lattice([1,0,0,0,1,0,0,0,1])
>>> l._angles
array([ 90., 90., 90.])
>>> l.is_orthogonal
True
>>> l._lengths
array([ 1., 1., 1.])
>>> l._matrix
array([[ 1., 0., 0.],
[ 0., 1., 0.],
[ 0., 0., 1.]])
このように、Latticeオブジェクトを使うと角度や長さなどにアクセスできます。その他にも便利な機能を持っているので、ドキュメントを読むことをお勧めします。
http://pymatgen.org/pymatgen.core.lattice.html
pymatgen.core.structure
http://pymatgen.org/pymatgen.core.structure.html
このモジュールでは、結晶構造の表現を可能にする機能を提供しています。
ここでは、もっとも基本的な機能を提供しているIStructure classを見ていきます。
class IStructure(SiteCollection, MSONable):
def __init__(self, lattice, species, coords, validate_proximity=False,
to_unit_cell=False, coords_are_cartesian=False,
site_properties=None):
...
いろいろと引数をとっているので、それぞれ見ていきましょう。
- lattice
pymatgen.core.lattice.Latticeクラスも使うことができます。
- species
これは、原子の種類です。以下のように様々な形式に対応しています。
# 原子の羅列
["Li", "Fe2+", "P", ...]
# 原子番号
(3, 56, ...)
# 占有率も含めたリスト
[{"Fe" : 0.5, "Mn":0.5}, ...]
- coords
これは、各原子の座標を指定します。
# NaClの時
coords = [[0, 0, 0], [0.5, 0.5, 0.5]]
これらを踏まえて、Structureはこのように定義できます。
from pymatgen import Lattice, IStructure
# CsCl構造
a = 4.209 #Å
latt = Lattice.cubic(a)
structure = IStructure(latt, ["Cs", "Cl"], [[0, 0, 0], [0.5, 0.5, 0.5]])
>>> structure.density
3.7492744897576538
>>> structure.distance_matrix
array([[ 0. , 3.64510092],
[ 3.64510092, 0. ]])
>>> structure.get_distance
<bound method IStructure.get_distance of Structure Summary
Lattice
abc : 4.2089999999999996 4.2089999999999996 4.2089999999999996
angles : 90.0 90.0 90.0
volume : 74.565301328999979
A : 4.2089999999999996 0.0 0.0
B : 0.0 4.2089999999999996 0.0
C : 0.0 0.0 4.2089999999999996
PeriodicSite: Cs (0.0000, 0.0000, 0.0000) [0.0000, 0.0000, 0.0000]
PeriodicSite: Cl (2.1045, 2.1045, 2.1045) [0.5000, 0.5000, 0.5000]>
このように、構造中の距離や位置関係、密度などにアクセスできるようになります。
では、次に解析機能はどんなものがあるのかを見ていきましょう。
解析用modules一覧
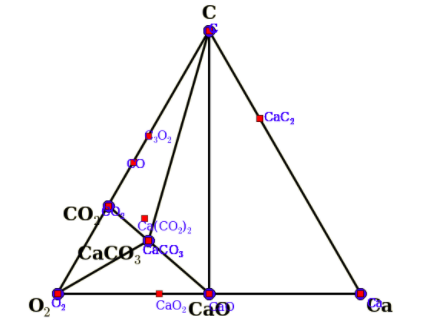
どんなモジュールがあるかだけ軽く紹介します。この中からいくつかピックアップして、実際に動かしてみます。この画像の右側が、pymatgenが提供している解析機能です。
- 相図の出力
- 反応計算
- 電子構造解析と可視化
- バッテリー特性などのアプリケーション特性解析
- 構造可視化
などなど。。。
それぞれ細かくソースから機能紹介する記事も、需要がある&気が向いたら書こうと思っていますので、要望がありましたらコメントとかください。
他ツールとの連携
基本的に、計算量の大きいシミュレーションなどはその他のソフトを用いますが、それらのデータを組み合わせた解析や、可視化の際にpymatgenを使うと効率が増します。
この画像の左側に、よく使われるデータ形式やツールとの連携が書かれてます。
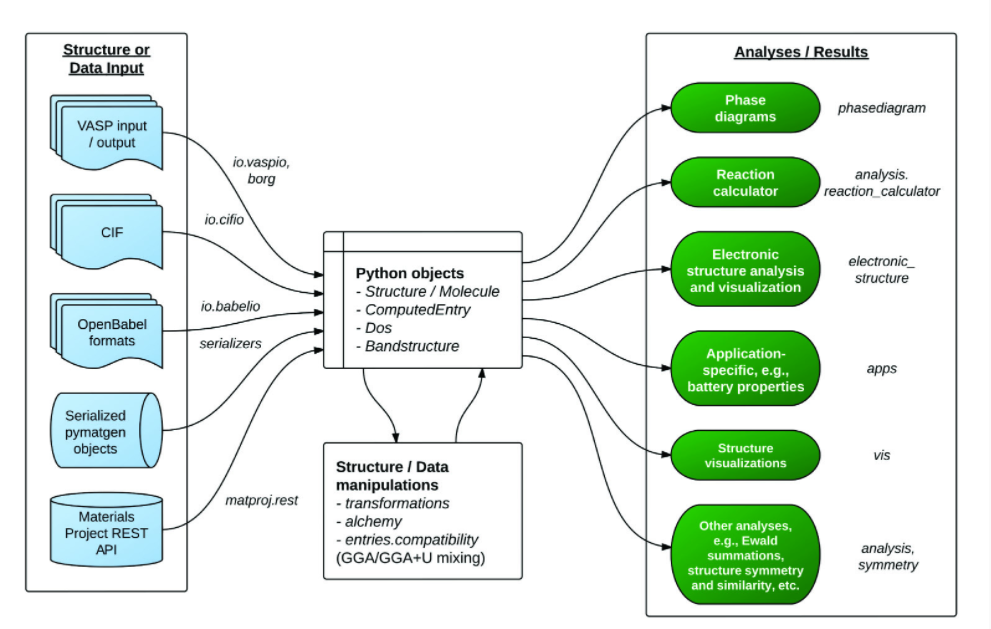
- VASPのinputやoutputの取り込みが可能
- MaterialStudioなどで使われるcifファイルも取扱が可能
- open babelフォーマットに対応
- Materials Project rest apiと連携可能
などが挙げられます。
今、第一原理計算などで上記のファイルやソフトを使っている人なんかは是非導入してみてください。
Materials Project rest apiに関しては、使い方を前回の記事にも書いたので是非参考にしてみてください。これを使えば、Materials Projectというところが大量のデータを公開してくれてて、自由にガンガンデータを取れちゃうので、機械学習とかやりたかったら必須です。
長くなりましたが、みんなでpymatgenを使っていきましょう!
pymatgenのインストール
インストールの基本的な流れは、
- pythonが使える環境(3系がおすすめ)
- condaをインストール
- pymatgenをcondaでインストール
です。condaのインストールまでは、下記の記事などを参考にしてください。
データサイエンティストを目指す人のpython環境構築 2016
そこまで終わったら、
conda install --channel matsci pymatgen
動作確認が終わったら、インストール完了です。
>>> import pymatgen
>>> pymatgen.__version__
'4.5.4'
これで準備も整ったので、実際に試してみましょう!
実際に動かしてみよう
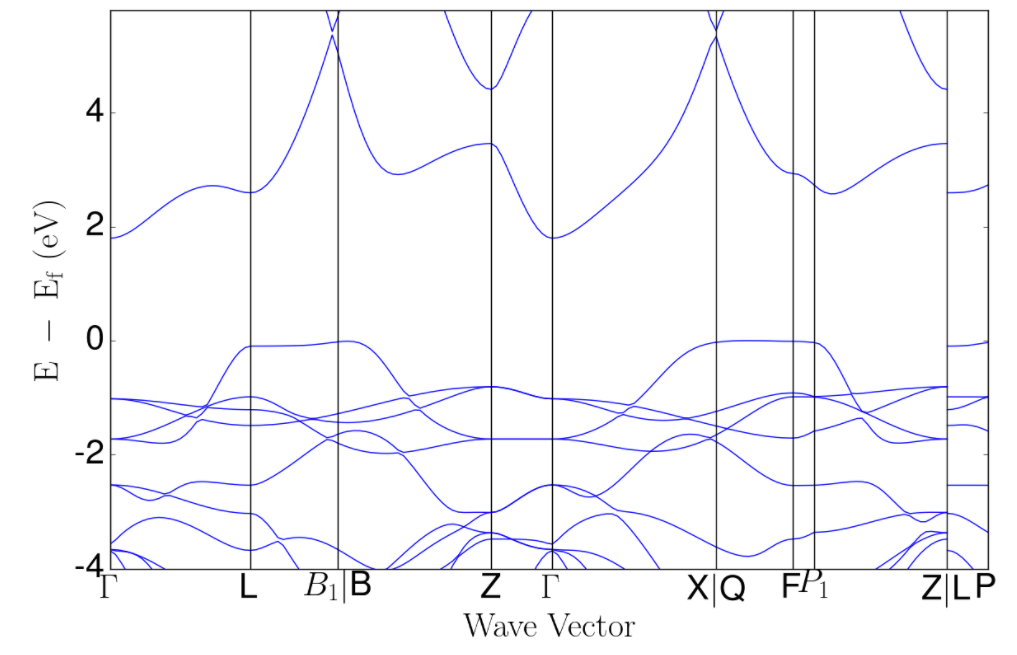
バンド構造解析
バンド構造とは、結晶の周期構造の中で電子などの分散を表します。
バンド構造を図示する際縦軸はエネルギーなのですが、横軸は逆格子空間の点になっていてかなり理解しづらいと思います。なので興味がない方は、"電子がどう分散しているのかがわかる"程度にご理解ください。
pymatgenでは、解析済みのバンド構造を可視化したり、データとして処理することできます。
まずは、必要なライブラリをインポートします。
# materials projectのREST APIを用いるためのmodule
from pymatgen.matproj.rest import MPRester
# バンド構造のプロット用
%matplotlib inline
from pymatgen.electronic_structure.plotter import BSPlotter
次に、解析済みのバンド構造を入手します。本当の研究だと、自前の解析ソフト(VASPなど)で解析済みのファイルをpymatgenのオブジェクトに変換しますが、今回は、解析済みオブジェクトをMaterials Project databaseからダウンロードします。
materials projectを使用する際、登録とAPI keyの取得が必要ですので、公式ページでAPI keyを取得してください。
# 自分のAPIkeyを指定
a = MPRester("自分のAPIkey")
# 欲しい材料のidを指定して、http通信で取得。CuAlO2(mp-3784を取得します)
bs = a.get_bandstructure_by_material_id("mp-3748")
これで、バンド構造の取得が完了しました!materials projectから直接BandStructureオブジェクトが取れました。
http://pymatgen.org/_modules/pymatgen/electronic_structure/bandstructure.html#BandStructure
ちなみに、materials project上ではこのように材料の情報を取得できます。
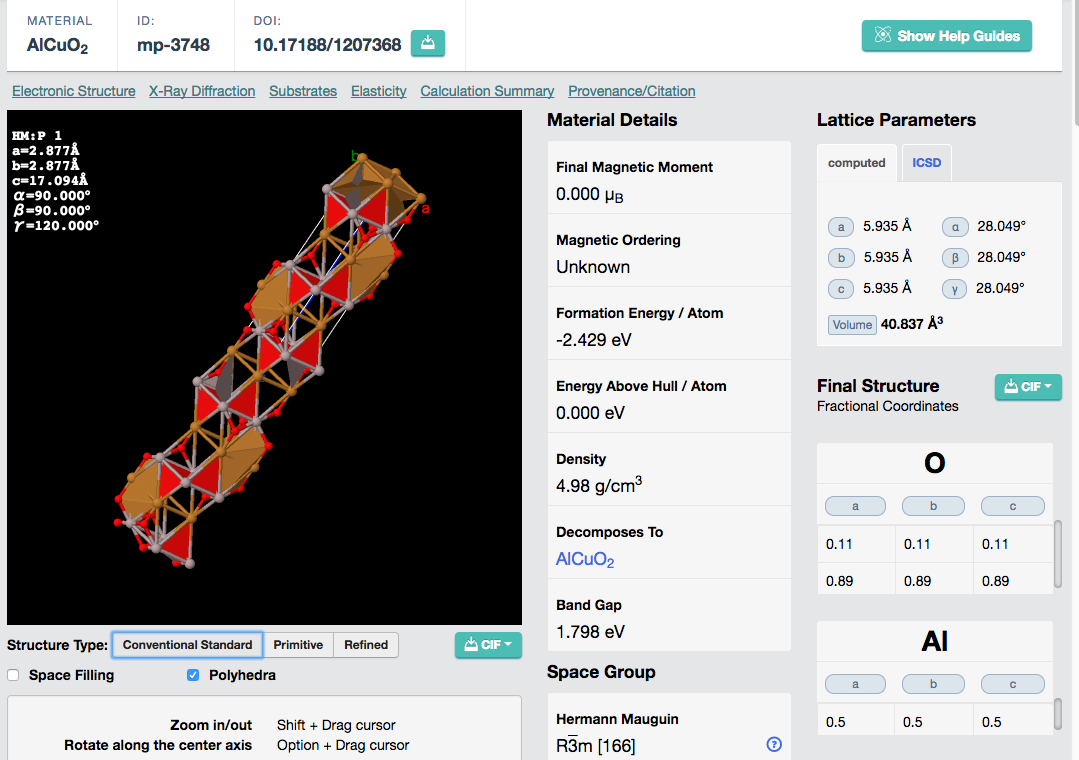
これらの情報を、python上で処理していきます。
>>> bs.is_metal()
False
金属ではないみたいですね。
>>> bs.get_band_gap()
{'energy': 1.7978000000000005, 'direct': False, 'transition': '(0.591,0.409,0.000)-\\Gamma'}
バンドギャップは1.797eV
>>> bs.get_direct_band_gap()
18.0201
直接遷移のバンドギャップは18.021eV。
では、バンド図をプロットしてみましょう。
%matplotlib inline
from pymatgen.electronic_structure.plotter import BSPlotter
plotter = BSPlotter(bs)
plotter.get_plot().show()
これは、materials project上のデータですが、普段は研究している物質などのシミュレーション結果などを出力できます。
ということで、他の材料との比較や機械学習、探索などにおいてpymatgenのオブジェクトが有用そうなのがだんだん伝わっていると嬉しいです。
まだまだできることは多いですが、今回はここで終わりにしておきます。(長かった・・・)
pymatgen勉強会とかmaterials project勉強会とかvasp勉強会とかマテリアルズインフォマティクス若手の会で開きたいと思ってるので、興味ある方は是非入ってみてくださいね〜
また次回もマテリアルズインフォマティクス系で記事書こうと思ってます。ご精読ありがとうございました〜