はじめに
ゲノム編集周辺のバイオインフォマティクス 、特にソフトウェアについての情報をまとめています。
(収録ソフトウェア総数165件)
2022/11追記
さらにツールを増量したバージョンはこちらです!!!
↓↓↓
↑↑↑
配列デザイン
CRISPRによるノックアウト・ノックイン設計・活性予測
概要:
CRISPRのガイド設計では、ターゲットとした配列領域と結合するガイドRNA配列を決定する必要があります。ガイドRNA設計において大事なことは、狙った領域のみに作用させる、という点です。
CRISPRはガイドRNAと1-4塩基ほどのミスマッチを起こす領域であっても誤切断が起こってしまうことがあるので、こうしたオフターゲットが起こらない領域を選ぶ必要があります。また切断の活性もガイドRNAの配列に依存している部分があり、これは目視での鑑別は困難です。さらに大きな欠失を導入する場合には二種類のガイドRNAを使う手法もよく報告されています。
こうした設計における煩雑さを解消するために、下記のようなガイドRNAに対する様々な自動設計・判定ツールが開発されています。
ウェブツール(53件)
:sgRNA設計に加えて、オフターゲット解析や発現ベクター構築に必要なプライマー情報など実験現場に即した情報も提供してくれる。
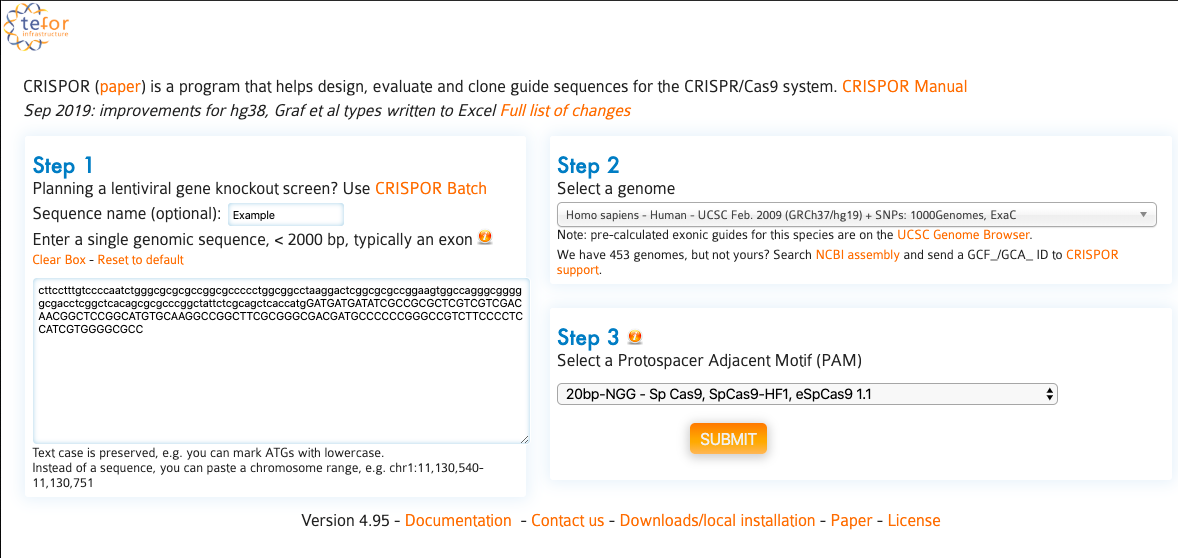
詳細な機能(クリックで展開)
| 対応ゲノム種 | 対応PAM種 | 検索可能な配列長 | 染色体領域指定検索 | オンターゲット予測 | フレームシフト予測 | オフターゲットスコア予測 | N bpミスマッチ領域検索 |
|---|---|---|---|---|---|---|---|
| ヒトを含む526 | 32 | <2,000 | ✅ | ✅ | ✅ | ✅ | ✅ |
| (Doench et al., 2016.)モデル | (Bae et al., 2014)モデル | (Hsu et al. 2013.)モデル | N<5 bp | ||||
| (Moreno-Mateos et al.)モデル | (Chen et al, 2019.)モデル | (Doench 2016 et al., 2016)モデル |
| その他機能 |
|---|
| sgRNA発現ベクター構築用プライマーの自動設計 |
| オフターゲット検証用プライマーの自動設計 |
| オフターゲット領域情報 |
| 検索フィルター機能 |
| アンプリコンシーケンシング解析コマンド表示(CRISPRessoPooled) |
2 CHOPCHOP(提供元:Valenグループ, ベルゲン大学, ノルウェー🇳🇴)
:インタラクティブなゲノム編集実験デザインサイト。ノックアウトだけでなくノックインやノックダウン実験、またTALENでのノックアウトにも対応し、PAMの指定等も自在となっているところからあらゆるゲノム編集ツールで利用できる。

詳細な機能(クリックで展開)
| 対応ゲノム種 | 対応PAM種 | 検索可能な配列長 | 染色体領域指定検索 | オンターゲット予測 | フレームシフト予測 | オフターゲットスコア予測 | N bpミスマッチ領域検索 |
|---|---|---|---|---|---|---|---|
| ヒトを含む252 | 任意入力可能 | 指定遺伝子座全領域 | ✅ | ✅ | ✅ | ❌ | ✅ |
| (Doench et al. 2014)モデル - ※NGG PAM限定 | (Shen et al. 2018)モデル | N<4 bp | |||||
| (Doench et al. 2016)モデル - ※NGG PAM限定 | |||||||
| (Chari et al. 2015)モデル - ※NGG and NNAGAAW PAM's in hg19 and mm10限定 | |||||||
| (Xu et al. 2015)モデル - ※NGG PAM限定 | |||||||
| (Moreno-Mateos et al. 2015)モデル - ※NGG PAM限定 | |||||||
| G20 |
| その他機能 |
|---|
| 設計sgRNAのプロトスペーサー長指定(14-30) |
| sgRNA5'側のヌクレオチド指定(GN(for the U6 promoter) もしくは GG(for T7 polymerase)) |
| sgRNAのSelf-complementarityチェック (Thyme et al.) |
| nCas9、Cpf1、Cas13、TALENの設計 |
| シード領域を考慮したオフターゲット検索 |
| ノックアウト、ノックダウン、ノックイン、activation、repression、大規模ターゲティング(Nanopore enrichmentモード)の設計 |
| 切断確認アッセイ(T7E1)用プライマー設計 |
| (Shen et al. 2018)モデルによるインデル傾向の予測 |
| genome browserによるインタラクティブな標的選択 |
3 E-CRISP(提供元:ドイツがん研究センター, ドイツ🇩🇪)
:オフターゲット検索の厳しさのレベルを3段階で選べたりと手軽に使えるようになっている。その一方でオプションでは細かく検索条件を指定できるようにもなっており、幅広い層にとって有用な構成。

詳細な機能(クリックで展開)
| 対応ゲノム種 | 対応PAM種 | 検索可能な配列長 | 染色体領域指定検索 | オンターゲット予測 | フレームシフト予測 | オフターゲットスコア予測 | N bpミスマッチ領域検索 |
|---|---|---|---|---|---|---|---|
| ヒトを含む55 | 任意入力可能 | 不明 | ✅ | ✅ | ❌ | ✅ | ✅ |
| (Doench et al., 2014.)モデル | 独自ルール(S-score) | N<21 bp | |||||
| (Xu et al. 2015)モデル | |||||||
| 独自ルール(E-score) |
| その他機能 |
|---|
| マッピングツールBowtie/Bowtie2を使ったオフターゲット検索 |
| gRNAペアの設計 |
| 設計sgRNAのプロトスペーサー長指定(7-30) |
| N末タグノックイン、C末タグノックイン、CRISPRi、CRISPRaの設計 |
| 厳密さを3段階に分けたオフターゲット検索オプション |
| タグノックイン、CRISPRi、CRISPRaの範囲指定 |
| エクソンの指定 |
| sgRNA5'側の塩基指定 |
| polyNモチーフをもつsgRNAの除外 |
| プロトスペーサ内の塩基含有率の指定 |
| ゲノムのアノテーションに応じた検索領域指定(CpG islands等) |
| 標的5'側のミスマッチ数の指定 |
| 切断領域と編集領域の距離指定 |
| 外来配列(GFPやgRNA scaffold)に対するオフターゲット検索 |
| 出力図へのアノテーション指定 |
| genome browserによる領域可視化 |
4 GUIDES(提供元:Sanjana研, ニューヨークゲノムセンター & ニューヨーク大学, アメリカ🇺🇸)
:sgRNA設計サイト。複数種の遺伝子座の名前さえ入力すれば一括でsgRNA設計可能な領域を検索することができる。
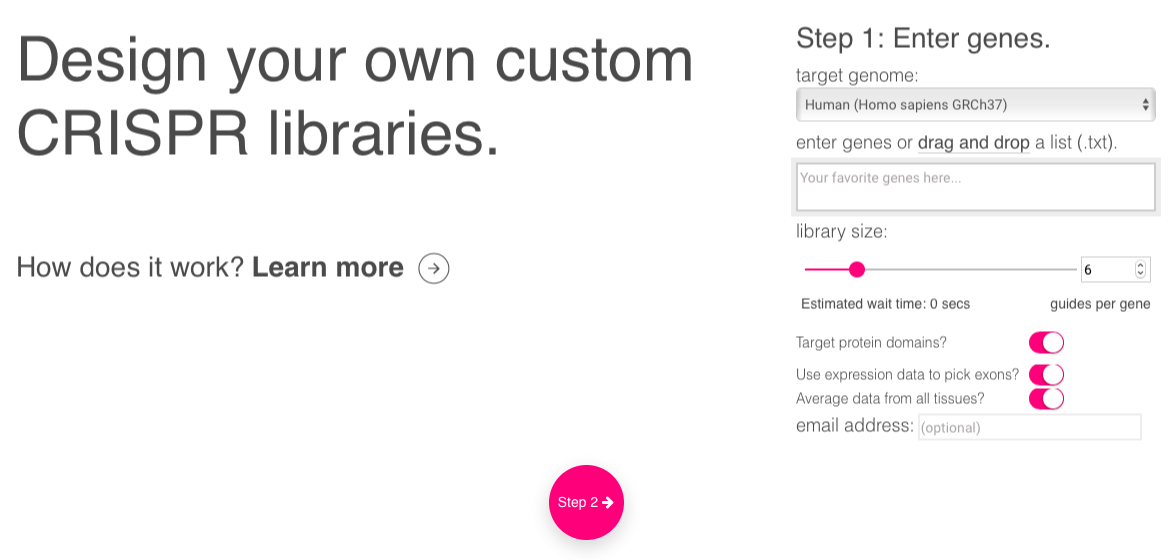
詳細な機能(クリックで展開)
| 対応ゲノム種 | 対応PAM種 | 検索可能な配列長 | 染色体領域指定検索 | オンターゲット予測 | フレームシフト予測 | オフターゲットスコア予測 | N bpミスマッチ領域検索 |
|---|---|---|---|---|---|---|---|
| ヒトを含む2 | NGG | 不明 | ❌ | ✅ | ❌ | ✅ | ❌ |
| (Doench et al., 2016.)モデル | 独自ルール(G-score) |
| その他機能 |
|---|
| 遺伝子座ごとの設計sgRNA数を設定 |
| タンパク領域に限定した設計 |
| 指定したエクソン発現データを表示 |
| 特定の組織に限定した発現データを取得 |
| 設計する遺伝子座名の入力のみで検索が可能 |
5 GuideScan(提供元:Leslie研 & Ventura研, Memorial Sloan Kettering Cancer Center, アメリカ🇺🇸)
:sgRNA設計サイト。染色体領域を指定すれば、その領域全体にわたってsgRNAを設計してくれる。Cpf1にも対応。
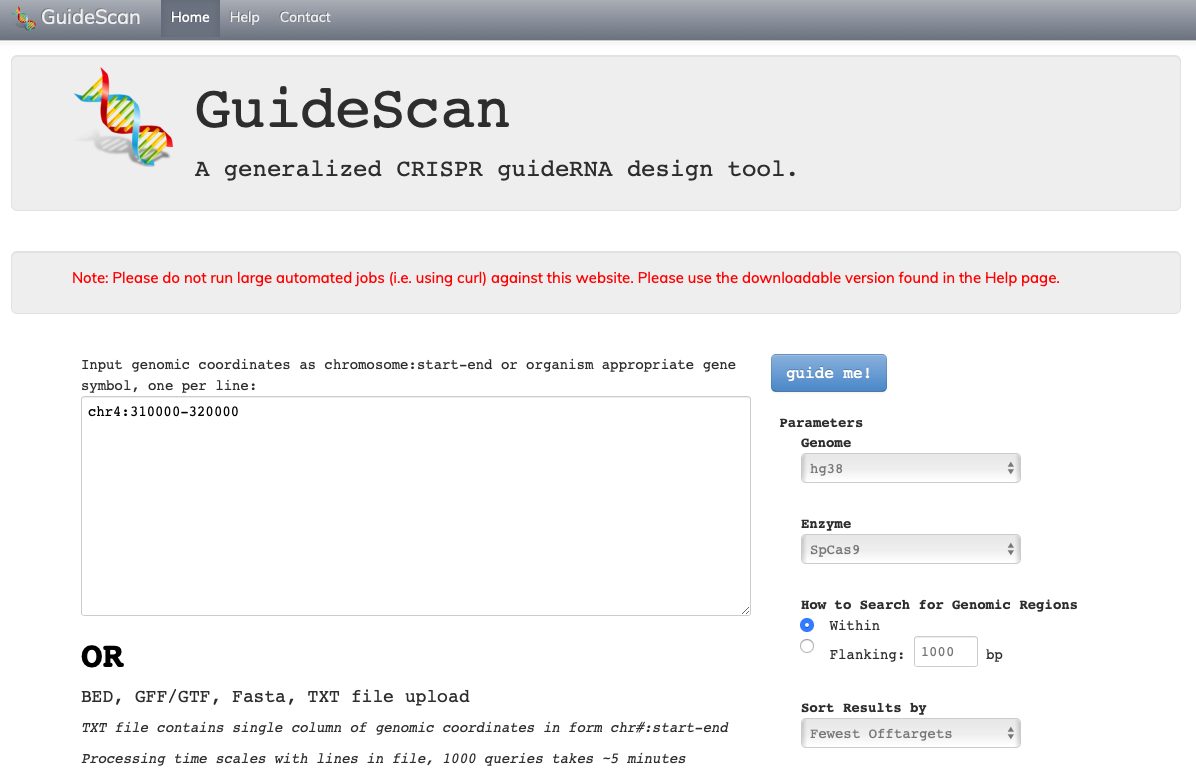
詳細な機能(クリックで展開)
| 対応ゲノム種 | 対応PAM種 | 検索可能な配列長 | 染色体領域指定検索 | オンターゲット予測 | フレームシフト予測 | オフターゲットスコア予測 | N bpミスマッチ領域検索 |
|---|---|---|---|---|---|---|---|
| ヒトを含む6 | NGG-3'/NAG-3'/5'-TTTN | 100kb | ✅ | ✅ | ❌ | ✅ | ✅ |
| (Doench et al., 2016.)モデル | (Doench et al., 2016.)モデル | N<4 bp |
| その他機能 |
|---|
| 指定範囲の隣接領域も検索可能 |
| 各スコアごとにソート可能 |
| 設計したsgRNAのなかで指定数だけをピックアップ可能 |
| BED, GFF/GTF, Fasta, TXTを入力として受けることが可能 |
| アノテーションのついた領域かどうかの判定を出力 |
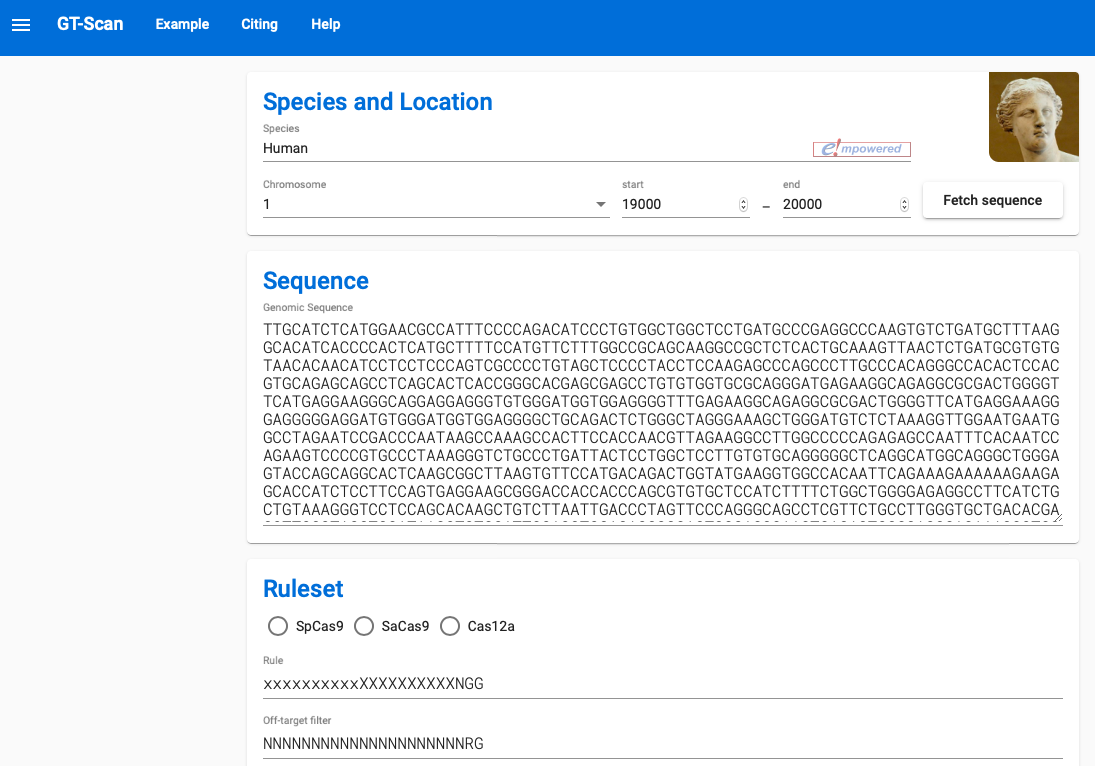
6 GT-Scan(提供元:[Institute for Molecular Bioscience](https://imb.uq.edu.au), クイーンズランド大学, オーストラリア🇦🇺)
:sgRNA設計サイト。染色体領域を指定すれば、その領域全体にわたってsgRNAを設計してくれる。SaCas9・Cpf1にも対応。論文:O’Brien and Bailey., 2014
7 GT-Scan2(提供元:[Institute for Molecular Bioscience](https://imb.uq.edu.au), クイーンズランド大学, オーストラリア🇦🇺)
:GT-Scanのアップデート版。細胞種に応じたオンターゲット活性予測もしてくれる。
8 CRISPick (GPP sgRNA Designer)(提供元:ブロード研究所, アメリカ🇺🇸)
:sgRNA設計サイト。設計結果ファイルに含まれる情報は多岐にわたっており、オフターゲットサイトに関してはその場所のアノテーション情報に応じて個々に集計した結果なども含まれる。
詳細な機能(クリックで展開)
| 対応ゲノム種 | 対応PAM種 | 検索可能な配列長 | 染色体領域指定検索 | オンターゲット予測 | フレームシフト予測 | オフターゲットスコア予測 | N bpミスマッチ領域検索 |
|---|---|---|---|---|---|---|---|
| ヒトを含む6 | NGG-3'/NNGRR-3'/5'-TTTV | 20kb | ❌ | ✅ | ❌ | ✅ | ✅ |
| (Doench et al., 2016.)モデル | 独自モデル(CFD score of rule set ("1")) | 不明 |
| その他機能 |
|---|
| CRISPRa/iの設計にも対応 |
| txtファイル形式の出力 |
| オフターゲット領域のアノテーションから判定を分類(Off-Target Tier Policy) |
| sgRNA周辺配列を出力(sgRNA Context Sequence) |
| sgRNAが標的とするエクソンの番号と位置を出力 |
| オフターゲットスコアに関してBinごとに該当数を表示 |
| 何らかの懸念があるsgRNAにはコメントを付与 |
:米国で核酸販売を行っているIntegrated DNA Technologies(IDT)社が運営するsgRNA設計サイト。sgRNAを設計すると購入の見積もりも同時にだしてくれる。
10 Knockout Guide Design - Synthego(提供元:Synthego, アメリカ🇺🇸)
:sgRNA設計ツール。オンターゲット予測とオフターゲット予測から最適な候補リストをサジェストしてくれる。Synthego社が提供しているModified sgRNAをそのまま注文することも可能。
11 Breaking-Cas(提供元:BioinfoGP!, CNB-CSIC, スペイン🇪🇸)
:sgRNA設計サイト。様々な生物種のゲノムに対応している。論文:Oliveros et al., 2016
12 CRISPR Design Tool(提供元:Horizon Discovery Ltd, イギリス🇬🇧)
:sgRNA設計ツール。タンパクコード領域だけでなく、miRNAやlncRNAを標的とすることもできる。自動的に最適なもののみをサジェストするためわかりやすい。設計した標的を基にHorizon Discovery社が提供しているレンチウイルス用のsgRNAなどをそのまま注文できる。
Horizon Discovery - [webserver] - It provides an intuitive one-stop location for guide RNA design and ordering. Use the design tool to order guide RNAs for targeted gene knockout or HDR-mediated genome editing.
13 CRISPR Targeted Gene Designer(提供元:Horizon Discovery Ltd, イギリス🇬🇧)
:CRISPR Design Toolと同様だが、ターゲットとする領域を選択することができる。
14 CT-Finder(提供元:Liang Lab, マイアミ大学, アメリカ🇺🇸)
:sgRNA設計サイト。Cas9の他、Cas9ニッカーゼやdCas9-Fok1、Cas13aなどに対応している。このサイトのアップデート版がCRISPR-DT。
15 PAVOOC(提供元:スイス連邦工科大学チューリッヒ校, スイス🇨🇭)
:sgRNA設計サイト。標的に関してGenome Browserでの座標表示や標的遺伝子のPDBファイルの立体画像表示もできる。論文:Schaefer et al., 2019
16 Wellcome Sanger Institute Genome Editing(WGE)(提供元:Wellcome Sanger Institute, イギリス🇬🇧)
:sgRNA設計サイト。Genome Browserを操作しながらの設計やオフターゲット予測ができる。ぱっと見だとツールへの入り口がわかりにくいが、画像をクリックすれば各ツールへジャンプできる。
17 Cas-Designer - RGEN Tools(提供元:Molecular Genome Engineering 研究室, 漢陽大學校, 韓国🇰🇷)
:sgRNA設計サイト。VQR SpCas9などあまり広くは知られていないCas9やCpf1の検索にも対応している。
18 CRISPR-ERA(提供元:Bioinformatics 部門, 清華大学, 中国🇨🇳)
:CRISPR-a/i用のガイド設計ツール。通常のCRSIPR-Cas9用の設計も可能
19 Spacer Scoring for CRISPR(SSC)(提供元:Cistrome Project, ハーバード大学, アメリカ🇺🇸)
:Xu., 2015のモデルを利用したsgRNA活性予測サイト。

20 WU-CRISPR(提供元:Xiaowei 研, セントルイス・ワシントン大学, アメリカ🇺🇸)
:Wong et al., 2015のモデルを利用したsgRNA活性予測サイト。
21 TUSCAN(提供元:CSIRO, AEHRC, オーストラリア🇦🇺)
:配列ベースのオンターゲット予測サイト。論文:Wilson et al., 2018
22 CCTop(提供元:Centre for Organismal Studies, ハイデルベルク大学, ドイツ🇩🇪)
:Labuhn et al., 2018のモデルを利用したsgRNAオフターゲット活性予測サイト。
23 CRISTA(提供元:Mayrose 研, テルアヴィヴ大学, イスラエル🇮🇱)
:Abadi et al, 2017に基づいたsgRNAオフターゲット活性(?)予測サイト。

24CINDEL(Bioinformatics & Genomics (BIG) Lab, 韓国🇰🇷)
:Kim et al., 2017に基づくAsCpf1のオンターゲット活性予測サイト。

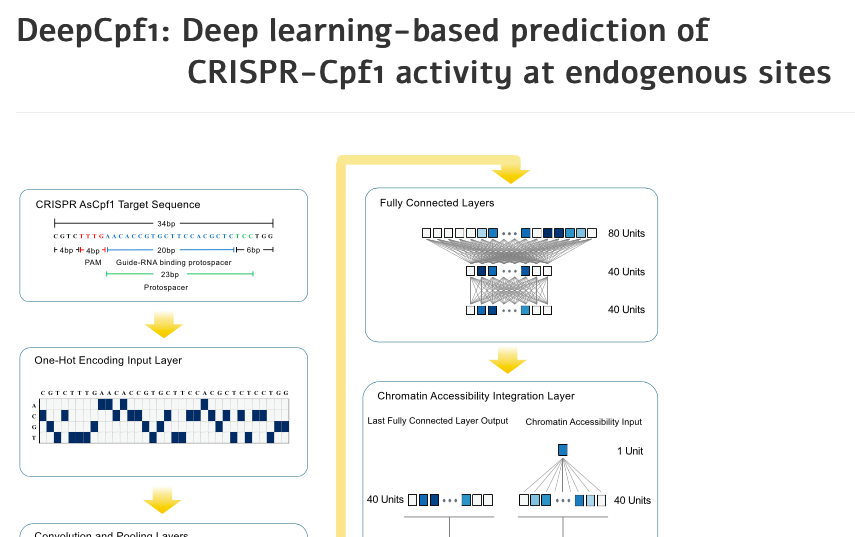
25 DeepCpf1(提供元:Laboratory of Genome Editing & Data Science & Artificial Intelligence Laboratory, 延世大学校 & ソウル大学校, 韓国🇰🇷)
:Cpf1のオンターゲット活性予測サイト。報告ではCINDELよりも高精度といわれている。。
26 CRISPR-DT(提供元:Liang Lab, マイアミ大学, アメリカ🇺🇸)
:Zhu and Liang., 2018に基づいたガイドのオン・オフターゲット活性予測サイト。Cpf1、Cas9、Cas9 nikase、dCas9-Fok1に対応。

27 DeepCRISPR(提供元:Bioinformatics department, 同済大学, 中国🇨🇳)
:Chuai et al., 2018に基づいたCas9ガイド設計・オンターゲット活性予測サイト(現在、サイトはアクセス不可?)。CUIバージョンはこちら。
28 DeepSpCas9variants(提供元:Laboratory of Genome Editing, 延世大学校, 韓国🇰🇷)
:Kim et al., 2020に基づいたCas9ガイド設計・オンターゲット活性予測サイト。SpCas9・Sniper-Cas9・xCas9・eSpCs9 (1.1)・HypaCas9:evoCas9。VRQR variant・SpCas9-NGといった様々なCas9バリアントでの予測値を算出できる。
29 DeepHF(提供元:State Key Laboratory of Genetic Engineering, 復旦大学, 中国🇨🇳)
:Cas9ガイド設計・オンターゲット活性予測サイト。RNNベースのモデルが特徴。Base EditorやBase Editorを使ったノックアウト(BeStop)にも対応している。
30 sgRNA Scorer 2.0(提供元:フレデリック国立研究所(FNLCR)[https://frederick.cancer.gov], アメリカ🇺🇸)
:複数のCas9オーソログも含めたsgRNA活性を予測を行える。論文:Chari et al., 2017
31 ATUM(提供元:ATUM, アメリカ🇺🇸)
:sgRNA設計サイト。大きめの欠失を導入できるpaired sgRNAの設定が可能。オフターゲット活性の予測もしてくれる他、標的領域がカバーできるスプライシングバリアント数も表示可能。
32 CRISPETa(提供元:CRG Bioinformatics Core Facility, スペイン🇪🇸)
:sgRNA設計サイト。BED・UCSCフォーマットを入力として大きめの欠失を導入できるpaired sgRNAの設定が可能。結果をCSV/Excel/BEDファイルでダウンロードできる。論文:Pulido-Quetglas et al., 2017
33 AsCRISPR(提供元:National Clinical Research Center for Geriatric Disorders, 中南大学, 中国🇨🇳)
:片アレルに変異があるゲノム領域を標的とするsgRNA(allele-specific sgRNA)を設計するツール。dbSNPとも連携していてとても使い勝手がよい。
34 SNP-CRISPR(提供元:DRSC/TRiP Functional Genomics Resources, ハーバード大学 医科大学院, アメリカ🇺🇸)
:SNPを標的としたガイドを設計するためのツール。ハエのゲノムにも対応していて、なおかつSNP情報はユーザー自身で定義できる。
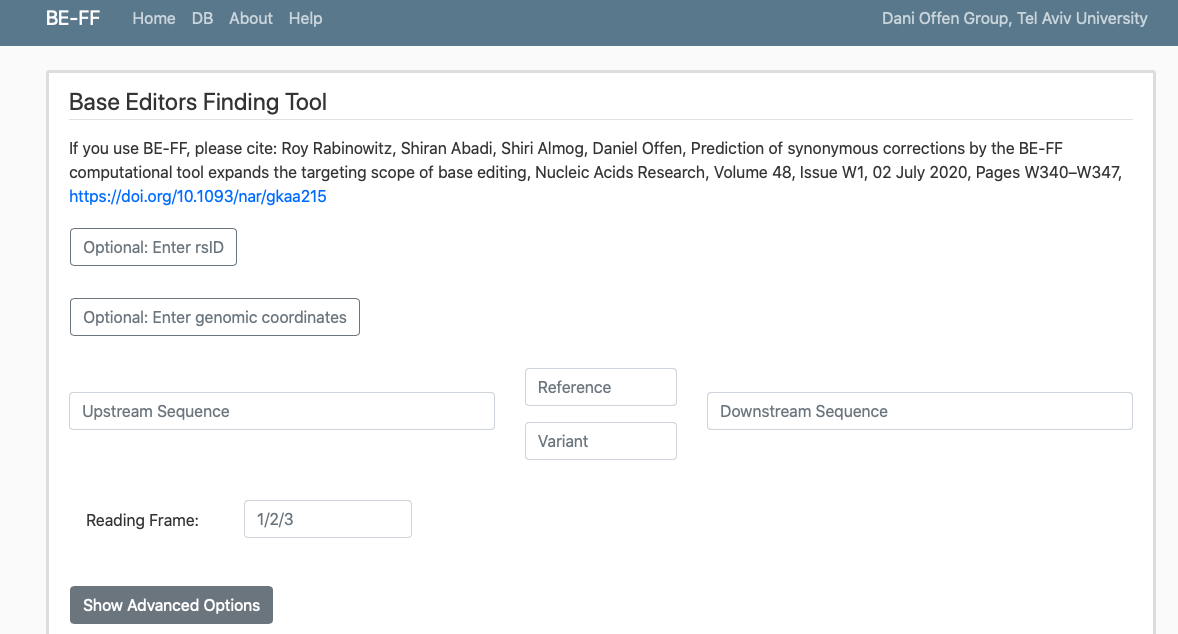
35 CrisPam(提供元:Dani Offen Group, テルアヴィヴ大学, イスラエル🇮🇱)
: SNP用sgRNA設計ツール。論文:Rabinowitz, Darnell, and Offen., 2019
36 CRISPR MultiTargeter
:一つのsgRNAで複数領域をターゲティングできるcommon sgRNAを検索するツール。論文:Prykhozhij et al., 2015(提供元:IWK ヘルス・センター, カナダ🇨🇦)

37 DRSC Find CRISPRs(提供元:DRSC/TRiP Functional Genomics Resources, ハーバード大学 メディカル・スクール, アメリカ🇺🇸)
:ショウジョウバエ用のsgRNA設計サイト。オンターゲット・オフターゲット・フレームシフト率の予測が可能。
38 CRISPR Optimal Target Finder(旧flyCRISPR)(提供元:ブラウン大学, アメリカ🇺🇸)
:sgRNA設計サイト。ショウジョウバエを中心としたゲノムを対象にできる。論文:Gratz et al., 2014

39 CRISPRscan(提供元:Giraldez Lab, イェール大学, アメリカ🇺🇸)
:ゼブラフィッシュでの知見を基にしたsgRNA設計ツール。Moreno-Mateos et al., 2015
40 CRISPR-P(提供元:National Key Laboratory of Crop Genetic Improvement & Center for Bioinformatics, 華中農業大学, 中国🇨🇳)
:植物での利用を念頭においたsgRNA設計ツール。オン・オフターゲット予測にも対応。
41 WheatCRISPR(提供元:カナダ国立研究評議会, カナダ🇨🇦)
:コムギ専用のsgRNA設計ツール。オン・オフターゲット予測にも対応。
42 CRISPR-Local(提供元:提供元:National Key Laboratory of Crop Genetic Improvement & Center for Bioinformatics, 華中農業大学, 中国🇨🇳)
:植物用のsgRNA設計サイト兼データベース。様々な植物のゲノムでの設計に対応している。オフラインでも利用できるようにソフトウェアやゲノムデータがダウンロード可能。論文:Sun et al., 2019
- [software, webserver] - A local single-guide RNA (sgRNA) design tool for non-reference plant genomes

43 CRISPR-Plant(提供元:ペンシルベニア州立大学 and Arizona Genomics Institute, アメリカ🇺🇸)
:植物用のsgRNA設計サイト。植物のゲノムに対して指定領域内のsgRNA設計可能領域を表示できる。オフターゲットに関してはCGとAGのミスマッチをそれぞれ分けてカウントすることが可能。論文:Kabin, Jianwei, and Yinong., 2014
44 EuPaGDT(提供元:Tarleton Research Group, The Center for Tropical and Emerging Global Diseases, The University of Georgia, アメリカ🇺🇸)
:病原性真核生物用のsgRNA設計サイト。予測オンターゲットスコアやオフターゲットスコア、Microhomology score等も算出可能論文:Peng and Tarleton., 2015
45 CRISPy-web(提供元:DTU Biosustain, デンマーク工科大学, デンマーク🇩🇰)
:バクテリア・真菌での利用を念頭においたsgRNA設計ツール。バクテリア・真菌ゲノムデータベースantiSMASHと連携している。
46 phytoCRISP-Ex(提供元:Institut de Biologie de l'ENS (IBENS), フランス🇫🇷)
:sgRNA設計サイト。藻類等のゲノムを対象としたsgRNAを設計できる。
47 Green listed(提供元:Wermeling 研, カロリンスカ研究所, スウェーデン🇸🇪)
:プール型CRISPRスクリーニング用ガイドライブラリ設計ツール
48 CRISPR-FOCUS(提供元:Xiaole Shirley Liu 研, ハーバード大学 & ダナ・ファーバー癌研究所, アメリカ🇺🇸)
:プール型CRISPRスクリーニング用ガイドライブラリ設計ツール。コントロール用のsgRNA検索機能やがん関連遺伝子の除外等のオプションが存在する。またdbSNPを自動参照してSNPが入っていそうな箇所はなるべく避けるようにデザインするようだ。
49 Slice It(提供元:Janga 研, インディアナ大学, アメリカ🇺🇸)
:RNA Binding Protein (RBP)が結合する領域を確認しながらガイドの設計ができるツール。おそらくgenome browserを使っても同様のことが可能だが、自前で設定することなく参照できるのは便利。
50 PITCh designer 2.0(提供元:Yamamoto 研, 広島大学, 日本🇯🇵)
:インタラクティブなMMEJノックイン配列デザインサイト。挿入配列はプリセットもあるが、ユーザー側で定義もできるようになっている。標的サイトをコドンを参照しながら選ぶなどなるべく手軽に使えるようにしている。
51 GTagHD - Gene Sculpt Suite(提供元:Gene Sculpt Suite, アイオワ州立大学&メイヨー・クリニック, アメリカ🇺🇸)
:MMEJを使ったノックアウト・ノックインデザインサイト。Gene ID等を入力すればツール側で入力配列を用意してくれる。
52 TAG-IN(提供元:Pollard 研, エディンバラ大学, イギリス🇬🇧)
:ssDNAベースのタグづけノックインを設計するツール。[RUN BATCH]から一度に複数遺伝子座のデザインを設計することも可能。
53 CUNE(提供元:CSIRO, AEHRC, オーストラリア🇦🇺)
:ssODNベースのノックイン設計ツール。O’Brien et al., 2019に基づくノックイン活性スコアも出力できる。
54 MENTHU - Gene Sculpt Suite(提供元:Gene Sculpt Suite, アイオワ州立大学&メイヨー・クリニック, アメリカ🇺🇸)
:ノックアウト時のMMEJの起こりやすさを基にガイド標的をサジェストしてくれるツール。ゲノム配列もしくはID名を入力すれば自動的に検索を行ってくれる。MMEJの起こりやすさを示す指標はMicrohomology-Predictorとは異なるものを使っている。
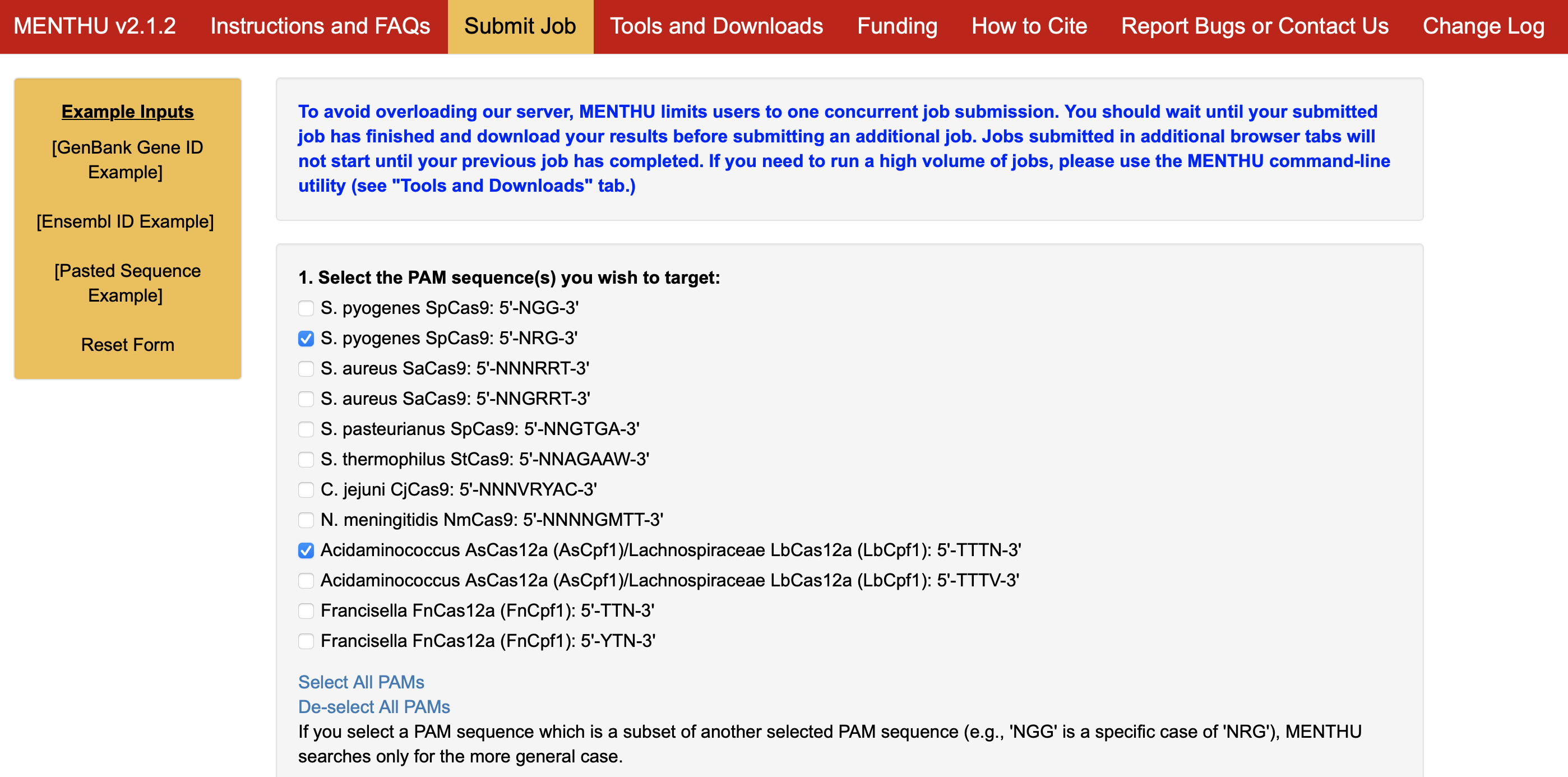
55 CRISPR-PN2(提供元:O'Halloran研, ジョージ・ワシントン大学, アメリカ🇺🇸)
:寄生線虫用のsgRNAターゲット検索ツール(論文:O'Halloran, 2021)。
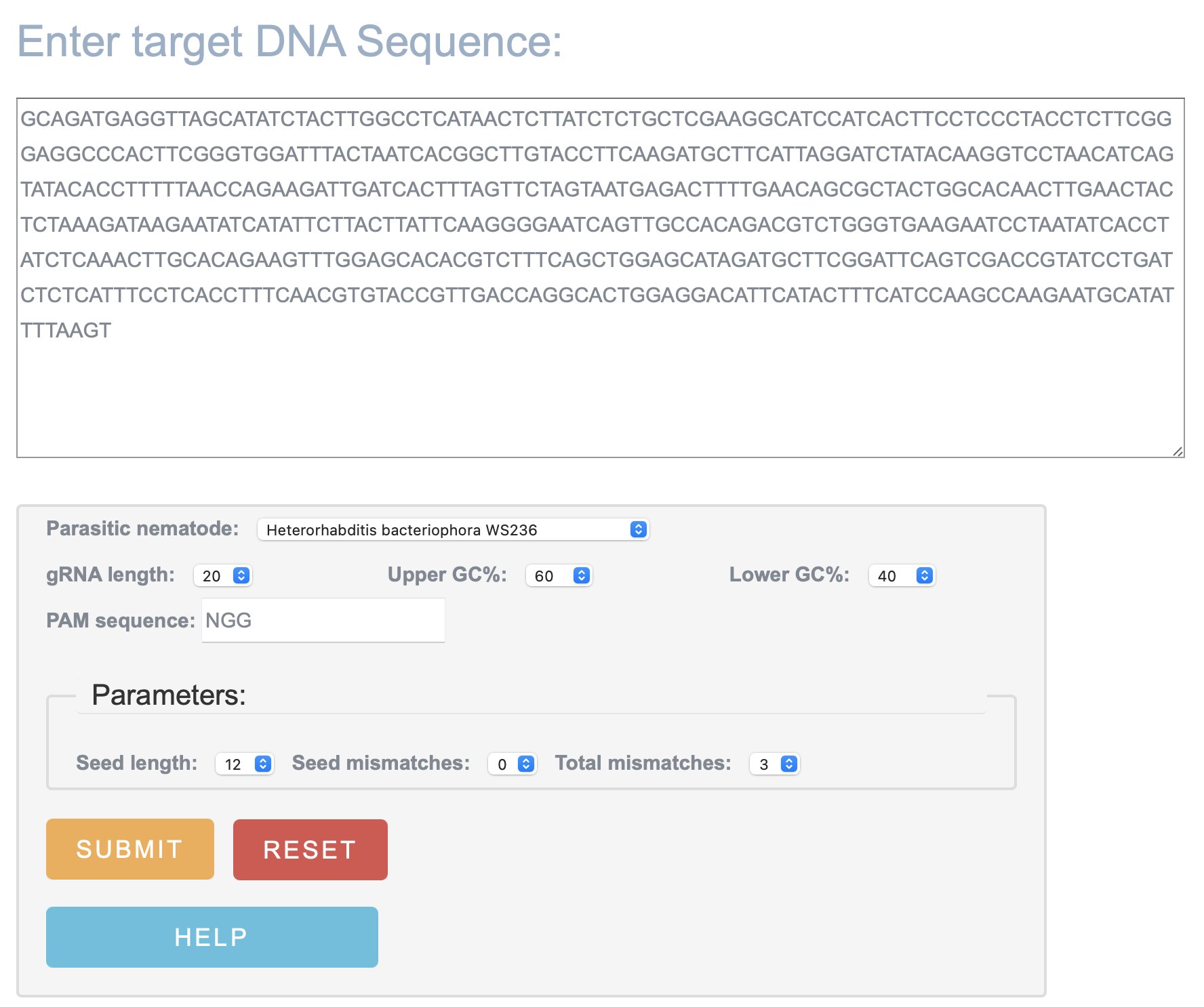
56 CRISPR-Cereal(提供元:National Key Laboratory of Crop Genetic Improvement, Center for Bioinformatics, 華中農業大学, 中国🇨🇳)
:コムギ・トウモロコシ・イネ用のsgRNA検索ツール。オフターゲット検索の他、RNA-seqやヒストン修飾、SNPの情報などにアクセスできるようになっている(論文:He et al., 2021)。
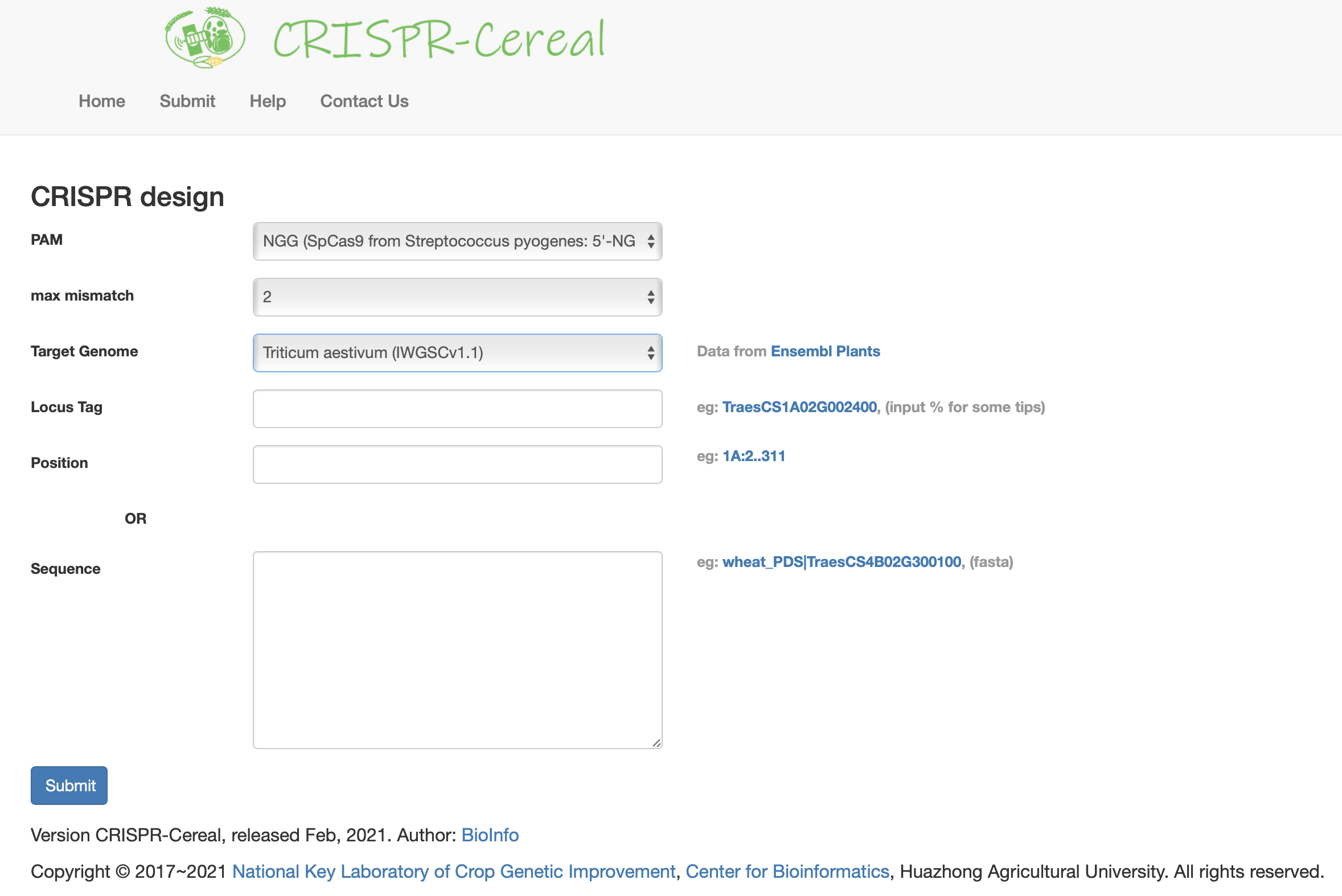
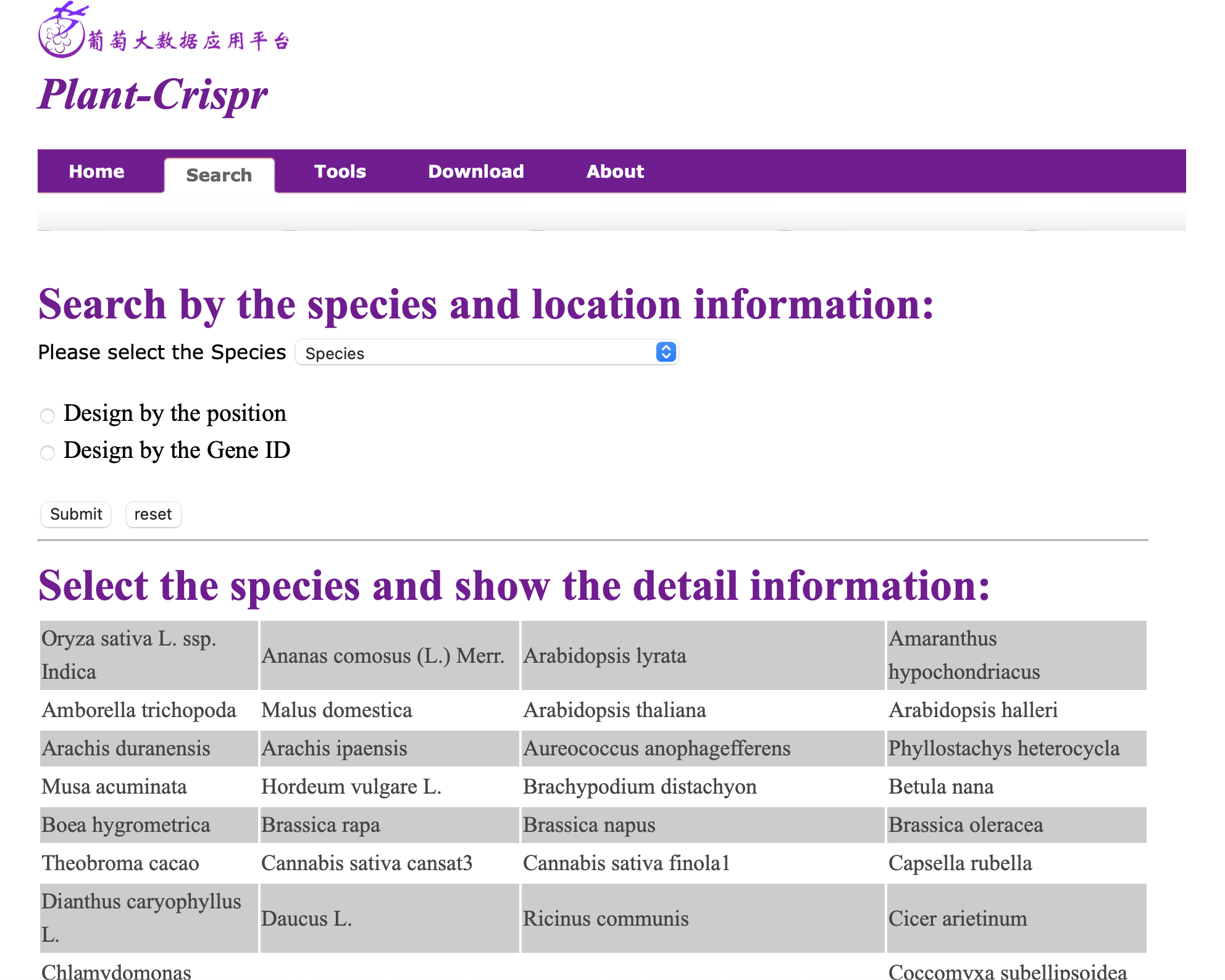
57 Plant-Crispr(提供元:中国科学院植物学研究所, 中国🇨🇳)
:138種の植物のsgRNAを検索できるツール(論文:Wang et al., 2021)。
58 GuideMaker(提供元:United States Department of Agriculture - Agricultural Research Service (USDA-ARS), アメリカ合衆国農務省, アメリカ🇺🇸)
:非モデル生物向けのゲノムワイドなガイド配列検索ツール。500MB以下のGZ圧縮ゲノム配列データをユーザーに提出させて検索する仕様(論文:Poudel et al., 2021)。
59 DeepPE(提供元:Hyongbum Henry Kim Lab, 延世大学校, 韓国🇰🇷)
:Kim et al., 2021のモデルに基づいてPrime Ediitng(特にPE2)を行う上でに最適なターゲットを提案するツール。論文:Kim et al., 2021。

60 KOnezumi(提供元:生命科学動物資源センター, 筑波大学, 日本🇯🇵)
:KOマウス作成のためのCRISPR/Cas9デザインを全自動でできるウェブツール。論文:Kuno, Mizuno, and Takahashi., 2019。
61 Machine learning-based end-to-end CRISPR/Cas9 guide design (提供元:Microsoft Research, アメリカ🇺🇸)
:機械学習予測モデルであるAzimuth/Elevationを利用したオンターゲット・オフターゲット予測が可能なガイドRNA検索ツール。利用には無償のログイン登録が必要。
論文:Listgarten et al., 2018, Doench et al., 2016

コマンドラインツール(23件)
C/C++
-
Crisflash
:sgRNA設計ツール。オフターゲット検索にも対応した高速検索が特長。 -
uCRISPR
:オンターゲット・オフターゲット予測ツール。Unified energetics analysisという手法を使っている。論文:Zhang et al., 2019
R
-
CRISPRseek
:オフターゲットを考慮しながらガイドの設計ができるツール。制限酵素サイトを含まないガイドのみを検索するなどちょっと変わった設計オプションもついている。 -
crispRdesignR
:ガイドの設計ができるツール。オン・オフターゲット予測対応。ローカルでGUIアプリを立ち上げることができるようになっている。 -
predictSGRNA
:Kuan et al., 2017でのモデルを利用したsgRNA活性予測ツール -
CRISPRpred
Rahman et al., 2017 でのモデルを利用したsgRNA活性予測ツール -
CRISPR Library Designer (CLD)
:CRISPRスクリーニング用ガイドライブラリ設計ツール
python
-
GuideScan
:sgRNA設計ツール。GuideScanの処理をマルチプロセス化して高速化させたもの。 -
AlleleAnalyzer
:allele-specific sgRNAを設計するツール。11種類のCas9オーソログに対応している。 -
pgRNAFinder
:paired-sgRNAsの設計ができるツール。論文ではウェブツールも紹介されているが、現在はアクセスできない。 -
TUSCAN
:Wilson et al., 2018に基づく高速なsgRNA活性予測モデル。 -
off_target_prediction
:Lin and Wong., 2018に基づく高速なsgRNAオフターゲット活性予測モデル。 -
GNL_Scorer
: Wang et al., 2020 でのモデルを利用したsgRNA活性予測モデル。 -
SSFinder
:sgRNA設計ツール。 -
getCRISPRY
:sgRNA設計ツール。Doench et al., 2016に基づいたオフターゲット予測が可能。 -
CRISPR TRAP-seq
:Xiang et al., 2020で作製したGNL_Scorerのアップデート版。ABE/CBEにも対応している。 -
pgRNADesign(提供元:X. Shirley Liu Lab, ハーバード大学, アメリカ🇺🇸)
:sgRNA設計サイト。lncRNAに対するpaired sgRNAを作製できるため確度の高いノックアウトが期待できる。 -
CRISPR-SURF
:遺伝子機能解析を行うためのsgRNA設計ツール。Hsu et al., 2018 -
sgRNA-cleavage-activity-prediction
:Guo et al., 2018に基づいたバクテリア用のsgRNAオンターゲット活性予測モデル。 -
DeepSgrnaBacteria
:Wang and Zhang., 2019に基づいたバクテリア用のsgRNAオンターゲット活性予測モデル。 -
AttnToCrispr_CNN
オフターゲット作用とオンターゲット活性の予測ツール。Attention-based transformerという学習システムを採用している。一部のモデルでは発現データも予測のための材料としている。論文:Liu, He, and Xie, 2019 -
CRISPR-ONT & CRISPR-OFFT
オフターゲット作用とオンターゲット活性の予測ツール。Attention-basedなCNNを使っているが、AttnToCrispr_CNNとはレイヤー構造が異なる。また、PAM配列を重視しているという点も特徴の一つとなっている。論文:Zhang et al., 2021 -
Azimuth
オンターゲット予測ツール。他ツールで示されるDoenchのモデルというのはこのモデルを指すことが多い。
論文:Doench et al., 2016 -
Elevation
オフターゲット予測ツール。
論文:Listgarten et al., 2018 -
Elevation-search(dsNickFury3)
オン/オフターゲット予測ツール。オンターゲット予測モデルAzimuthとオフターゲット予測モデルElevationでの評価が可能。
論文:Doench et al., 2016, Listgarten et al., 2018
perl
-
sgRNAcas9 - BiooTools
:オフターゲットを考慮しながらsgRNAをデザインするツール。
JavaScript
-
Guide Picker(提供元:DeskGen, イギリス🇬🇧)
sgRNA設計ツール。ウェブ版はDeskGenで利用可能。npmライブラリでMITライセンス。
アプリケーション
Protospacer Workbench
:sgRNA設計アプリ。
TALEN設計
TALENに関しても設計ツールがいくつか開発されています。
ウェブツール(1件)
-
TAL Effector Nucleotide Targeter 2.0
:TALENの配列設計サイト。広島大の設計プロトコルも併せて参照するとよい。

コマンドラインツール(1件)
JAVA
-
TALENoffer
:TALE用のオフターゲット検索ツール。幅広いオプションが用意されている。
ZFN設計
ZFNに関しても設計ツールがいくつか開発されています。
ウェブツール(1件)
-
ZFN-Site
:ZFNのオフターゲット評価サイト。ヒトとマウスゲノムに対応。
論文:Cradick et al., 2011

Base Editing用ガイド設計
デアミラーゼを使ってDSB切断なしで一塩基置換を誘導するBase Editingは、主にCRISPRの派生技術であるため、ガイド設計の基本的な部分は変わりません。ただし編集ウインドウといった独特の問題が存在するため、そういったものを考慮に入れた専用のツールが存在しています。
ウェブツール(3件)
-
BE-Designer
:一塩基置換を入れることができるBase EditorとTarget-AIDのsgRNA設計ツール
-
BEable-GPS
:dCas12a型のBase Editorも含む様々なBEのガイド設計が可能。BEで対処可能な病原SNPを表示する機能(Pathogenic SNVs)もついている。
コマンドラインツール(2件)
-
Base editing prediction model
:Sakata et al., 2019に基づいたTarget-ACEの活性予測ツール
Prime Editing用ガイド設計
逆転写酵素を使って置換・小規模欠失・小規模挿入を導入するPrime Editingは、CRISPRのガイドRNAに逆転転写テンプレート等を付加したpegRNAを利用します。この設計に関してもいくつかツールが存在しています。
ウェブツール(3件)
-
PrimeDesign
:Prime EditorのpegRNAと非標的鎖sgRNAの設計ツール
論文:Hsu et al., 2021。

-
Prime Editing Design Tool(旧Primeedit)(提供元:Sanjana研, ニューヨークゲノムセンター & ニューヨーク大学, アメリカ🇺🇸)
:Prime Editing用のpegRNA設計サイト。PE2, PE3, PE3b用にそれぞれ設計が可能で、病原変異の作製・修正を目的としたpegRNA設計を行うことができる。論文:Morris et al., 2020。

-
pegFinder(提供元:Sidi Chen研, イェール大学, アメリカ🇺🇸)
:Prime Editing用のpegRNA設計サイト。目的の配列を入力すればそれに応じたpegRNA、またその発現ベクターのクローニングに使うオリゴ配列等を出力してくれる。論文:
Chow et al., 2020

-
PlantPegDesigner(提供元:State Key Laboratory of Plant Cell and Chromosome Engineering, 中国科学院, 中国🇨🇳)
植物でのゲノム編集に最適化したPEデザインツール。dual型のPrime Editing設計が可能。
論文:Lin et al., 2021

-
pegIT(提供元:Department of Biomedicine, オーフス大学, デンマーク🇩🇰)
遺伝子名やClinVarのID等から設計可能なPEデザインツール。
論文:Anderson et al., 2021

-
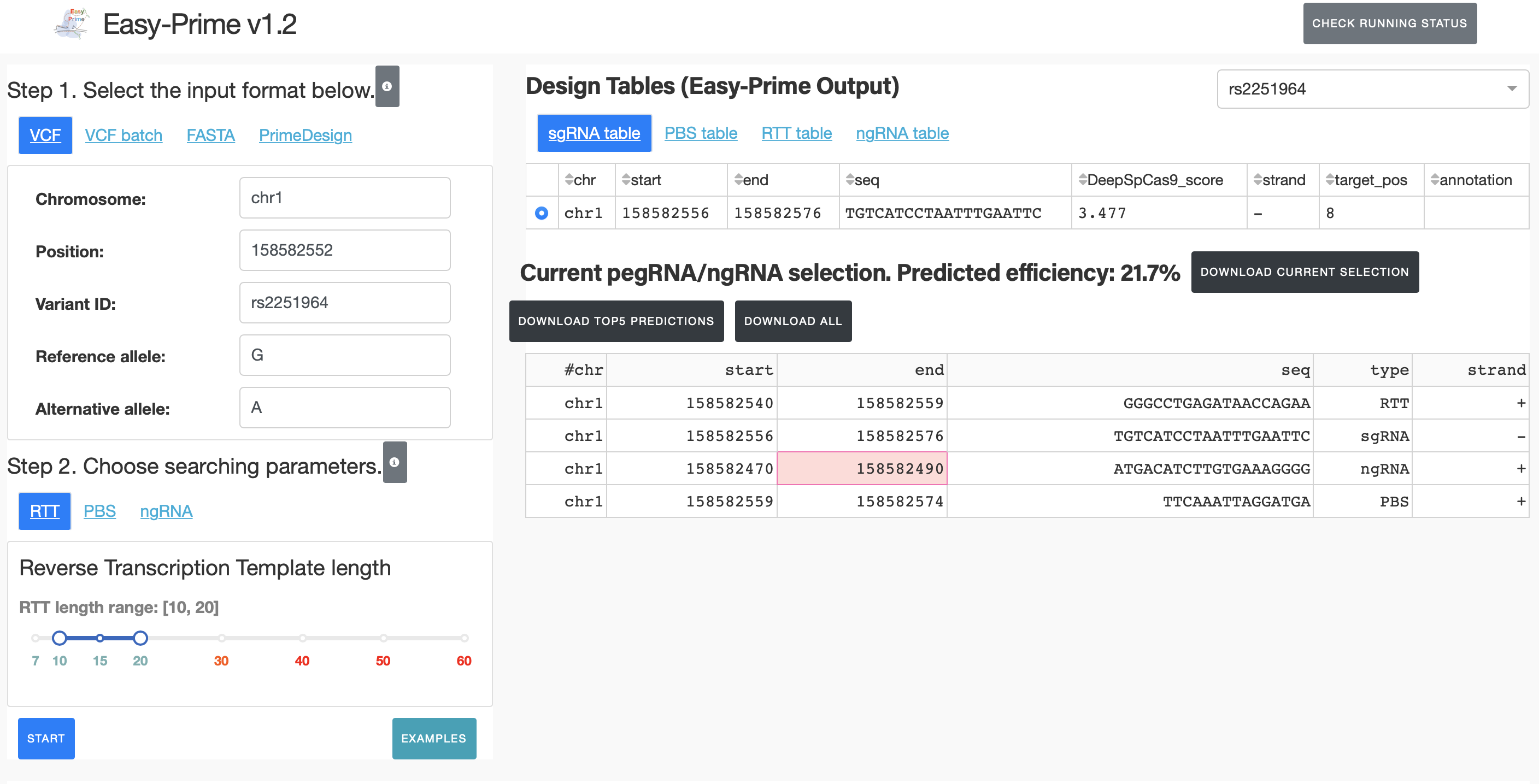
Easy-Prime(提供元:Cheng Lab, セント・ジュード・チルドレンズ・リサーチ病院, アメリカ🇺🇸)
様々な入力形式に対応したPEデザインツール。DeepSpCas9のスコアを参照しながら設計することができる。
論文:Li et al., 2021
-
PE-Designer - RGEN Tools(提供元:Molecular Genome Engineering 研究室, 漢陽大學校, 韓国🇰🇷)
インタラクティブに設計できるPEデザインツール。あらゆる生物種に対してオフターゲットの評価をすることも可能。
論文:Hwang et al., 2021。

-
Prime-del(提供元:Shendure lab, ワシントン大学, アメリカ🇺🇸)
dual型のPrime Editing設計用ツール。
論文:Choi et al., 2021。

-
pegLIT(提供元:Liu lab, ブロード研究所, アメリカ🇺🇸)
epegRNAの作成において必要なリンカー配列の最適な設計をサジェストするツール。
論文:Nelson et al., 2021。

コマンドラインツール(1件)
-
MinsePIE
:Koeppel et al., 2021に基づいたPrime Editingによる挿入編集活性予測ツール。
Cas13用ガイド設計
ゲノム編集では二本鎖DNAを標的としますが、Cas13を使ったRNA編集ではmRNAなどの一本鎖RNAを標的とします。ガイドRNAはCas9などとはまた違うので異なった設計手順となります。それを補助するのが以下のツールです。
ウェブツール(1件)
-
CRISPR-RT
:Zhu, Richmond, and Liang., 2017に基づいたCas13aガイドのオン・オフターゲット活性予測サイト。

-
Cas13design
:ガイドRNAの設計ツール。Wessels et al., 2020に基づいたオンターゲット活性予測も行える。

-
CasRx guide design
:RfxCas13d(CasRx)の標的検索ツール。Wel et al., 2021に基づいたオンターゲット活性予測を行ってくれる。

コマンドラインツール(1件)
-
CASowary
:Krohannon et al., 2021に基づいたオンターゲット活性予測ツール。
標的変異パターン予測
ゲノム編集による人工的なゲノム切断では、修復されてできる変異には一定の法則性があることが報告されており、以下のツールではそれを予測することが可能です。
ウェブツール(7件)
-
Microhomology-Predictor
:ノックアウト時のMMEJの起こりやすさを予測してくれる。シンプルだけど便利。

-
MEDJED - Gene Sculpt Suite
:ノックアウト時のMMEJの起こりやすさを予測するツール。MMEJの起こりやすさを示す指標はMicrohomology-Predictorとは異なるものを使っている。

-
inDelphi
:深層学習モデルベースのノックアウト変異予測サイト。一塩基挿入予測対応。SpCas9ユーザ向け。

-
FORECasT
:機械学習モデルベースのノックアウト変異予測サイト。一塩基挿入予測対応。SpCas9ユーザ向け。

-
SPROUT
:T cellに特化した機械学習モデルベースのノックアウト変異予測サイト。SpCas9ユーザ向け。
*HEK293やHCT116などのラボでよく使われる不死化細胞株とPrimary T cellの変異パターンは違うことが知られている(Leenay, Ryan T., et al.)。

-
VBC-Score
:フレームシフトの起こりやすさをsgRNAの予測変異パターンやタンパクの保存性等から予測するツール。論文:Michlits et al., 2020

-
BE-Hive
:機械学習ベースのBase Editorの変異予測サイト。論文:Arbab et al., 2020

-
BE-DICT
:機械学習ベースのBase Editorの変異予測サイト。論文:Marquart et al., 2021

コマンドラインツール(2件)
R
-
MEDJED
:MEDJEDのCUI版。
Python
-
inDelphi-model
:inDelphiのpython module。大量に予測したい場合は非常に使いやすい。 -
SelfTarget
:FORECasTのCUI版。大量に予測したい場合は非常に使いやすい。Dockerでの利用を推奨。 -
Lindel
:ロジスティック回帰ベースの変異予測ツール。論文:Chen et al., 2019 -
CROTON
:NASつきのCNNをベースとした変異予測ツール。論文:Li, Zhang, and Troyanskaya, 2021 -
MENdel
Martínez-Gálvez et al., 2021のモデルに基づいた変異パターン予測ツール。MENTHUとLindelをベースとしている。
オフターゲット検索
CRISPRの設計ツールでもオフターゲット検索ができますが、以下のものはそれに特化にしたものとなります。
ウェブツール(5件)
-
blastn
:当たり前すぎるがやはり欠かせない。

-
COSMID
:ミスマッチだけでなく挿入・欠失にも対応。とても使えるツール。
*Google Chrome・Firefoxで開くとセキュリティエラーになるが、これはSymantec社のサーバー証明書を使っているため。(私個人としては使っても問題ないかと・・・)

-
CRISPRdirect
:数百種類のゲノムで検索できてかつとても高速。5塩基のミスマッチ・ギャップまで検索が可能。

-
Off-Spotter
:入力した配列から網羅的にガイドを設計し、さらにそこからそれぞれのオフターゲット候補を5ntミスマッチまで検索してくれる。計算量はものすごいはずだがその割には高速。C/C++ ソースコードも配布されている。

-
Cas-OFFinder - RGEN Tools
:許容するバルジやミスマッチを指定できる。VQR SpCas9などあまり広くは知られていないCas9やCpf1の検索にも対応している。

コマンドラインツール(1件)
パイプライン・プラットフォーム
-
CRISPRitz
:オフターゲット検索をするためのパイプライン。エピゲノム情報などのアノテーションも付加できる。
python
-
CNN_predict
:PAMの制限が緩いSpRY用オフターゲット予測ツール。論文:Zhang et al., 2021。
TIDE解析
変異導入したポピュレーションのサンプルをサンガーシーケンスにかけるとピークがぐちゃぐちゃになったデータが見れることはご存知の方も多いかと思います。そういったときに勿体ないので2番目に大きなピークを辿ったりとしてなんとなく複数のクローンを推定したという経験をされた方も少なくないのではないかと思います。
TIDE解析はそういった混合シーケンスデータのベースコールを自動的に行うものです。
ひと昔前ならばこういった方法は不適切と評されたかもしれませんが、最近ではNature本誌論文でも使われたりしていて一般的になりつつあります。
ウェブツール(5件)
-
TIDE
:元祖のTIDE解析ツール。

-
ICE Analysis
:TIDEとは別のアルゴリズムによるTIDE解析。精度や頑強性において非常に高い成績を誇る。
(参照;開発元のSynthegoによるICEとTIDEの比較)

-
CRISP-ID
:二重のシグナルが混在したサンプル解析に特化したTIDE解析ツール。解析アルゴリズムは、TIDEのような重回帰アルゴリズム等の小難しい計算を使ってはおらず、セカンドピークの抽出と簡易アライメントで分離を行うという非常にシンプルなもの。

-
Poly Peak Parser
:二重のシグナルが混在したサンプル解析に特化したTIDE解析ツール。二重よりも多くのシグナルは解析できない。

-
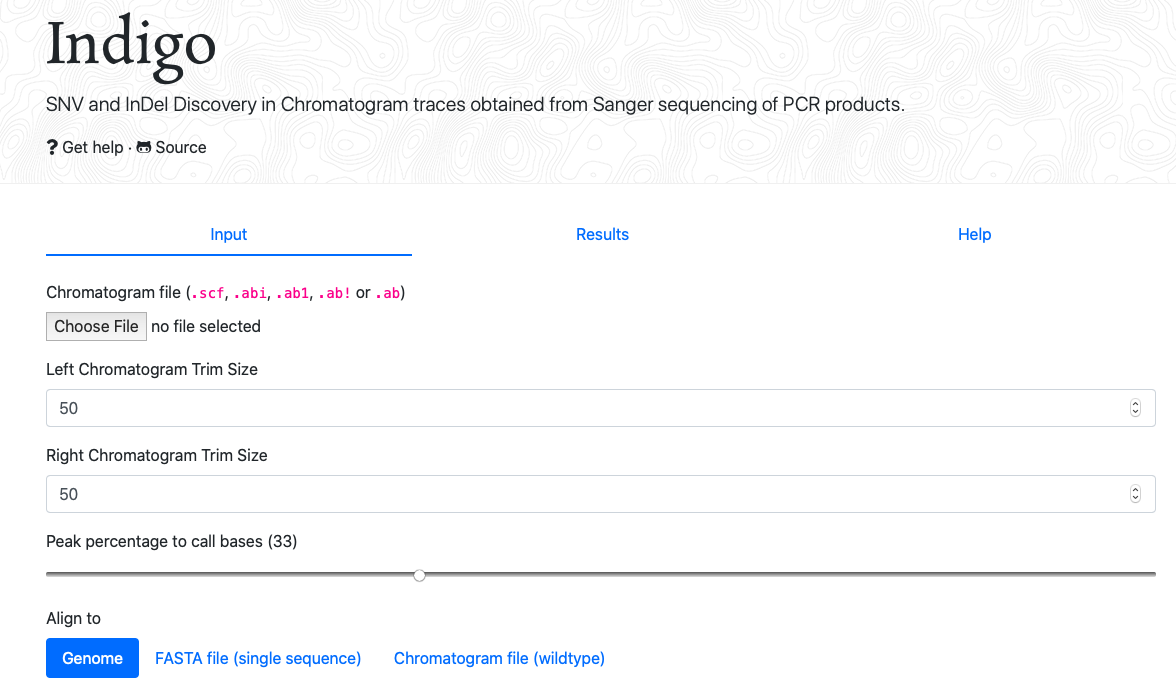
Indigo - GEAR-GENOMICS
:ゲノムリファレンスを選択して解析できる。シーケンスデータがリファレンス配列と合致する領域であるか確信をもてない場合は有用かもしれない。論文は未発表、EMBLがサポート。
-
TIDER
:ノックインサンプルも解析できるようにしたツール。ネガティブ/ポジティブコントロールが必須。論文:Brinkman et al., 2017

-
EditR
:置換検出に特化したBase Editor用のTIDE解析ツール。ネガティブ/ポジティブコントロールが必要ないのでコスト面でも優れている。論文:Kluesner et al., 2018。

-
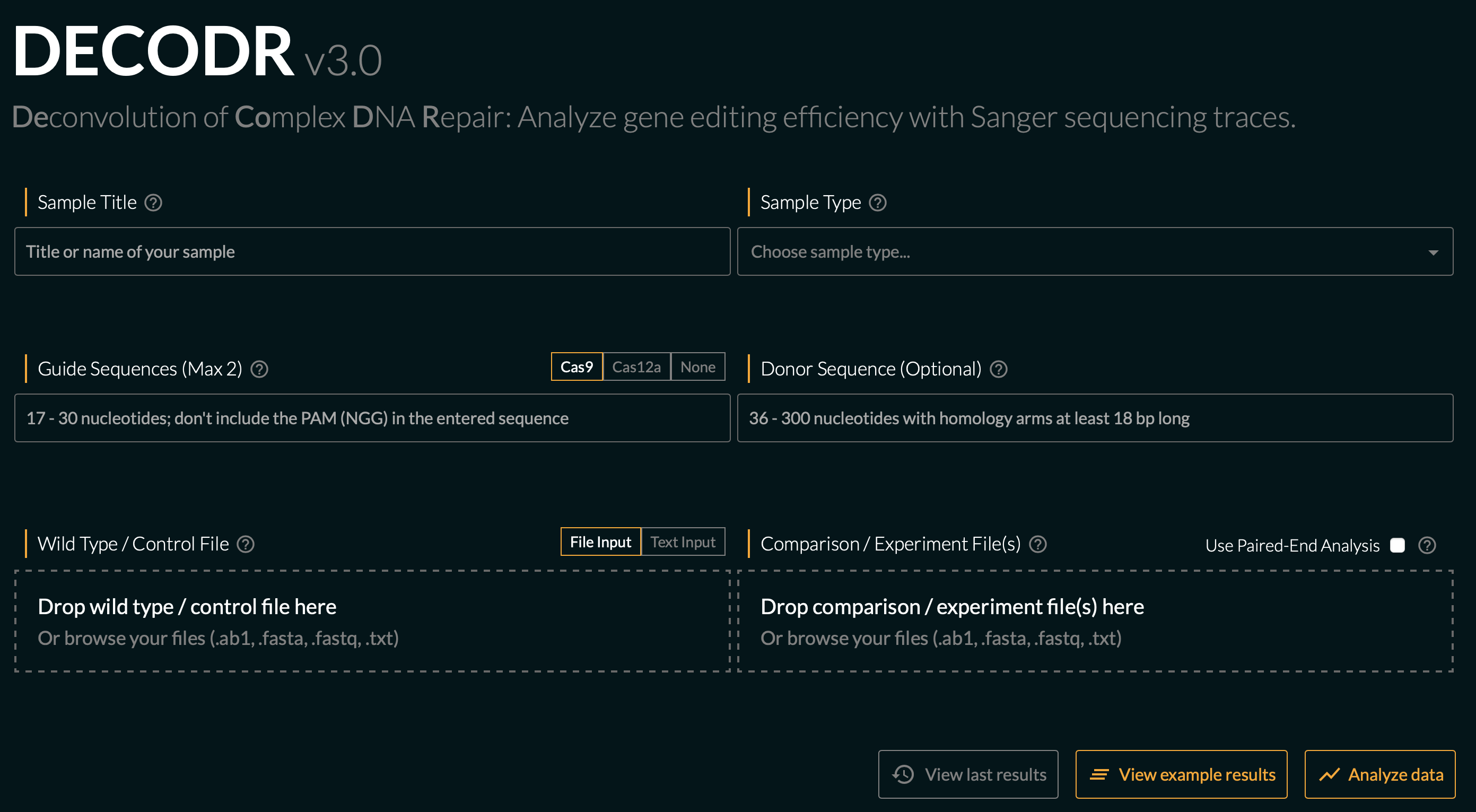
DECODR
:Cas9/Cas12aのインデル検出が可能。ノックインや100bp以上のインデル解析も可能となっている。論文:Bloh et al., 2021。
コマンドラインツール(1件)
R
EditR
:ウェブ版のローカルバージョン
NGSを利用したアンプリコンシーケンシング解析
ゲノム編集の変異解析を(TIDEのような予測なしで)高解像度で行おうとした際には、NGSによるアンプリコンシーケンシングが第一選択となる。
ただ、通常のNGS解析ツールは、CRISPRによる編集の効率や修復パターン判別などゲノム編集で求められる解析やデータには対応していないものが多い。そのため、ゲノム編集用のアンプリコンシーケンシング解析ツールが開発されている。
ウェブツール(6件)
-
CRISPR-GA
:ゲノム編集用NGS解析ソフト。内部でBLATを動かして目的配列の数を定量している。

-
AGEseq
:ゲノム編集用NGS解析ソフト。内部でBLATを動かして目的配列の数を定量して、アライメント図を作成する。

-
CRISPResso2
:ゲノム編集用NGS解析ソフト。おそらく一番使い勝手が良いが、分類精度はAmpliCan・CrispRVariants・AmpliconDIViderに劣る(参照:Labun et al. 2019)。

-
Cas-Analyzer
:ゲノム編集用NGS解析ソフト。アンプリコン配列の両サイドに特異的な配列(indicator)を定めてその間を解析するという簡易的なアルゴリズムで動いているが精度はそれほど悪くない。

-
CLiCKAR
:ゲノム編集用NGS解析ソフト。複数の遺伝子座のアンプリコンが混在したプール型のシーケンスデータの解析が手軽にできる。論文:Iida et al., 2020。

-
Outknocker 2
:ゲノム編集用NGS解析ソフト、ZFN・TALEN実験データでも使えるのが強み。現状ベンチマーク検証はされていないので精度に関しては不明。

-
Hi-TOM
:バーコード付きアンプリコンシーケンシングデータを解析するためのツール。(現在は使用不可?)。

-
BE-Analyzer - RGEN tools
:Base Editing用のアンプリコンシーケンス解析ツール

-
CRISPR-Sub - RGEN Tools
:アンプリコンシーケンスにおいて置換変異が本当にゲノム編集ツールによるものなのかという判定はなかなか難しい。このツールではコントロールサンプルとの同時解析からCas9による編集で出現した置換が本当に編集による置換なのかどうかを評価する。(論文未公表:2020/05/25現在)

-
PE-Analyzer - RGEN Tools(提供元:Molecular Genome Engineering 研究室, 漢陽大學校, 韓国🇰🇷)
Prime Editing用のアンプリコンシーケンス解析ツール。
論文:Hwang et al., 2021。

コマンドラインツール(14件)
R
-
CrispRVariants
:ゲノム編集後の変異シーケンス解析・可視化ツール。NGS解析データにも対応している。仕様がややこしいが慣れるとTALENによる変異シーケンスも解析できるので便利 -
amplican
:ゲノム編集後の変異NGSシーケンス解析・可視化ツール。報告論文の中ではCRISPResso2の精度よりも優れているようだ。 -
AlleleProfileR
:ゲノム編集後の変異NGSシーケンス解析ツール。バッチモードを備えている。
python
-
CRISPResso
:ゲノム編集後の変異NGSシーケンス解析・可視化ツール。使いやすさを重視しているためか近年の利用報告は多い。またマッピングを必要としないのも特徴の一つ。ただCrispRVariantsの報告論文等では変異種の分類がやや不正確であることが指摘されているので、解析結果の配列は毎回見直すようにしたほうがよい。 -
CRISPResso2
:CRISPRessoのアップデート版。主たる仕様は変わっていないが、Base editor用の設定等が追加されている。分類精度も若干の改善がみられており、著者グループはこちらの使用を推奨している。ちなみにconda環境では動かないことがあるので、Dockerのほうの利用を推奨したい。 -
CRISPRMatch
:ゲノム編集後の変異NGSシーケンス解析ツール。BWAMEMベースのアライメント。Cpf1の解析にも対応している。 -
CRIS.py
:ゲノム編集後の変異NGSシーケンス解析ツール。アルゴリズムはCas-Analyzerと似たようなものだが、indicatorに該当するseq_start・seq_endは12-20 bpの間で定義できる。 -
CRISPRpic
:ゲノム編集後の変異NGSシーケンス解析ツール。アルゴリズムはCas-Analyzerと似たindicatorによったものだが、このツールでは切断位置からタイル状に複数のindicatorを定義しておいて変異状況を細かくスキャンできるようになっている。精度はCRISPRessoやCas-Analyzerより高い。 -
crispr-DART - [Python, R] - A workflow to analyze CRISPR-Cas-induced indel mutations in targeted/amplicon DNA sequencing. Can work with single/multiplexed sgRNAs per region(s).
:アンプリコンシーケンス解析ツール。sgRNAの切断効率等を算出可能。リッチなHTML形式のレポートを出力してくれる。論文:Froehlich et al., 2020 -
knock-knock
:ノックイン配列を対象にしたアンプリコンシーケンス解析ツール。イルミナの数百レベルのアンプリコンシーケンスデータだけでなくPacbio CCSの数千レベルのアンプリコンシーケンシングデータも解析できる。
Perl
-
ampliconDIVider
:ゲノム編集後の変異NGSシーケンス解析ツール。novoalignベースのアライメントでCRISPRおよび、TALEN、ZFNの解析が可能。分類精度はBWAMEMベースのCrispRVariantと同等という報告がある。 -
CRISPR-DAV
:ゲノム編集後の変異NGSシーケンス解析ツール。BWAMEMベースのアライメントに加えてABRAによるリアライメントで大規模なインデル等も検出できるようになっている。
パイプライン・プラットフォーム
-
BATCH-GE
:ゲノム編集後の変異NGSシーケンス解析ツール。複数サンプルを解析するのに向いている。BWAMEMベースのアライメント。Linux限定だが非公式(?)にdockerバージョンがあるのでMacOSX等でも利用可能。 -
CRISPRamplicon - BiooTools
:アンプリコンシーケンス解析ツール。CRISPRessoと似たような構成のアルゴリズムだがアライメントを二重で行っているようだ。(論文未発表、20200526現在)
NGSベースのオフターゲット解析
ゲノム編集したサンプルに含まれるオフターゲットを網羅的に調べるためのNGS手法がいくつか開発されており、それには専用のツールが必要となるものもある。
ウェブツール(1件)
-
Digenome-seq web tool
:WGSベースのオフターゲット解析手法「Digenome-seq」のデータを解析するためのツール。

コマンドラインツール(1件)
パイプライン・プラットフォーム
ナノポアシーケンサーを利用したアンプリコンシーケンシング解析
ナノポアシーケンサーは数十kbの長鎖DNAを一度に読むことが可能なシーケンサーとして近年注目を集めている。従来のNGSによるアンプリコンシーケンシングでは数百bp程度の範囲での変異検出しかできないため、オンターゲット/オフターゲット領域周辺(数kb)における広範な変異導入や大規模変異を検出することは簡単ではなかった。しかし、ナノポアシーケンサーを使って長鎖アンプリコンを読んだ場合は、こういった変異も同時に捉えることが原理上可能であるため、大きな期待が寄せられている。
ただし、2022年1月現在では、ONT社製のナノポアシーケンサーはNGSに比べるとシーケンスエラー率が非常に高いため、完全な上位互換技術とはなっていないことに注意したい。ツール面でもこの点を考慮した工夫がなされている。
コマンドラインツール(1件)
パイプライン・プラットフォーム
-
DAJIN
:Kuno et al.,2022に基づく機械学習および一連のアルゴリズムを利用してアレル分類を行い、ゲノム編集サンプルの遺伝型を定量するツール。点変異や構造変異、ノックアウト/ノックイン、逆位など様々な変異パターンの検出が可能。
論文:Kuno et al.,2022
プール型CRISPRスクリーニング解析
大量のCRISPRによって大規模ノックアウトスクリーニングを実施したサンプルの解析には、導入遺伝子の特定やクオリティ調整、データの正規化など専門的なバイオインフォ・統計解析が必要となる。これらを実行するためのツールがいくつか公開されている。
ウェブツール(2件)
-
CRISPRcloud
:インタラクティブに解析設定を行なって解析できるツール。

-
PinAPL-Py
:インタラクティブに解析設定を行なって解析できるツール。CRISPRcloudのようにウインドウ設定をいちいちする必要がないようになっている。処理プログラムの大半はpythonコードで書かれている。

-
VISPR-online
: MAGeCK、BAGEL、JACKSなど種々の解析アルゴリズムで解析した結果を可視化するツール。論文:Cui et al., 2021。

コマンドラインツール(21件)
R
-
gCrisprTools
:CRISPRスクリーニングデータを処理するためのツール -
CaRpools
:CRISPRスクリーニングデータを処理するツール -
bcSeq
:バーコードで紐つけられたCRISPRスクリーニングデータをマッピングするツール。高速処理がウリらしい。 -
PBNPA
:CRISPRスクリーニングデータを解析するツール。sgRNAラベルのpermutingによって有意な値をみつけだすといったノンパラメトリックなアルゴリズムを採用しているのが特徴。 -
ScreenBEAM
:CRISPRあるいはRNAiによるFunctional genomics(FG)スクリーニングデータを処理するためのツール -
MAGeCKFlute
:CRISPRスクリーニングデータに対して遺伝子機能解析を実施するためのツール。 -
CERES
:必須遺伝子を対象としたCRISPRスクリーニングデータに対して個々のsgRNAの効果を測定するツール。CRISPRスクリーニングではしばしばコピー数によるバイアスが問題となるが、このツールでは事前にコピー数情報をインプットすることでそのバイアスを抑えることができる。 -
CRISPRcleanR
:必須遺伝子を対象としたCRISPRスクリーニングデータに対して個々のsgRNAの効果を測定するツール。CRISPRスクリーニングではしばしばコピー数によるバイアスが問題となるが、このツールではゲノムをセグメント状に捉えて個々の領域での値の傾向を考慮することでそのバイアスを抑えている(コピー数情報の入力必要なし)。 -
drugZ
:薬剤に対する感受性を目的としたCRISPRスクリーニングデータを解析するためのツール。 -
GEMINI
:combinatorial CRISPRノックアウトスクリーニングデータを解析するためのツール。 -
gscreend
:モデルベースのCRISPRスクリーニング解析ツール。論文:Imkeller et al., 2020 -
Orthrus
:combinatorial CRISPRスクリーニング解析ツール。論文:Ward et al., 2021。 -
CoRe
:複数のCRISPRスクリーニングデータから組織やゲノムコンテキストに依存しないcore-fitness geneを探索するツール。論文:[Vinceti et al., 2021]。
python
-
BAGEL
:必須遺伝子を対象としたCRISPRスクリーニングデータに対して個々のsgRNAの効果を測定するツール。ベイジアンベースの推定という点に技術的な特異性がある。 -
pyCRISPRcleanR
:CRISPRcleanRのpython版。必須遺伝子を対象としたCRISPRスクリーニングデータに対して個々のsgRNAの効果を測定するツール。CRISPRスクリーニングではしばしばコピー数によるバイアスが問題となるが、このツールではゲノムをセグメント状に捉えて個々の領域での値の傾向を考慮することでそのバイアスを抑えている(コピー数情報の入力必要なし)。 -
JACKS
:必須遺伝子を対象としたCRISPRスクリーニングデータに対して個々のsgRNAの効果を測定するツール。同じsgRNAライブラリを使った際に算出されるsgRNAのスコアの再現性が高いのが特長。 -
casTLE
:CRISPRスクリーニングデータ解析ツール。RNAi・CRISPR KO・CRISPRi/aスクリーニングデータを解析でき、必須遺伝子の特定が可能。 -
STARS
:CRISPRスクリーニング解析ツール。二項分布を利用したランク付けを行う。 -
DSCN
:シングルターゲットのCRISPRスクリーニング解析結果等を参照して、有効な合成致死の組み合わせ等を予測するツール。論文:Liu et al., 2021。
JAVA
-
PoolQ
:バーコード配列を識別して、スクリーニング等でのDNAシーケンスデータをカウントするツール
matlab
HitSelect
:CRISPRスクリーニング解析ツール。論文:Diaz et al., 2014。
GENE-E拡張パッケージ
RIGER
:CRISPRスクリーニング解析ツール。RNAi Gene Enrichment Ranking (RIGER)という名前のとおり、元々はRNAi用の解析ツールだったが初期のCRISPRスクリーニング解析ではよく流用されていた。
パイプライン・プラットフォーム
-
MAGeCK
:CRISPRスクリーニングデータのQC処理等を実施するためのパイプライン。この分野においてはスタンダードなツールとなっている。 -
scMAGeCK
:シングルセルRNA-seqと組み合わせたプール型CRISPRスクリーニングデータ解析ツール。 -
MAGeCK-VISPR
:CRISPRスクリーニング解析ツール。論文:Li et al., 2015。
プール型CRISPRスクリーニング・シミュレーション
コマンドラインツール(1件)
Julia
-
CRISPulator
:CRISPRスクリーニングは画期的な手法だが、ちゃんと数値的に結果が出せるようにガイドライブラリのパラメータを設定するのは簡単ではない。このツールでは実際にシミュレーションを実施することで、ライブラリデザインのロバスト性を事前評価できる。
タイル型CRISPRスクリーニング解析
一般的にゲノムワイドな標的を対象とするプール型CRISPRスクリーニングが一般的だが、特定の遺伝子の機能解析などを目的としたタイル型CRISPRスクリーニングも実施されている。これ対する専用の解析ツールも発表されている。
コマンドラインツール(1件)
パイプライン・プラットフォーム
-
PASTMUS
:タイル型CRISPRスクリーニングで機能的ドメインを特定するための解析パイプライン。
RNA編集
Cas13に限らずRNA編集全般で必要な作業を行うツールも紹介する。
ウェブツール(1件)
-
PREP Suite
C->U編集を加えた際にアミノ酸が変化する領域をリストとして表示するツール。

CRISPRの探索
CRISPRのような核酸に作用する活性をもつリピート配列はドメインを超えた幅広い生物種でみられる。これらを同定するためのツールもいくつか公開されている。
ウェブツール(4件)
-
CRISPRCasFinder [online]
:CRISPR配列検索サイト。

-
CRISPRDetect
:CRISPR配列検索サイト。検索条件を細かく調整することができる。CRISPRCasFinderの旧版とは予測結果が大きく異なっていたが現版でどうなっているかは不明

-
HMMCAS
:hidden Markov modelsベースのCasタンパク配列検索ツール

-
CRISPRTarget
:CRISPR RNAsを予測するツール。論文:Biswas et al., 2013

-
CRISPRCasTyper
勾配ブースティングベースのCRISPR同定ツール。論文:Russel et al., 2020

-
CRISPRloci
CRISPR-Casの同定とアノテーションを行ってくれるツール。論文:Alkhnbashi et al., 2021

コマンドラインツール(8件)
C/C++
-
Crass
:メタゲノムデータから事前情報なしでCRISPRを同定するツール。Skennerton, Imelfort, and Tyson -
PILER-CR
:CRISPRリピートを同定するツール。Edgar, 2007
JAVA
-
CRT
:CRISPR探索ツール。論文:Bland et al., 2007
python
-
SPADE
:リピート配列検索ツール。CRISPRに限らずリピート配列はゲノム編集ツールで頻繁にみられる特徴であるため、そのような配列を検索することで新規ツールを見つけられる可能性がある。 -
CRISPRcasIdentifier
:CRISPR-Casの種類を判定するツール。判定には隠れマルコフモデルと機械学習モデルを利用している。 -
CRISPRdisco
:CRISPR arrayやCasタンパクを同定するツール。論文:Crawley, Henriksen, and Barrangou., 2018 -
metaCRISPR
:メタゲノムシーケンスデータからCRISPRを同定するツール。Lei and Yanni Sun., 2016 -
CRISPRleader
[Alkhnbashi et al., 2016]
:ゲノムデータからCRISPRを同定するツール。論文:Alkhnbashi et al., 2016
Perl
-
MetaCRAST
:メタゲノム情報を考慮しながらCRISPR arraysを探索できるツール。
R
-
Spacer2PAM
:CRISPR array spacerからPAM配列を予測するパッケージ。論文:Rybnicky et al., 2021
Anti-CRISPR(Acr)の探索
CRISPRは主にファージウイルスに対する防御機構だが、ファージウイルスはこれに対抗して、CRISPRを無力化するタンパク質(Anti-CRISPR)をもっている。これも様々なウイルス・生物種でみられており、同定ツールがいくつか発表されている。
ウェブツール(2件)
-
AcrFinder
:ホモロジーベース、guilt-by-association(GBA)ベース、self-targetingアプローチベースの3手法を組み合わせて抗CRISPRタンパク候補[Acrサブタイプを伴ったAcr-Acaオペロン])を検索するツール。タンパク・ゲノムの両方を入力として受け取ることができる。


コマンドラインツール(1件)
python
-
Anti-CRISPR
:*Anti-CRISPRタンパクを配列パターンから推定する機械学習モデルベースの判定ツール。
*Anti-CRISPRとは、CRISPRの阻害タンパクのこと。対ウイルス免疫の一つであるCRISPRであるが、これを阻害するタンパクも近年多く知られてきている。
CRISPR系統解析・マッピング・アノテーション
ウェブツール(2件)
-
CRISPRmap
:CRISPRに対する系統分類ツール。

-
CRISPRone
:CRISPR-Cas9に関わるエレメントのアノテーションをつけるツール。論文:Zhang and Ye

-
CRISPRcompar - CRISPRdb
:CRISPR配列の比較を行うツール。論文:Grissa, Vergnaud ,and Pourcel

遺伝子機能解析
CRISPRスクリーニング実験の中には、標的のより込み入った表現型・相互作用を推定する方法もある。そういった実験のための解析ツールもいくつか報告されている。
ウェブツール(1件)
-
NEST
:プール型CRISPRスクリーニングのデータを利用して必須遺伝子を特定するツール。

コマンドラインツール(1件)
python
CRaftID
マイクロラフト (microraft)技術とイメージングを伴うプール型CRISPRスクリーニングのデータを解析し、RNA結合タンパク質 (RBP) の機能同定を行うツール。
ゲノム編集に基づいたゲノム機能予測
コマンドラインツール(1件)
R
CCS
:Niu et al., 2020に基づいたcritical enhancer regions予測ツール。
データベース(20件)
-
UCSC genome browser
:UCSC genome browserではCRISPR-Cas9のターゲットをオン・オフターゲット予測結果を含めて表示できる。

-
grID
:sgRNAのデータベース。エキソン、エントロン、UTR、プロモータを分けて表示することができる。論文:Jaskula-Ranga and Zack., 2016

-
Cas-Database - RGEN Tools
:Park, Kim, and Bae., 2016に基づいて作製したCas9標的データベース。オフターゲットやエキソン領域などといったの絞り込みも可能。

-
dbGuide
:1,000を超えるヒトとマウスの研究データで利用されたsgRNA標的とその効率のデータベース。論文:Gooden et al., 2020

-
CRISPRz
:NHGRI/NIHのラボ等で利用実績のあるsgRNA標的のデータベース。ゼブラフィッシュのものが中心的。論文:Varshney et al., 2016

-
Cpf1-Database - RGEN Tools
:Park and Bae., 2018に基づいて作製されたCpf1標的データベース。Cas-Databaseと同様の機能をもつ。 -
MHcut Browser
:小さな相同配列を介したDSB修復経路であるA-EJやSSA(MMEJと総称されることも多い)でSNPの再現が可能な領域のデータベース。変異予測スコアもついている。ClinVarに登録されている領域のみを表示することもでき、治療標的の検討にも役立つものとなっている。

-
CRISPRlnc
:lncRNA領域をターゲティングするsgRNAのデータベース。論文:Chen et al., 2019

-
CRISPRdb
:CRISPRFinderの検索結果に基づいて作製されたCRISPR配列データベース

-
CRISPRCasdb
:CRISPRFinderを基軸とした検索によって作製されたCRISPR配列とCas9遺伝子のデータベース

-
Anti-CRISPRdb
:CRISPR-Casシステムを阻害するタンパク質(Anti-CRISPR)のデータベース。論文:Dong et al, 2018

-
CRISPI
:CRISPR特有のリピート構造を指標として作製されたCRISPR配列データベース。論文:Rousseau et al., 2009

-
CasPDB
:HMMCASとCRISPRFinderの検索結果に基づいて作製されたCasタンパクのデータベース。

-
BioGRID Open Repository of CRISPR Screens (ORCS)
:CRISPRスクリーニングライブラリのデータベース。遺伝子名を入力するとその遺伝子を含むライブラリが表示される。登録情報は論文情報だけなく細胞腫や使用した解析ツール、ランクまで記録されており、一目で大体どういうスクリーニング結果だったのか把握できるようになっている。

-
DepMap
:がん細胞株に関する遺伝子情報をまとめたデータベース。Broad InstituteとSanger Instituteが実際に実験を行ってデータベースを構築している。遺伝子名を入力すると、非必須遺伝子かどうか、細胞株ごとにみられる変異、発現、コピー数、遺伝子の概要といった内容が表示され、CRISPRスクリーニングでの結果やRNAi実験の情報なども参照可能。

-
CRISPR atlas
:Xiang et al., 2020に基づいたCas9, ABE, CBEの変異パターンデータベース

-
Project Score (Sanger DepMap)
:CRISPRスクリーニングのデータベース。薬剤標的にできるがん関連遺伝子にフォーカスしている。論文:Behan et al.,2019

-
PICKLES
:CRISPRスクリーニングのデータベース。全体的に見やすく、正規化された値等を変更して表示できる。論文:Lenoir, Lim, and Hart, 2017

-
CRISP-View
:CRISPRスクリーニング・CRISPRスクリーニングa/i・RNAiスクリーニング結果のデータベース。論文:Li et al., 2020

-
COMBAT crRNA Repository(提供元:Stanley Qi Lab, スタンフォード大学, アメリカ🇺🇸)
RNAウイルスゲノムに対するCRISPR-Cas13 crRNAデータベース。論文:Lin et al., 2021

-
The Genome Targeting Catalogue
:欧州バイオインフォマティクス研究所(EMBL-EBI)によるガイドRNA標的データベース。過去に使用された標的に関して、その時の実験手法や結果を詳細に記録できる仕様となっている。APIも利用可能。*現在準備中のためか詳細に関する記載はなし。

統合プラットフォーム(2件)
-
CRISPR-GE
:ガイドデザインやオフターゲット予測、TIDE解析等が実施できる。

-
Benchling
:ガイドのデザインを含めた基本的な分子生物学実験をサポートしている。
その他CRISPR関連ツール
ウェブツール(1件)
-
CReVIS-Seq - RGEN Tools
:CRISPR実験でもよく使うレンチウイルスベクター導入に関して、そのインテグレーションサイトを解析する実験手法CReVIS-Seqのシーケンスデータを解析するツール。(論文未公表:2020/05/25現在)

コマンドラインツール(4件)
Scala
-
Cas9FateMapping
CRISPRを利用したlineage tracing法(GESTALT法)を解析するためのツールセット
R
-
CSESA
:CRISPR配列からSalmonella entericaのSerotypeを予測するツール。チフス菌等を含むSalmonella entericaは食中毒の原因として世界的に有名な菌種となっているためそのSerotypeの同定は重要とされている。CRISPR配列はそのためのマーカーとして利用価値が高いようだ。ゲノム編集と関係がない観点のツールだが一応紹介しておく。 -
MAUDE
:分布の違いを検出するツール。CRISPRiスクリーニングの評価をFACSで行うような実験での利用を想定。論文:Boer et al., 2020
Python
-
CRISPRbuilder-TB
WGSデータから結核菌(Mycobacterium tuberculosis)CRISPR locusのreconstructionを行う。論文:Guyeux et al., 2021
その他情報検索(3件)
-
PubMed
:生物系論文検索 -
Google Scholar
:論文検索 -
UCSC Genome Browser
:ゲノムブラウザ、もはや必須
他リンク集(12件)
以下はバイオインフォやコーディングに関する雑多なリンクです。
ツールに関するレビュー記事
総合
NGS解析
マニュアル
-
CrispRVariants
:ゲノム編集用NGS解析ソフト。すごく便利なんだけどちょっとBioconductorに習熟してないときつい・・・ -
ggplot2
:グラフ描写Rパッケージ。生物系の論文では最近よく使われているのを見るのでここで紹介。
ggplot2はかなり独特な仕様なので、下手にブログ記事をあさるよりここの公式マニュアルを見た方が答えがみつかりやすい。
新着論文情報
- crisp_bio(http://crisp-bio.blog.jp)
:ゲノム編集関連の新着論文を日本語(時々英語)解説とともに紹介している。更新頻度や情報の網羅度がすごい。。
ツイッター版もある(ID:@ScienceCrisp)。
ブログ
その他
-
Anaconda
:環境構築はAnaconda(Miniconda)の仮想環境内で行うのが私のオススメ。現状Pythonソフトウェアかどうかを問わずほとんどのものが揃っている。
※解析にある程度慣れている方はDockerやCWLを利用するほうがいいかもしれません。 -
OmicX
:バイオインフォ系のツールリンク集。かなり広範にカバーしている。ツール検索機能が一時期有料化されていたが無料に戻ったようだ。(閉鎖するらしい(2020年5月)) -
Biostars
:英語圏のバイオインフォBBS。やや初心者にはキツイ感があるがトラブル時にはここをあさるとよいかも。でも最近は未回答多いような気も。。。 -
Stackoverflow
:英語圏の情報系BBS。コーディングで遭遇する有名なエラーは大体載っている。
あと最後に私が書いているブログ記事も掲載しておきます。
自ブログ記事一覧(更新中)
環境設定関係
配列デザイン関係
NGS解析関係
- BWA-MEMを使ったマッピング
- Trimmomaticを使ったアダプタートリミング
- CrispRVariantsを使ったCRISPR-Cas9編集サンプルのアライメント
- Cas-Analyzerを使ったCRISPR-Cas9編集サンプルの評価
- CRISPResso(CUI版)を使ったCRISPR-Cas9編集サンプルの評価
おわりに
ゲノム編集のような新規実験分野では実験手法が多様化していくので中々in silico側での定法が定まらない傾向にあると思います。
ある程度理解ある人なら自分で取捨選択すればいいんですが、ウェットの実験を中心に仕事している人にとっては学びづらいことこの上ないので、実験研究者・インフォ初学者向けに何かしら実例を発信していければいいかと考えているところです。