ゲノム位置の特定をネットを介さずに一瞬で済ませる方法について説明します。
Blat の導入
まずはじめにゲノム検索に用いるBlat というプログラムを用意します。
ダウンロードはhomebrewをインストール済みであれば、以下の命令をbashに叩いてください。
(homebrewのインストールはこちら)
brew tap homebrew/science
brew install blat
blatのインストールが完了したら以下のコマンドでヘルプが見れます。
blat
この中のusageをみてください。
usage:
blat database query [-ooc=11.ooc] output.psl
blat ではウェブにつながず、ローカル上のファイルをデータベースとして利用します。
blat データベースファイル 検索する配列が入ったファイル 結果出力先ファイル
hg19.fa の取得
では次にデータベースを用意しましょう。
今回はヒトゲノムのリファレンスとして一般的なhg19 をデータベースとして使います。
以下のアドレスをウェブブラウザでアクセスしてください。
hgdownload.cse.ucsc.edu/goldenPath/hg19/bigZips/chromFa.tar.gz
唐突に905MBのファイルがインストールされます。しばらく待ちましょう。
ダウンロードできたら、作業をしたいディレクトリへファイルを移動します。
以下のコマンドでファイルを展開して、hg19.faを作成します。
tar -zxvf chromFa.tar.gz;
cat chr*.fa > ./hg19.fa;
6GBくらい食われるので注意してください。
hg19.fa ができたら残りは捨てて構いません。
これでデータベースファイルが取得できました。
fastaファイルにはいった配列のゲノム上の位置を配列を検索する
今回検索する配列はこちらです。
>1
CAGCCAACAGTGGATATTCC
>2
AGAACTACGTGGAAGTGACC
>3
GAGCTGCGCGCGGGGCCACA
ではhg19.fa があるディレクトリ上で検索しましょう。
blat hg19.fa test.fa -minMatch=0 -minScore=20 output.psl
-minScore=20 は検索配列が20塩基ということに由来します。
これに関しては各自書き換えてください。
しばらく待つと以下の表示がでます。
Searched 60 bases in 3 sequences
テキストエディタでoutput.pslは以下のようになっているはずです。
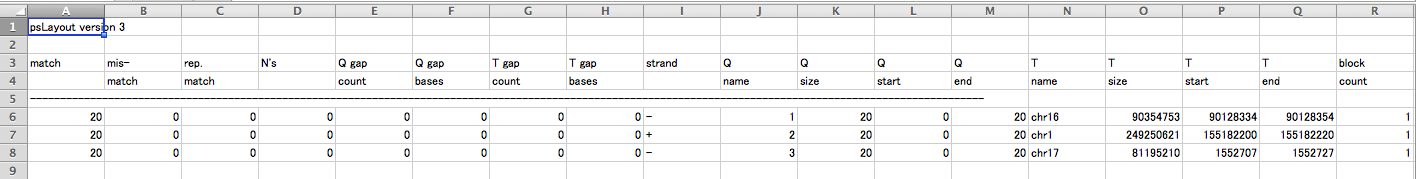
psLayout version 3
match mis- rep. N's Q gap Q gap T gap T gap strand Q Q Q Q T T T T block blockSizes qStarts tStarts
match match count bases count bases name size start end name size start end count
---------------------------------------------------------------------------------------------------------------------------------------------------------------
20 0 0 0 0 0 0 0 - 1 20 0 20 chr16 90354753 90128334 90128354 1 20, 0, 90128334,
20 0 0 0 0 0 0 0 + 2 20 0 20 chr1 249250621 155182200 155182220 1 20, 0, 155182200,
20 0 0 0 0 0 0 0 - 3 20 0 20 chr17 81195210 1552707 1552727 1 20, 0, 1552707,
T欄の
nameが染色体番号
start,endはゲノム上の位置となります。
これで位置が定まりました。
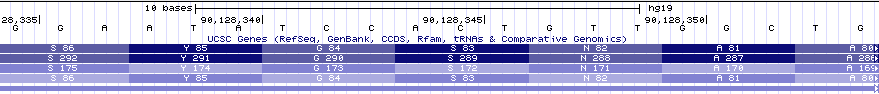
念のためtest.faの番号1の配列(CAGCCAACAGTGGATATTCC)をUCSCのgenome browserでも確認してみましょう。
strandがマイナス(-)なので逆相補鎖が表示されていますが、間違いありませんね。
この方法で1000個程度の配列も一気に検索できます。
驚くべきことにその所要時間は先ほどの場合と大差ありません。
これならblast の前で腕を組む生活にはさよならできそうです。
また、補足として
blat hg19.fa test.fa -minMatch=0 -minScore=20 -noHead output.psl
とすればヘッダーがない状態で保存されます。
Rに直接データフレームとして読み込むときはこの形式がおすすめです。
以上です。ありがとうございました。