この投稿の趣旨は簡単な生物学的現象や分子生物学実験を UNIX コマンド上で再現することです。生物学に馴染みのない方はまず以下のページを見ていただくと良いかもしれません。
ランダムな塩基配列を生成
生物の遺伝情報は DNA 分子中のアデニン・チミン・グアニン・シトシンの4つの塩基の配列としてコードされています。それぞれが A、T、G、C の4文字に割り当てられているものとして、次のコマンドでランダムな塩基配列が生成され標準出力に書き出されます。
# DNA
$ cat /dev/urandom | base64 | tr -dc 'ATGC'
# RNA
$ cat /dev/urandom | base64 | tr -dc 'AUGC'
終了するときは ctrl + C でキャンセルしてください。
コマンドの詳細
/dev/urandom
乱数生成器です。
cat /dev/urandom を実行すると標準出力でランダムなビット列が得られます。
base64
パイプ元から得られたランダムなビット列を base64 でエンコードします。
tr -dc 'ATGC'
ATGC 以外の文字を削除します。 チミンの代わりにウラシルが含まれれる RNA の場合は T を U に変えると対応できます。
転写
DNA は RNA ポリメラーゼの働きで RNA に転写されます。rna-polymerase-stream というライブラリを使ってこの働きを再現してみます。このライブラリでは Node.js の TransformStream のインターフェースを実装して、標準入力された ATGC の文字列に対して A -> U、 T -> A、 G -> C、 C -> G の変換を行います。
$ cat /dev/urandom | base64 | tr -dc 'ATGC' | \
npx rna-polymerase-stream
翻訳
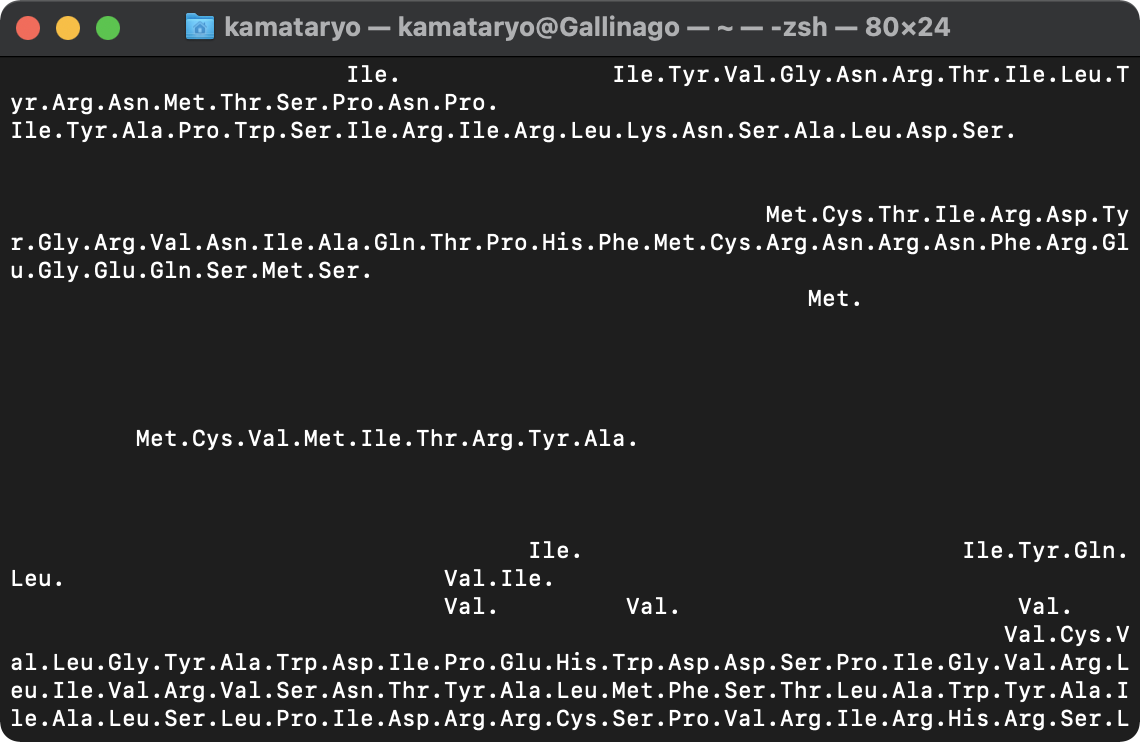
mRNA 中の3文字の塩基配列の組み合わせ(コドン)はそれぞれ20種類のアミノ酸に対応していて、この順序に従ってアミノ酸が重合することで様々なタンパク質になります。この塩基配列を元にタンパク質を合成する仕組みを翻訳といい、細胞内にあるリボソームで行われます。
次のライブラリはリボソームの働きを模し、コドン表に基づいて RNA の塩基配列をタンパク質に翻訳します。イニシエーター配列(AUA、AUG、GUG)とターミネーター配列(UAA、UAG、UGA)を認識してその間の塩基配列をタンパク質に翻訳し、 Arg.(アルギニン) などの記号に置換します。
$ cat /dev/urandom | base64 | tr -dc 'ATGC' | \
npx rna-polymerase-stream | \
npx ribosome-stream
制限酵素
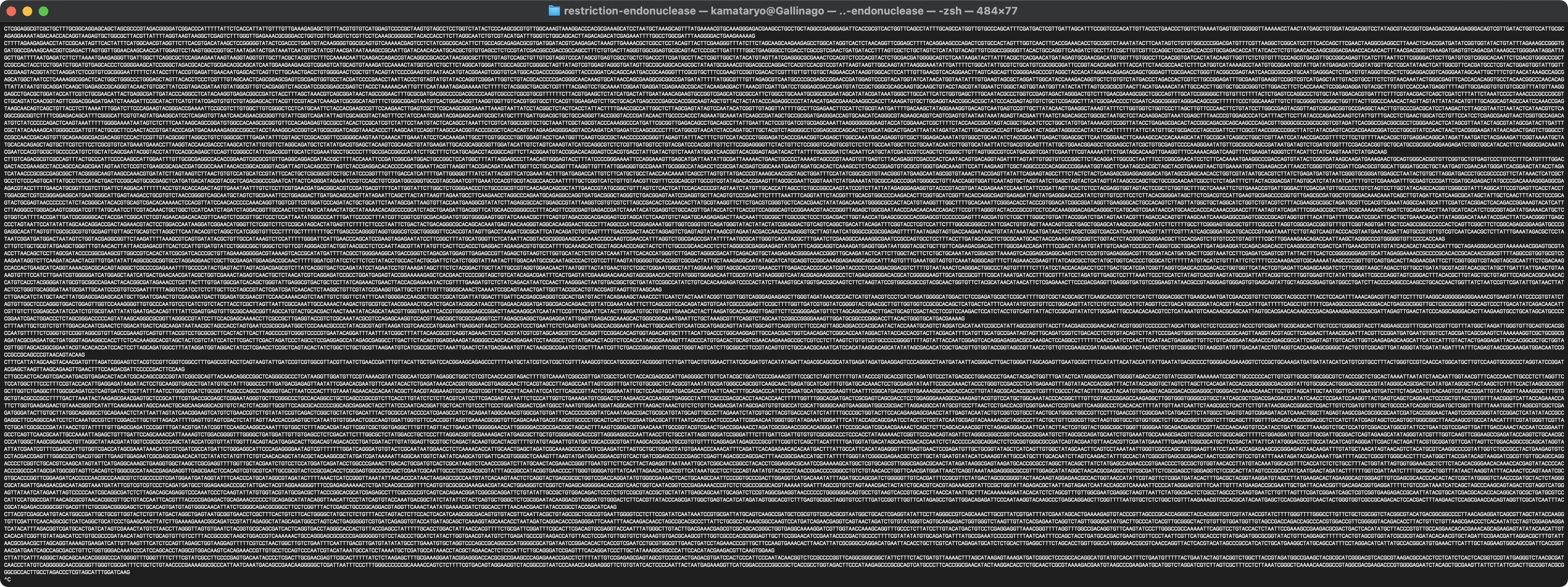
制限酵素の働きを模して特定の塩基配列を認識し切断します。回文配列が対象です。
例: AAGCTT GATATC
制限酵素で切断された DNA 断片は改行区切り文字列としてストリームに流します。
$ cat /dev/urandom | base64 | tr -dc 'ATGC' | \
npx restriction-endonuclease AAGCTT
電気泳動
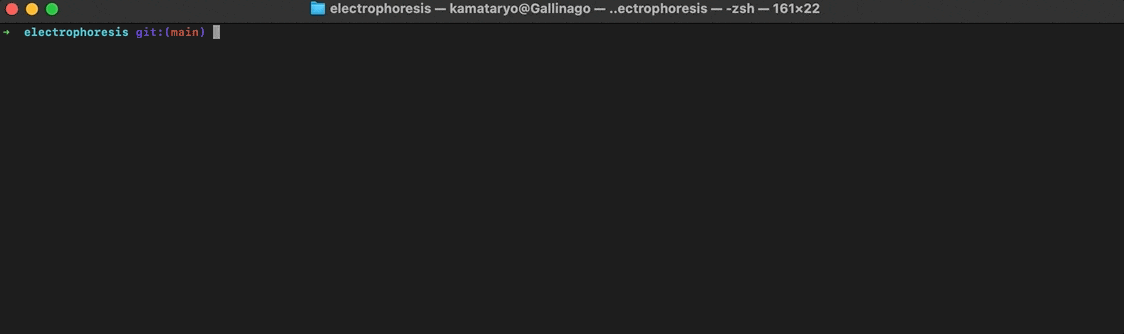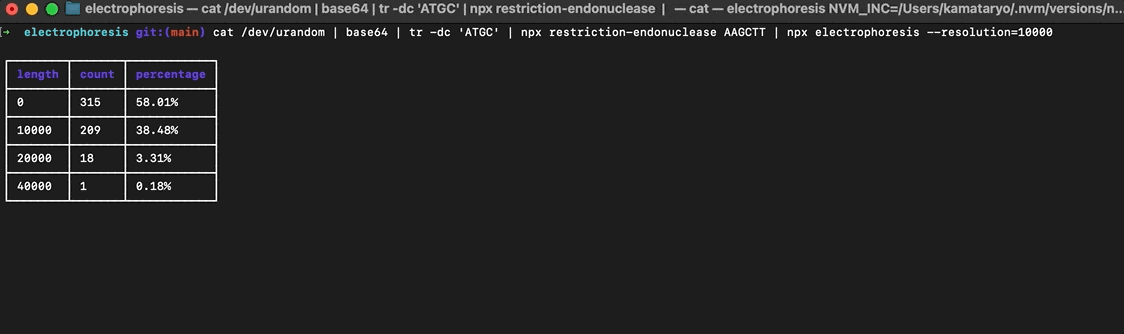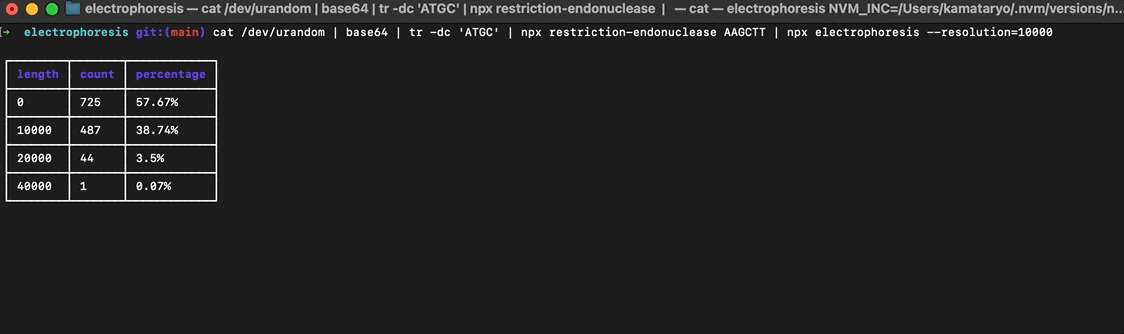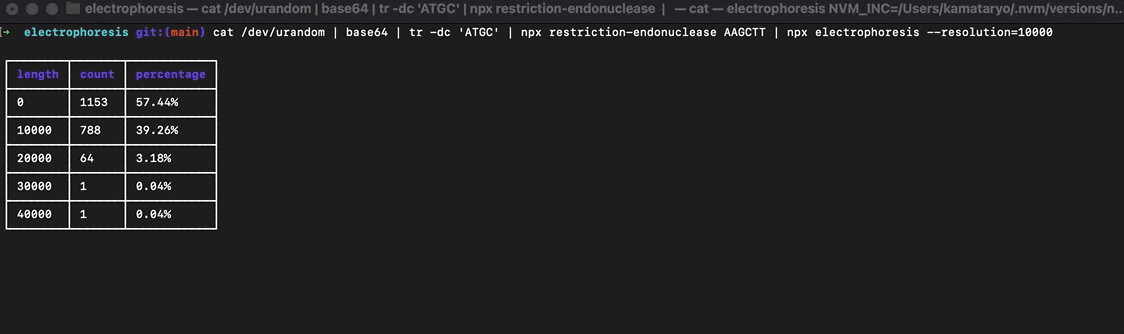
DNA 断片が得られたら最後は電気泳動です。DNA 断片の重さ = 文字数の長さとしてカウントしヒストグラムを作成します。以下のライブラリを使います。 --resolution オプションはヒストグラムのステップ間隔で小さい値を指定するとヒストグラムが細かくなります。
$ cat /dev/urandom | base64 | tr -dc 'ATGC' | \
npx restriction-endonuclease AAGCTT | \
npx electrophoresis --resolution=10000
そのほかに CLI で再現できそうなおもしろい生物学的現象や実験方法などがあればぜひコメントで教えてください。