初めに
私は感染症の専門家ではありませんので、理解した上で読んでください。
コロナウィルスの感染者数が毎日増加しています。
厚生労働省のページには、使いやすいオープンデータがありませんが、オープンデータポータルサイトでデータを公開している県があります。PCR検査数と感染者数のデータがあるので、感染者数の推移を見てみました。
1週間後のデータでグラフを更新しましたが、先週より感染率が低下していることが読み取れます。
- 更新履歴
2020/4/19 : 初回投稿
2020/4/27 : 2020/4/26のデータでグラフを更新
- データ
日本全体のPCR検査人数と感染者数のデータは、厚生労働省がWEBで公開されているのですが、WEBでの表示で使いやすいCSV形式などの数値データになっていません。
一方、各県ごとのデータはCSV形式で公開されている県があります。
埼玉県:https://saitama.stopcovid19.jp/
埼玉県のデータが使いやすいので、埼玉県のPCR検査数と感染者数から、感染率の推移を調べました。ただし埼玉県のデータは検査人数ではなく、検査数(延べ人数)と書かれているので、
感染率=感染者数/検査数(延べ人数)
を計算しています。
県ごとのデータフォーマットが統一されていたら、他の県もすぐに調べられるのですが、残念ながら統一されていませんでした。
感染率の推移
- パッケージ読み込み
import pandas as pd
import matplotlib.pyplot as plt
- データ読み込み
data = pd.read_csv('data/saitama_kensa_20200418.csv')
data.columns = ['day', 'PCR', 'Confirmed']
print(data.head())
print(data.tail())
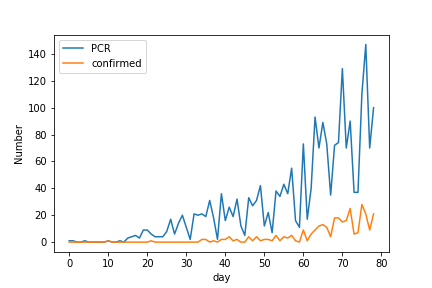
PCR検査数のデータは、2020/1/31から2020/4/18のデータです。
day PCR Confirmed
0 2020/1/31 1 0
1 2020/2/1 1 0
2 2020/2/2 0 0
3 2020/2/3 0 0
4 2020/2/4 1 0
day PCR Confirmed
74 2020/4/14 37 7
75 2020/4/15 110 28
76 2020/4/16 147 21
77 2020/4/17 70 9
78 2020/4/18 100 21
- PCR検査数と感染者数の表示
plt.plot(data.loc[:,'PCR'])
plt.plot(data.loc[:,'Confirmed'])
plt.xlabel('day')
plt.ylabel('Number')
plt.legend()
plt.savefig('fig1.png')
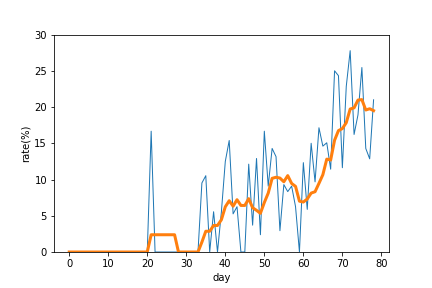
- 感染率の推移
日々の感染率は大きく変動しているので、7日間の移動平均も表示します。
移動平均は最初の50日間は10%以下でしたが、その後上昇し20%前後になっています。
rate = data.loc[:,'Confirmed']/data.loc[:,'PCR'].fillna(0.0)
rate_ma = rate.rolling(7).mean().fillna(0)
plt.plot(rate * 100, label='rate', linewidth=1)
plt.plot(rate_ma * 100, label='rate_ma', linewidth=3)
plt.ylim(0.0, 30.0)
plt.xlabel('day')
plt.ylabel('Rate(%)')
plt.legend()
plt.savefig('fig2.png')
2020/4/19投稿
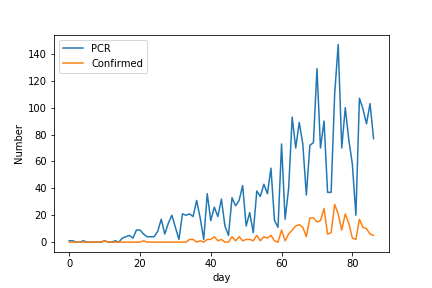
2020/4/27投稿
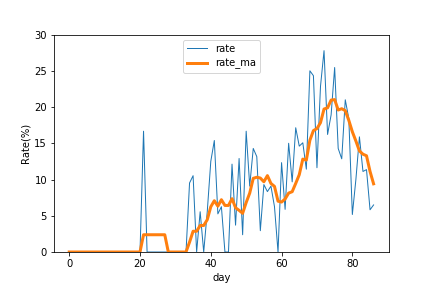
2020/4/19のデータでは感染率の移動平均が20%前後ありました。
しかし、2020/4/26のデータでは10%前後に低下してきています。
外出自粛により、感染者数が減ることを期待しています。