目的
2品種間の交雑に由来する遺伝解析材料において、親品種のWhole genome resequenceデータから、PCRベースのジェノタイピングに使えるSNP・InDel変異とそれらをPCR増幅するためのプライマーセットを自動で網羅的に作成できるツール「mkdesigner」を開発した。これをBiocondaに登録し、
conda install mkdesigner
でインストールできるようにしたい。
ディレクトリ構造
├── LICENSE
├── MANIFEST.in
├── README.md
├── build/...
├── dist/...
├── mkdesigner
│ ├── __init__.py
│ ├── __pycache__/...
│ ├── addprimertovcf.py
│ ├── haplocall.py
│ ├── mergevcf.py
│ ├── mkprimer.py
│ ├── mkselect.py
│ ├── mkvcf.py
│ ├── mvinputfiles.py
│ ├── params.py
│ ├── refindex.py
│ ├── selectsnp.py
│ ├── utils.py
│ └── visualize_marker.R
├── mkdesigner.egg-info/...
├── setup.py
└── test/...
PyPIへの登録
基本的に以下の記事を参考に行った。
rm -f -r mkdesigner.egg-info/* dist/*
python setup.py sdist bdist_wheel
twine upload --repository pypi dist/*
2行目で、
python setup.py sdist
python setup.py bdist_wheel
と分けるとなぜかエラーが出たが、合わせると問題なかった。
biocondaへの登録
基本的に以下の記事を参考に行った。
bioconda-recipesを自分のレポジトリにフォークする
自分のアカウントでgithubにログインした状態で、
https://github.com/bioconda/bioconda-recipes
にアクセスし、右上から、Fork>Create new forkをクリックし自分のレポジトリにフォークした。
bioconda-recipesをダウンロード
cd ~
git clone https://github.com/KChigira/bioconda-recipes.git
cd bioconda-recipes
git remote add upstream https://github.com/bioconda/bioconda-recipes.git
後で最新版に更新する場合は下の手順を行う(未確認)
cd ~/bioconda-recipes
git checkout master
git pull upstream master
パッケージを作成するブランチの作成
git checkout -b <package name>
Switched to a new branch 'mkdesigner'
CondaレシピをGrayskullを利用して作成
conda install -c conda-forge grayskull
cd ~/bioconda-recipes/recipes
grayskull pypi mkdesigner
実行後、
~/bioconda-recipes/recipesディレクトリ内に
/mkdesigner/meta.yamlが生成したことを確認した。
Condaレシピテンプレート(meta.yaml)の設定
meta.yamlを開いて確認した。
参考記事では、レシピ管理者設定項目を自分のGitHub IDへ書き換える必要があるとされていたが、今回はすでに自分のIDが書かれていた。
extra:
recipe-maintainers:
- KChigira
次に依存する外部パッケージの記述を追加した。
原則開発環境のバージョン以上とし、Pythonのみ3.11だったが、3.8以上とした。
requirements:
host:
- python
- pip
run:
- python >=3.8,<4.0
- pandas >=2.0.2,<3.0.0
- samtools >=1.6,<2.0
- bcftools >=1.5,<2.0
- blast >=2.14.0,<3.0.0
- gatk4 >=4.4.0.0,<5.0.0.0
- picard >=2.18.29,<3.0.0
- r-base >=4.2.3, <5.0.0
登録
まずは自分のリポジトリにあるbioconda-recipesにプッシュする。
cd ~/bioconda-recipes/recipes
git add mkdesigner
git commit -m "Add mkdesigner"
git push --set-upstream origin mkdesigner
ここで再びhttps://github.com/bioconda/bioconda-recipes
に行くと、ページ上部に
KChigira:mkdesigner had recent pushes less than a minute ago
と表示されていた。
Compare & Pull requestボタンをクリック、続くページでそのままPull requestを押下した。
すると、自動で審査が開始した。
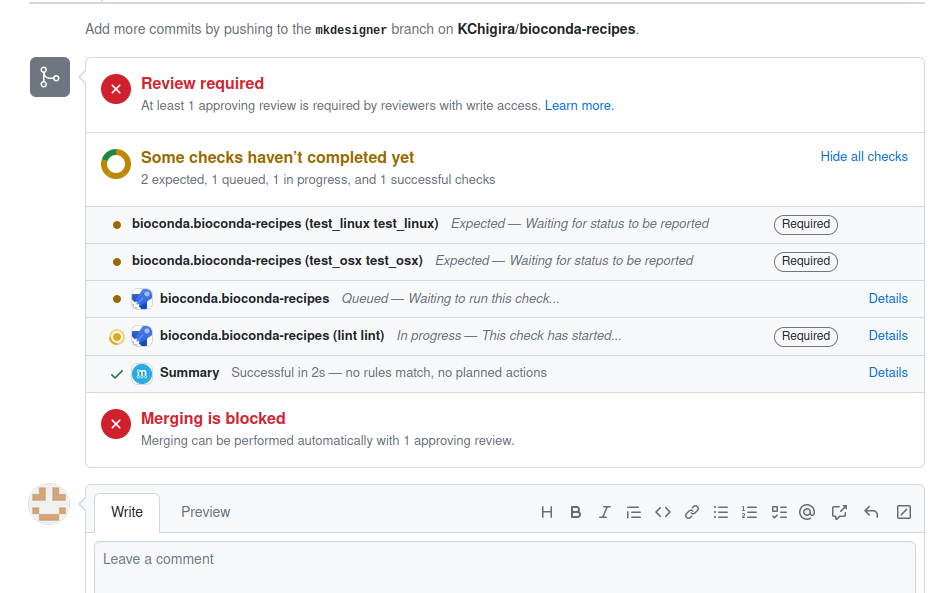
https://github.com/bioconda/bioconda-recipes/pulls
から進行状況を確認できる。
指示通り、"@BiocondaBot please add label"とコメントを入れるとラベルがついた。
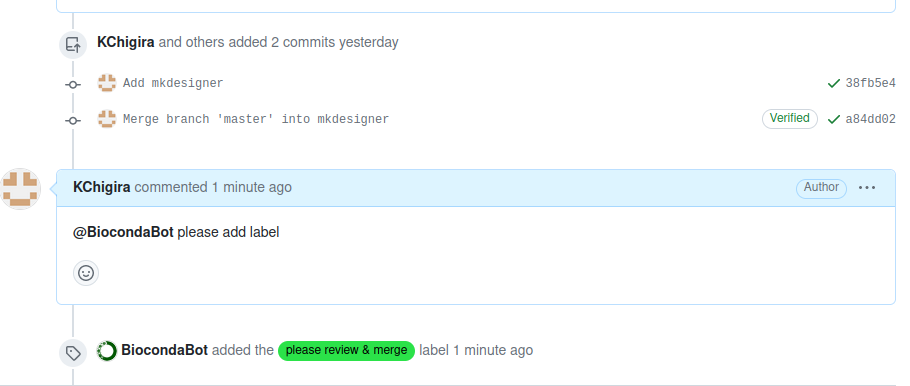
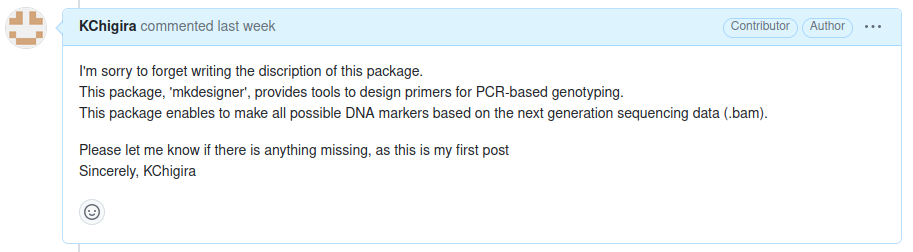
しかし、1週間過ぎても反応がなく、よく確認すると、プログラムの内容を書いておく必要があった。
そうすると2日後にレビュアーから返信があり、pull requestが承認された。
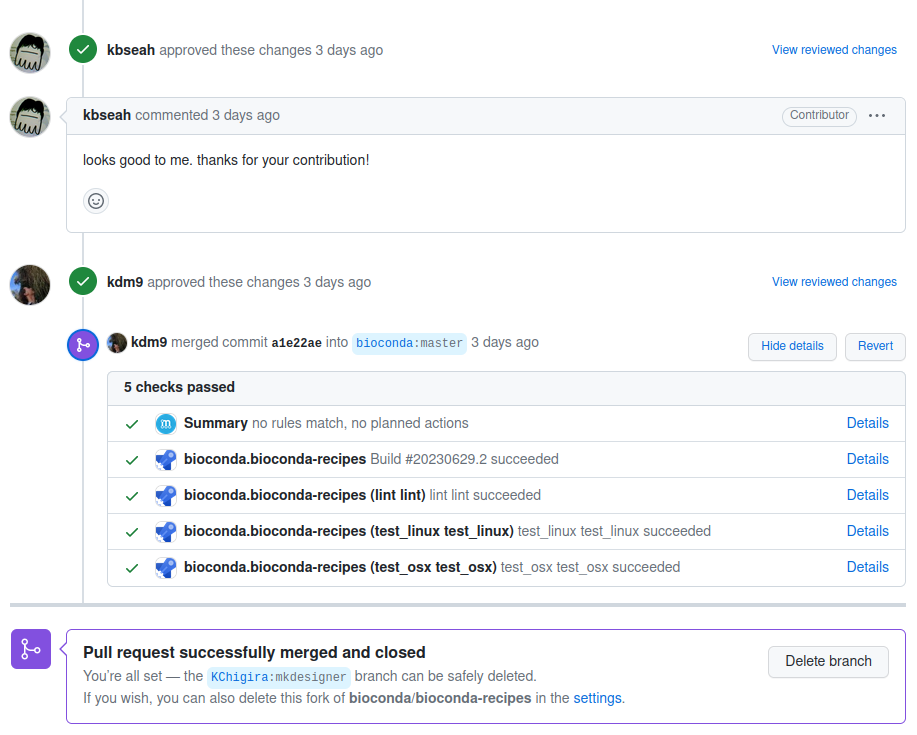
テスト
conda install -c bioconda mkdesigner
依存パッケージが多すぎて時間がかかったが、インストールできることを確認した。
bioconda 更新
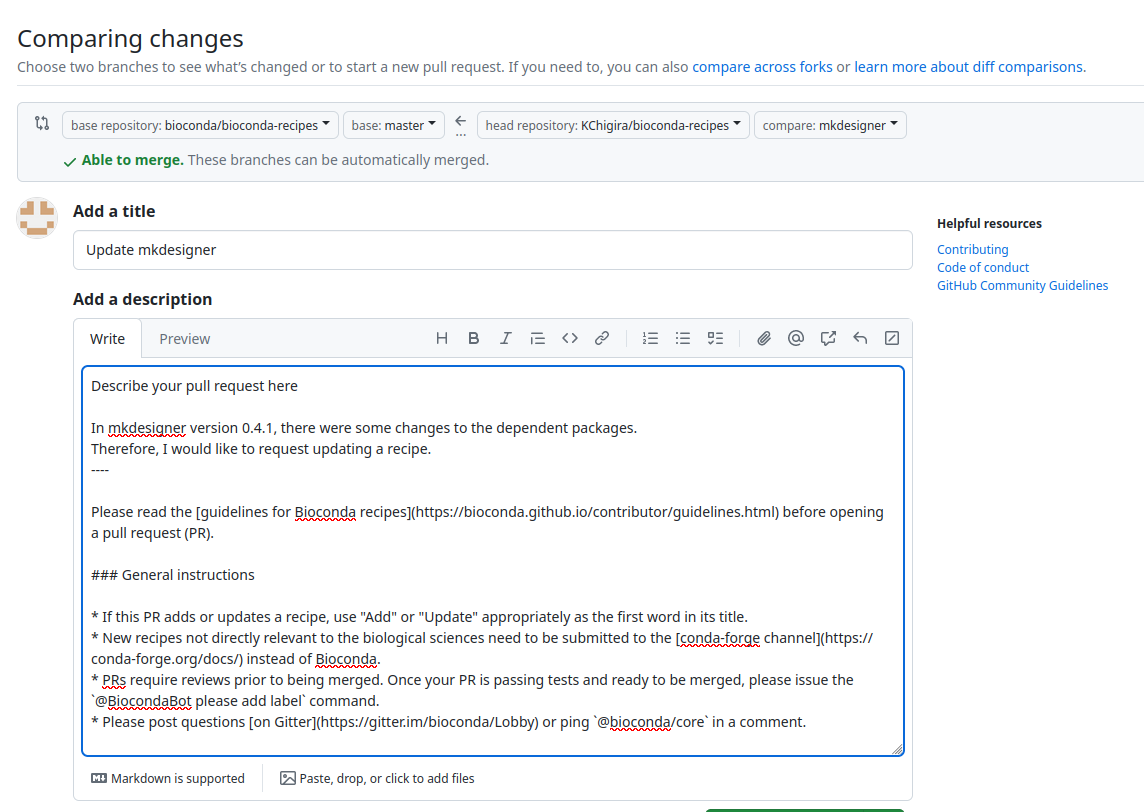
2024年6月26日追記
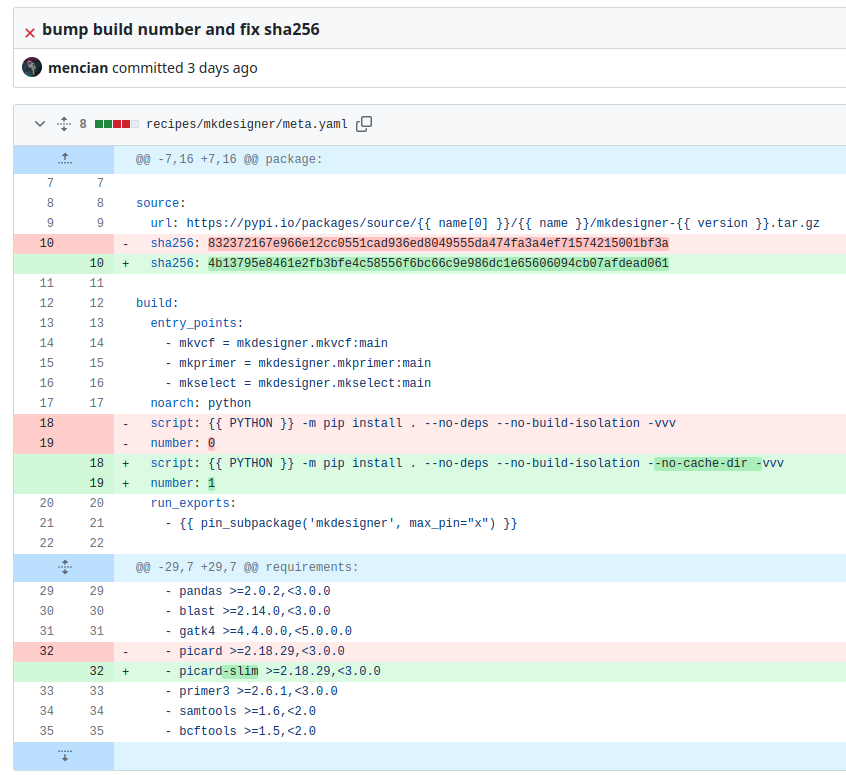
依存するプログラムに変更があったのでレシピを更新する。
bioconda-recipesのフォークの更新
一度フォークしたbioconda-recipesは、
右上のSync forkで更新できる。
bioconda-recipesを改めてダウンロード
今回、前回からPCが変わっていたので、改めてダウンロード。
以前ダウンロードしたものがあれば更新で対応できるはず(前述)
cd ~
git clone https://github.com/KChigira/bioconda-recipes.git
cd bioconda-recipes
git remote add upstream https://github.com/bioconda/bioconda-recipes.git
パッケージを作成するブランチの作成
git checkout -b mkdesigner
今回はすでに
~/bioconda-recipes/recipes/mkdesigner/meta.yamlがある。
Condaレシピテンプレート(meta.yaml)を更新
余計なところはいじらず、依存パッケージのリストとバージョンのみ更新
requirements:
host:
- python
- pip
run:
- python >=3.8,<4.0
- pandas >=2.0.2,<3.0.0
- blast >=2.14.0,<3.0.0
- primer3 >=2.6.1,<3.0.0
- gatk4 >=4.4.0.0,<5.0.0.0
- picard >=2.18.29,<3.0.0
- samtools >=1.6,<2.0
- bcftools >=1.5,<2.0
自分のリポジトリのbioconda-recipesにプッシュ。
cd ~/bioconda-recipes/recipes
git add mkdesigner
git commit -m "Update mkdesigner"
git push --set-upstream origin mkdesigner
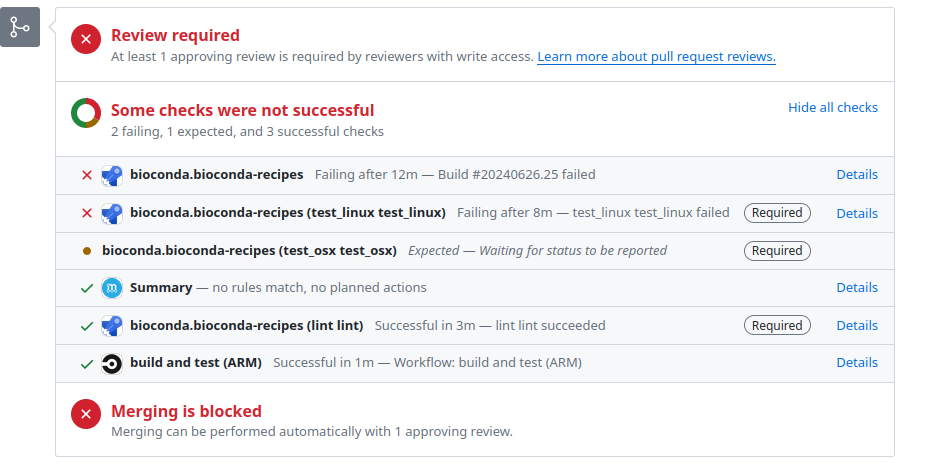
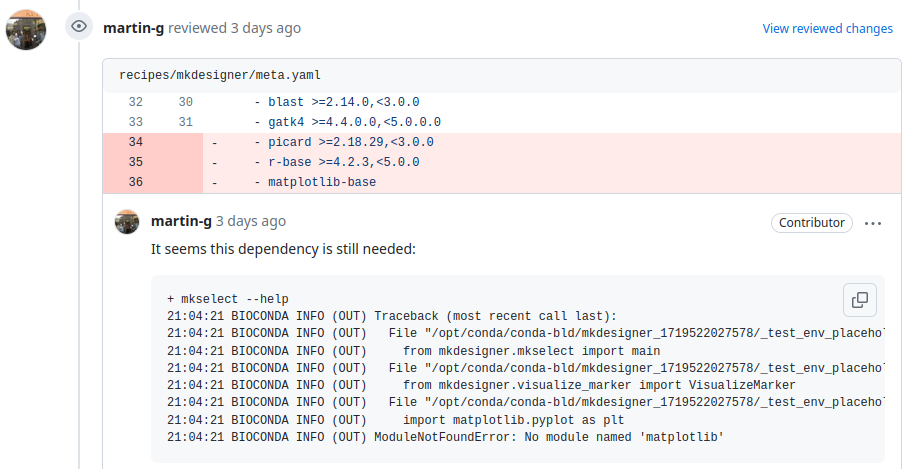
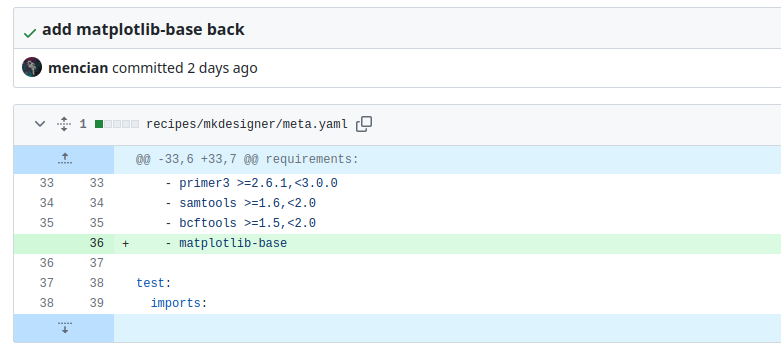
どうしようかと考えている間に、レビュアーの方が原因を特定し解決策を提示してくれた。
おそらく、プログラムのハッシュ値が前のバージョンのものになっていたのが原因。
あと、picardがpicard-slimに変更された。調べると、Rに依存するコマンドを抜いて、軽量化したバージョンらしい。今回依存パッケージからRを抜いたので、こっちでいいだろうということか。