はじめに
近年における細胞のセグメンテーションには,主に学習ベースであるMask-RCNNが使用されてきました.しかし,細胞が重なっている場合において,全ての細胞を正しく検出することが困難であるといった問題がありました.そこで,StarDistというアルゴリズムが開発されました.StarDistとは,星型凸多角形(Star-convex)を用いた細胞核のセグメンテーションを行うための手法です.
StarDistはGithubにて論文やコードがまとめられており,チュートリアルが充実しています.特に,学習やテストだけでなく,学習済みモデルの使用も可能なため,気軽に試すことができます.しかし,英語の説明が中心であり日本語での説明が全くないため,本記事で大まかにまとめてみることにしました.
Stardistの仕組み
StarDistは,バウンディングボックスに比べ優れた形状表現である星型凸多角形を使用することによる細胞核のセグメンテーション手法です.モデルはU-Netに基づく軽量なニューラルネットワークを使用します.モデルを使用し,オブジェクト確率$d_i, _j$だけでなく$k$方向(デフォルト:$k=32$)に対する半径距離$r^k _{i, j}$を予測することにより,そのピクセルがどのラベルに属するかを出力します.最後に,非最大抑制(NMS)により最終的な出力を行います.
実装
GitHubページから引用し,コメントを追加しました.本コードではJupyter Notebookを使用しているため,他の開発環境を使用したい場合はコードの書き換えが必要となることに注意してください.
from stardist.data import test_image_nuclei_2d
from stardist.models import StarDist2D
from stardist.plot import render_label
from csbdeep.utils import normalize
import matplotlib.pyplot as plt
# 入力画像をテストデータのインスタンスとして読み込み
img = test_image_nuclei_2d()
# StarDistの学習済みモデルをインスタンスとして読み込み
model = StarDist2D.from_pretrained('2D_versatile_fluo')
# 入力画像を正規化した画像に対し,StarDist適用によりラベリングを実行
labels, _ = model.predict_instances(normalize(img))
# 入力画像の描画
plt.subplot(1,2,1)
plt.imshow(img, cmap="gray")
plt.axis("off")
plt.title("input image")
# ラベリング結果画像の描画
plt.subplot(1,2,2)
plt.imshow(render_label(labels, img=img))
plt.axis("off")
plt.title("prediction + input overlay")
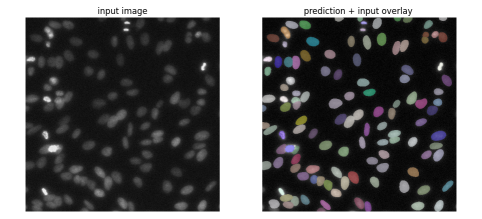
上の図より,高精度でインスタンスセグメンテーションができていることが確認できます.あとは,ブロブ解析によるノイズ判断を行ったり,関心領域(Region of Interest: ROI)の設定後に機械学習に回したりすることで細胞を解析,種類を識別することができます.
最後に
本記事では,StarDistについて大まかにまとめてみました.自身の研究に関連するためデータを記載できませんが,StarDistの精度は非常に高く,体感8~9割の細胞を捕捉できていました(私は医者でないので,あくまでも体感です).また,StarDistを適用する前の段階でノイズ軽減などのフィルタ処理を追加することで,セグメンテーションが向上しました.
また,本記事では2次元での話を中心に行いましたが,時間を追加した3次元バージョンもあるみたいです.今後は,そちらがベースとなり,細胞のリアルタイムな評価が可能になるのではと考えています.
ここまで読んでくださり,ありがとうございました.
参考
[1] https://github.com/stardist/stardist
[2] https://stardist.net
[3] https://www.youtube.com/watch?v=Amn_eHRGX5M&ab_channel=NEUBIAS
[4] https://www.youtube.com/watch?v=3yk9sBja7YI&ab_channel=DigitalSreeni