生存時間解析って?
生存時間解析とは何らかのイベントが発生するまでの時間を生存時間として統計的に分析する方法のことを言います。
医学で使用されることが多いです。
代表的な例としてがん患者の余命期間に関する分析があります。がん発症から死亡までの期間に対して分析を行います。
経済の分野だと経済学、経営学、マーケティングでも使用されます。
労働者が労働市場に参加している期間、企業が設立してから倒産するまでの期間、顧客が会員登録してから登録解除するまでの期間など、、、
その他さまざまな領域で応用されます。
すでに多くの方がこの手法について解説されていますのでここでは基本のおさらいとちょっとした発展の内容を記事にしようと思います。
カプランマイヤー推定法
生存時間解析では打ち切りデータを考慮して分析できるという特徴があります。
「時間」に関する分析を行っているので分析対象を観察できない場合があります。
(がん患者の例だと、例えば途中で病気が悪化して別の病院に移ったとか、がんとは別の原因で死亡してしまったとか、、、)
この場合は生存時間がはっきりしません。
生存時間がはっきりしないからといって欠損値にするのは分析に支障がでそうですね。
実際に分析上の問題があります。観察ができなくなるまでイベントは発生していないわけですから生存率を過小評価することになります。
生存時間関数はこの打ち切りデータに対しても分析対象をした分析手法です。
しかし、打ち切りデータが多いと精度が落ちる傾向にあるので、この点には注意が必要です。
カプランマイヤー推定法の式は次のようになります。
S(t)= \Pi_{t=1,2,3...}^T \left(1-\frac{d_{j}}{l_t} \right)
\\
j \in R(t_j)
$R(t_j)$は時間 $t_j$ におけるリスク集合です。
イベント発生のリスクのある、つまりこの時点までにイベントが発生していないデータの集合と言えば良いでしょうか。
カプランマイヤー法は生存時間と打ち切り時間に注目して生存率を推定したモデルとなっています。
しかし、これでは生存時間に影響を与える要因について全く考慮していません。
生存時間に影響を与える因子をモデルに組み込んだのが次のCox比例ハザードモデルです。
Cox比例ハザードモデル
モデル
Cox比例ハザードモデルでは生存関数のほかにハザード関数を定義します。
ここでは次節のためにハザード関数のモデルを仮定しないセミパラメトリックモデルを書きます。
$H(t)$は累積ハザード関数と言われハザード関数を時間tを増やすごとに加算した値です。
$h_0(t)$は基準ハザードと言われます。$X=0$ のときのハザード関数と一致します。
またハザード関数はイベントの発生のしやすさを表します。大きいほど生存関数は小さくなるのがわかりますね。
生存関数
S(t) = exp(-H(t))
ハザード関数
h(t|X) = h_0(t)exp(X'\beta)
$X$はデータで$\beta$は係数ベクトルです。
係数ベクトルの解釈は次のようになります。
正 → イベントが発生しやすくなる
負 → イベントが発生しににくなる
満たすべき条件
主に二つの条件を満たす必要があります。
-
対数線形性
対数を取ったときに説明変数と目的変数が線形関係になっているlog(h(t|X)) = log(h_0(t)) + X'\beta
-
比例ハザード性
イベント発生のしやすさが時間を通して一定である\frac{h(t|X_1)}{h(t|X_2)} = \frac{h_0(t)exp(X_1'\beta)}{h_0(t)exp(X_2'\beta)} = \frac{exp(X_1'\beta)}{exp(X_2'\beta)}
この二つの条件は非常に強い制約です。 とくに比例ハザード性については現実的に満たすことはそう多くありません。
推定方法
部分尤度関数と呼ばれる関数を最大化することでパラメータ $\beta$ を推定します。
L(\beta) =\prod_{i=1}^{n} \frac{exp(X_{i}'\beta)}{\sum_{j\in R(t_{i})}exp(X_{j}\beta)}
しかし、これは生存時間が同じレコードを含まない場合の式です。
生存時間が同じレコードが含まれる場合には少し面倒な計算をする必要があり、計算量が膨大になります。
ですのでこれを緩和するための近似式があります。
Breslow近似が一番使用されると思いますのでこれを紹介します。
L(\beta) =\prod_{i=1}^{n} \frac{exp(s_{i}'\beta)}{[\sum_{j\in R_(t_{i})}exp(X_{j}\beta)]^{d_j}}
この部分尤度関数を最大化するための手法としてはニュートン法が用いられます。
ここでは説明を省きます。
Python で実装
実装には Google Colaboratory を使用します。
lifelines というライブラリを使用することで簡単にCox比例ハザードモデルを実行することができます。
部分尤度関数とニュートン法の実装は面倒なのでありがたいですね。
lifelinesをインストール、その他必要なライブラリをインポートします。
!pip install lifelines
import pandas as pd
import numpy as np
import matplotlib.pyplot as plt
import lifelines
from lifelines.datasets import load_regression_dataset
from lifelines import CoxPHFitter
import sklearn
from sklearn.model_selection import train_test_split
今回はMayo Clinic Primary Biliary Cholangitis Data(PBC)というデータを使用します。データの詳しい説明はここから参照してください。
Pythonでダウンロードする方法がわからなかったので一度Rでダウンロードしたデータを保存して使用しました。
※lifelinesに練習用のデータがあります。興味のある方はそちらで試してみてください!私はなんとなく他のデータを使った例を見たかったので面倒ですがわざわざこのデータを使いました。
データには id、共変量、生存時間、イベント発生の有無の情報が記載されています。
- id
- データのid
- time
- 生存時間
- status
- イベント発生の有無
- その他
- 共変量
PBCのstatusには
2:イベント発生 1:移植 0:打ち切り
と三種類ありますが、今回はわかりやすいように移植は打ち切りとしてイベント発生と打ち切りの二つに分けます。
from google.colab import drive
drive.mount('/content/drive')
PATH = "自身のドライブのパス"
pbc = pd.read_csv(PATH + "PBC.csv", usecols=['id', 'time', 'status', 'trt', 'age', 'sex', 'ascites',
'hepato', 'spiders', 'edema', 'bili', 'chol', 'albumin', 'copper',
'alk.phos', 'ast', 'trig', 'platelet', 'protime', 'stage'])
pbc["d_sex"] = np.where(pbc["sex"]=="m", 1, 0)
pbc["status"] = np.where(pbc["status"]==2), 1, 0)
pbc = pbc.sort_values("time").fillna(method="ffill") # 今回欠損値は適当に埋めます
pbc.dropna(axis=0, how="any", inplace=True)
Cox比例ハザードモデルに fit させます。
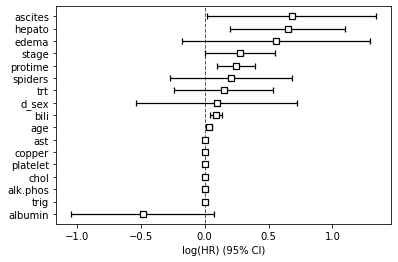
対数部分尤度や各係数ベクトルとその95%信頼区間、p値などが出力されます。
また C index(Concordance) や AIC なども見ることができるのでモデルの当てはまりを確認することもできます。
train, test = train_test_split(pbc, test_size=0.2, random_state=seed)
cph = CoxPHFitter()
cph.fit(train, duration_col="time", event_col="status", show_progress=True, fit_options={"step_size":0.5})
cph.print_summary()
cph.plot()
次に各共変量の影響を考慮した生存率関数を出力します。
統計的に有意な変数についてその影響を見てみます。
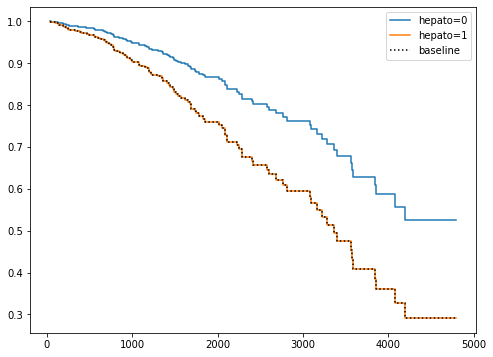
ダミー変数の"hepato"と連続変数の"bili"の生存関数をそれぞれプロットします。
"hepato"と"bili"の係数はともに正なので値が大きい(ダミー変数の場合は1のとき)ほどイベントは発生しやすくなり、生存率は低くなります。
dummy = [0, 1]
cph.plot_partial_effects_on_outcome(
figsize=(8,6),
covariates="hepato",
values=dummy)
values = [train["bili"].max(), train["bili"].mean(), train["bili"].min()]
cph.plot_partial_effects_on_outcome(
figsize=(8,6),
covariates="bili",
values=values)
また以下のコードでテストデータに対する生存率とハザードの予測を計算することができます。
cph.predict_survival_function(test)
cph.predict_partial_hazard(test)
生存時間解析×機械学習
派生モデル
Cox比例ハザードモデルには拡張モデルが多くあります。
この方の記事が非常に参考になりました。
この記事ではニューラルネットワークの手法を組み合わせたモデルだけではなくランダムフォレストやサポートベクターマシンを用いたモデルなどについても紹介しています。
またニューラルネットワークを用いたモデルにはDeepSurv, Cox-nnet, pycoxなどさまざまなモデルがあり、それぞれ微妙に違うようです(それぞれの細かな違いまでは理解できず、、、)
ニューラルネットワークを用いたモデル
Cox比例ハザードモデルの満たすべき条件のうち、
一つ目の対数線形性を仮定しないモデルになります。
二つ目の比例ハザード性は仮定しています。
ハザード関数は次のようになります。
h(t|X)=exp(h_0(t)f(X))
$X'\beta$だった部分を多層にすることで非線形のモデルにしようという発想ですね。
部分尤度関数もハザード関数にしたがってそのまま $f(X)$ に置き換わります。
L(\beta) =\prod_{i=1}^{n} \frac{exp(f(X))}{\sum_{j\in R(t_{i})}exp(f(X))}
しかし、この場合、ニュートン法を使って部分尤度関数を最大化することはできません!
ではどうすれば良いのでしょうか...?
多くの機械学習では「損失関数を最小化する」ことで最適なパラメータを推定するのでこれを応用します。
この問題では損失関数を部分尤度関数で置き換えればよいですね。
部分尤度関数にマイナスをつけた値を損失関数として、最小になるパラメータを推定します。
Cox-nnetという名前でモデルが公開されているのでこれを利用します。
、、、と思っていましたが、Python3系にバージョンアップされるとエラーが起きてしまい実行できません!
どうやらPython2系の環境であれば実行できるみたいです。
google colab を使用する場合にはPython2系の環境はサービスが終了しているので使用できません。
なので、Python3系の環境でも実行できるDeepSurvを使用します。
まずは必要なパッケージをインストールします。
!pip install --upgrade https://github.com/Lasagne/Lasagne/archive/master.zip
!pip install h5py
!git clone https://github.com/jaredleekatzman/DeepSurv.git
!cd DeepSurv
!pip install DeepSurv
!pip install tensorboard_logger
from DeepSurv import deepsurv
from DeepSurv.deepsurv import *
次にデータを学習できる形に整形していきます。
"x", "t", ",e"をそれぞれ共変量、時間、イベント発生の有無として辞書を作成します。
x = pbc.drop(["time", "status"], axis=1)
y = pbc["time"]
status = pbc["status"]
x_train, x_test, x_time, y_time, y_train_status, y_test_status = train_test_split(x, y, status, test_size=0.2, random_state=seed)
traindata = {
"x": x_train.to_numpy().astype(np.float32),
"t": x_time.to_numpy().astype(np.float32),
"e": y_train_status.to_numpy().astype(np.int32)
}
testdata = {
"x": x_test.to_numpy().astype(np.float32),
"t": y_time.to_numpy().astype(np.float32),
"e": y_test_status.to_numpy().astype(np.int32)
}
ハイパーパラメータを設定して、学習させます。
上から順に
- 学習率
- 中間層の数をユニット数
- weight decay
- momentumのハイパーパラメータ
- L2,L1正則化
- 活性化関数
- ドロップアウト
- 共変量の数
- バッチ正規化をするかどうか
- 最初に学習データを標準化するかどうか
となります。
学習中はloss(対数尤度のマイナス値)と C index(ci)が出力されます。
seed = 1111
np.random.seed(seed)
hyperparameter = {
"learning_rate": 0.005,
"hidden_layers_sizes" : [16, 16, 16],
"lr_decay" : 1e-5,
"momentum": 0.9,
"L2_reg": 1e-4,
"L1_reg": 1e-4,
"activation": "rectify",
"dropout": 0.1,
"n_in": traindata["x"].shape[1],
"batch_norm": True,
"standardize": True
}
# 学習の様子を記録
experiment_name = 'test_experiment_sebastian'
logdir = './logs/tensorboard/'
logger = deepsurv_logger.TensorboardLogger(experiment_name, logdir=logdir)
# エポック数を指定
epochs = 1000
network = deepsurv.DeepSurv(**hyperparameter)
log = network.train(traindata, testdata, n_epochs=epochs, logger=logger)
# 2023-02-25 04:36:05,702 - Training step 0/1000 | | - loss: 5.8520 - ci: 0.2988
# INFO:test_experiment_sebastian:Training step 0/1000 | | - loss: 5.8520 - ci: 0.2988
# 2023-02-25 04:36:48,580 - Training step 250/1000 |****** | - loss: 3.7974 - ci: 0.9386
# INFO:test_experiment_sebastian:Training step 250/1000 |****** | - loss: 3.7974 - ci: 0.9386
# 2023-02-25 04:37:31,986 - Training step 500/1000 |************ | - loss: 3.7351 - ci: 0.9607
# INFO:test_experiment_sebastian:Training step 500/1000 |************ | - loss: 3.7351 - ci: 0.9607
# 2023-02-25 04:38:14,118 - Training step 750/1000 |****************** | - loss: 3.4983 - ci: 0.9731
# INFO:test_experiment_sebastian:Training step 750/1000 |****************** | - loss: 3.4983 - ci: 0.9731
# 2023-02-25 04:38:52,067 - Finished Training with 1000 iterations in 168.56s
# INFO:test_experiment_sebastian:Finished Training with 1000 iterations in 168.56s
最後にモデルの当てはまりを確認します。
モデルを比較するための指標はC indexやAIC、BICを用いることが多いです。
C indexは観測できるデータのうち予測が一致する比率のことを言い、1に近いほどよいモデルであることを示します。
ここで、予測が一致するというのは
「任意のデータのペアについて生存の予測確率がより高いデータがより長く生存する」
ということを指します。
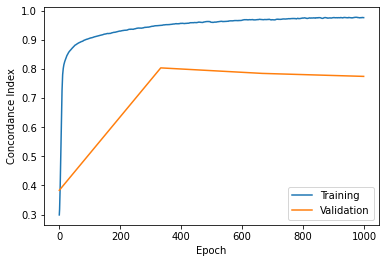
deepsurv.viz.plot_log(log)
学習データに対するC indexはすさまじいです。
ただvalidation(testdata)のC indexはあまりよろしくないですね。
学習データだけを見るのであればDeepSurvのほうがよいと考えられます。
また対数部分尤度を見ると収束していないように見えます。
今回の場合、原因はサンプルサイズが小さいことだと考えられます。
ニューラルネットワークの学習にはそれなりに大きなサンプルサイズが必要ですからね。サンプルサイズがもっと大きければうまく収束すると思います。
おわりに
Cox比例ハザードモデルとそこからニューラルネットワークを組み合わせたモデルについて説明しました。ニューラルネットワークを用いた生存時間解析の記事が少なかったように感じたので、今回記事を作成しました。内容については数学的な部分を省いているので物足りないと感じる人もいるかも知れません。
DeepSurvなどの機械学習モデルはあくまで予測精度を求める場合に役に立ちます。
各変数の影響について検定はできません。
しかし、モデルの解釈についてはshap値を使った方法が考案されています。
気になる方は調べてみてください。
参考
Anny Xiang, Pablo Lapuerta, Alex Ryutov, Jonathan Buckley, Stanley Azen(2000)
Comparison of the performance of neural network methods and Cox regression for censored survival data
H˚avard Kvamme, Ørnulf Borgan, Ida Scheel(2019)
Time-to-Event Prediction with Neural Networks
and Cox Regression
PBC
lifelines
DeepSurvの論文
DeepSurv
cox-nnetの論文
cox-nnet
今回コードを参考させていただきました